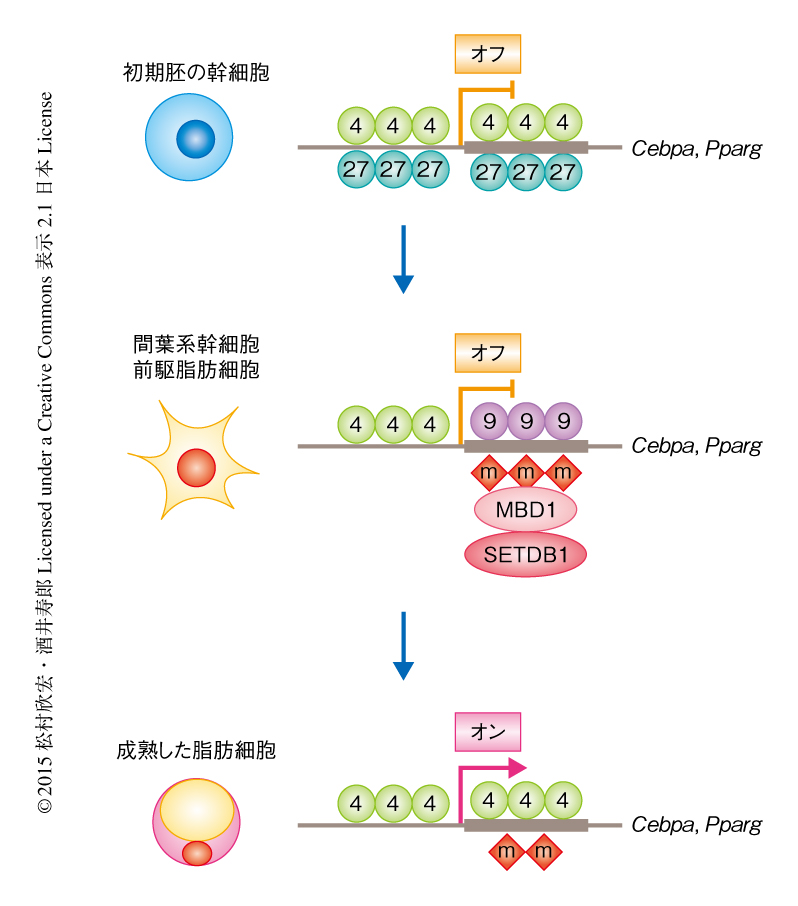
細胞系譜に特異的なDNAメチル化により形成されるヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインは脂肪細胞の分化を休止させる
松村欣宏・酒井寿郎
(東京大学先端科学技術研究センター 代謝医学分野)
email:松村欣宏,酒井寿郎
DOI: 10.7875/first.author.2015.138
H3K4/H3K9me3 bivalent chromatin domains targeted by lineage-specific DNA methylation pauses adipocyte differentiation.
Yoshihiro Matsumura, Ryo Nakaki, Takeshi Inagaki, Ayano Yoshida, Yuka Kano, Hiroshi Kimura, Toshiya Tanaka, Shuichi Tsutsumi, Mitsuyoshi Nakao, Takefumi Doi, Kiyoko Fukami, Timothy F. Osborne, Tatsuhiko Kodama, Hiroyuki Aburatani, Juro Sakai
Molecular Cell, 60, 584-596 (2015)
ヒストンH3のLys4のトリメチル化修飾は転写の活性化,ヒストンH3のLys27のトリメチル化修飾およびヒストンH3のLys9のトリメチル化修飾は転写の抑制に機能する.初期胚の幹細胞において,ヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインは,発生にかかわる遺伝子の発現を低く維持している.筆者らは,間葉系幹細胞および前駆脂肪細胞において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインを見い出した.このクロマチンドメインは脂肪細胞の分化におけるマスター遺伝子であるPparg遺伝子およびCebpa遺伝子の発現を分化が必要なときまで低く維持していた.細胞系譜に特異的なDNAメチル化によりヒストンH3のLys9のメチル化酵素であるSETDB1がリクルートされ,このSETDB1はヒストンH3のLys4がトリメチル化修飾された転写開始点のすぐ下流のヒストンH3のLys9をメチル化し,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインを形成した.Cebpa遺伝子において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインは転写因子C/EBPβの結合,ヒストンのアセチル化,さらに,ヒストンH3のLys4のトリメチル化修飾が広がることをさまたげていた.これにより,RNAポリメラーゼIIを転写開始点に停滞させ,Cebpa遺伝子の発現および脂肪細胞の分化を抑制していた.
発生にともない,多分化能をもつ初期胚の幹細胞は多分化性幹細胞や前駆細胞へと運命づけられ,最終的にそれぞれの機能をもつ成熟した細胞へと分化する.自己の再生や傷害からの修復のため,成熟した組織は運命決定された前駆細胞を維持する必要がある.これまで,ES細胞が多分化能を維持するエピゲノム機構については広く研究されてきたが1),運命決定された前駆細胞が未分化な状態を維持する機構については明らかではなかった.
DNAのメチル化やヒストンの修飾は細胞の分化にともない変化し,遺伝子の発現を制御する.ヒストンの修飾はヌクレオソームの構造を変化させクロマチンの動態を制御する.ヒストンのメチル化はそのアミノ酸残基の位置により転写の活性化あるいは転写の抑制に機能する.ヒストンH3のLys4のトリメチル化修飾は転写の活性化に,ヒストンH3のLys9のトリメチル化修飾およびヒストンH3のLys27のトリメチル化修飾は転写の抑制に機能することが知られている2).
ES細胞において発生にかかわる約2500の遺伝子においては,転写を活性化するヒストンH3のLys4のトリメチル化修飾,および,転写を抑制するヒストンH3のLys27のトリメチル化修飾をもつクロマチンドメインが形成される3).このクロマチンドメインは発生にかかわる遺伝子の発現を低く保つことによりES細胞の多分化能を維持する一方,必要なときに遺伝子の発現を活性化する役割をもつ.細胞の分化にともない,このクロマチンドメインの多くはヒストンH3のLys4あるいはLys27にのみトリメチル化修飾をもつクロマチンドメインへと変化する4).活性化される遺伝子においては転写を抑制するヒストンH3のLys27のトリメチル化修飾が消失し,転写を活性化するヒストンH3のLys4のトリメチル化修飾が転写開始点の下流に広がる.これにともないRNAポリメラーゼIIが伸長する.しかし,細胞の分化にともないヒストンH3のLys27のトリメチル化修飾を消失した遺伝子のすべてが活性化されるわけではない.
ヒストンH3のLys9のトリメチル化修飾も転写を抑制するヒストン修飾であり,ヘテロクロマチンの形成と関係する5).培養条件にあるES細胞とは異なり,生体における初期胚の幹細胞は一時的に多分化能をもつ.マウスの原外胚葉においてはヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインが存在するが,一部の細胞運命が決定した栄養芽層や胚外外胚葉においてはヒストンH3のLys27のトリメチル化修飾の大部分が消失し,代わりに,ヒストンH3のLys9のトリメチル化修飾が存在することが報告されている6).このことから,細胞運命が決定する過程において,ヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインはヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインへと変化する可能性が示唆されるが,どのような機構によりエピゲノムが変化するかは不明である.
脂肪細胞の分化は前駆脂肪細胞への運命決定,終末分化などいくつかの段階をへて進行する.その過程において,脂肪細胞の分化のマスター転写因子であるPPARγおよびC/EBPαが一連の転写プログラムを制御する7).ES細胞においてはPPARγをコードするPparg遺伝子にヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインが形成されているが,前駆脂肪細胞株である3T3-L1細胞においてはPparg遺伝子に転写を抑制するヒストンH3のLys27のトリメチル化修飾が存在しないにもかかわらず遺伝子の発現は低く維持されている8).分化を誘導する刺激により初期の転写因子C/EBPβの発現が誘導され,Pparg遺伝子およびC/EBPαをコードするCebpa遺伝子の発現が活性化される.C/EBPβの発現は分化を誘導する刺激ののち2~4時間で誘導されるが,14~24時間までPparg遺伝子およびCebpa遺伝子には結合できない9).これはC/EBPβがDNAに結合するにはリン酸化が必要なためであるが,クロマチンの構造のレベルにおいてDNAへの結合が制御されている可能性も考えられる.
今回,筆者らは,間葉系幹細胞および前駆脂肪細胞においてエピゲノムを解析し,脂肪細胞の分化の鍵となる遺伝子においてヒストンH3のLys4およびLys9にトリメチル化修飾をもつ新規のクロマチンドメインを見い出した.
脂肪細胞へと運命決定された細胞において未分化性を維持するエピゲノム機構について明らかにするため,TT2 ES細胞株,10T1/2間葉系幹細胞株,3T3-L1前駆脂肪細胞株において,転写を活性化するヒストンH3のLys4のトリメチル化修飾,および,転写を抑制するヒストンH3のLys27のトリメチル化修飾について,クロマチン免疫沈降-シークエンシング法により解析した.以前に報告されたとおり,ES細胞においてはPparg遺伝子およびCebpa遺伝子にヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインが存在した.しかし,運命決定された間葉系幹細胞と前駆脂肪細胞においては,これらの遺伝子にヒストンH3のLys4のトリメチル化修飾は存在したがヒストンH3のLys27のトリメチル化修飾は存在しなかった.転写を抑制するヒストンH3のLys27のトリメチル化修飾が存在しないにもかかわらずPparg遺伝子およびCebpa遺伝子の発現は低く維持されていたことから,これらの細胞においては別のエピゲノム機構がこれらマスター遺伝子の発現を抑制している可能性が示唆された.
以前に筆者らは,ヒストンH3のLys9のメチル化酵素であるSETDB1が前駆脂肪細胞において脂肪細胞の分化を抑制することを報告した10).間葉系幹細胞および前駆脂肪細胞においてSETDB1をノックダウンすると脂肪滴の蓄積が促進された.SETDB1の標的となる遺伝子について明らかにするため,SETDB1およびヒストンH3のLys9のトリメチル化修飾についてクロマチン免疫沈降-シークエンシング法により解析した.その結果,前駆脂肪細胞において7005のSETDB1結合領域が見い出された.その約半分は遺伝子の転写領域に存在し,多くは転写開始点の下流1 kb以内に存在した.転写を活性化するヒストンH3のLys4のトリメチル化修飾と転写を抑制するヒストンH3のLys9のトリメチル化修飾は同じヌクレオソームには存在しないと考えられている4).しかし,前駆脂肪細胞においてヒストンH3のLys4のトリメチル化修飾をもつ約1万の遺伝子のうち,約400の遺伝子はヒストンH3のLys9のトリメチル化修飾ももっていた.ヒストンH3のLys4のトリメチル化修飾およびヒストンH3のLys9のトリメチル化修飾をもつ遺伝子のうち,約200の遺伝子においてSETDB1との結合も認められた.このなかには発生に関与する遺伝子が濃縮されており,脂肪細胞の分化のマスター遺伝子であるPparg遺伝子およびCebpa遺伝子も含まれていた.Pparg遺伝子およびCebpa遺伝子は特有のヒストン修飾のパターンをもち,転写開始点の上流においてヒストンH3のLys4のトリメチル化修飾,転写開始点の下流においてヒストンH3のLys9のトリメチル化修飾をもち,ヒストンH3のLys9のトリメチル化修飾部位にはSETDB1が局在していた.間葉系幹細胞においても,ヒストンH3のLys4のトリメチル化修飾およびヒストンH3のLys9のトリメチル化修飾をもつ遺伝子のうち,Cebpa遺伝子を含む約200の遺伝子においてSETDB1との結合が認められた.これらの結果から,運命決定された間葉系幹細胞および前駆脂肪細胞において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインが存在することが示された.
マイクロアレイ解析の結果,SETDB1のノックダウンにより129の遺伝子の発現が誘導され,そのうち,Pparg遺伝子およびCebpa遺伝子を含む10の遺伝子にはSETDB1が結合することが明らかにされた.SETDB1のノックダウンによりSETDB1の結合する遺伝子の一部のみで発現が誘導されたことから,転写を抑制するヒストンH3のLys9のトリメチル化修飾の消失だけではなく,転写因子やクロマチンタンパク質のはたらきも遺伝子発現の誘導には必要であると考えられた.成熟した脂肪細胞においてはSETDB1の発現が低下し,Pparg遺伝子およびCebpa遺伝子の転写開始点の下流にはヒストンH3のLys9のトリメチル化修飾は存在しなかった.また,前駆脂肪細胞においてSETDB1をノックダウンすると,Pparg遺伝子およびCebpa遺伝子の転写開始点の下流のヒストンH3のLys9のトリメチル化修飾は消失した.これらの結果から,SETDB1はヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインの形成にかかわることが示唆された.
ES細胞においてヒストンH3のLys27のトリメチル化修飾はRNAポリメラーゼIIの停滞に関与することが知られている.前駆脂肪細胞においては,ヒストンH3のLys27のトリメチル化修飾の代わりに,ヒストンH3のLys9のトリメチル化修飾がPparg遺伝子およびCebpa遺伝子においてRNAポリメラーゼIIの停滞に関与している可能性が考えられた.前駆脂肪細胞のCebpa遺伝子においては転写を活性化するヒストンH3のLys4のトリメチル化修飾は転写開始点の上流にのみにみられ,遺伝子の転写領域には認められなかった.一方,Pparg遺伝子およびCebpa遺伝子の発現している成熟した脂肪細胞においては,ヒストンH3のLys4のトリメチル化修飾は転写開始点の上流だけでなく遺伝子の転写領域にも認められた.SETDB1のノックダウンにより遺伝子の転写領域におけるヒストンH3のLys9のトリメチル化修飾が消失すると,Pparg遺伝子およびCebpa遺伝子において遺伝子の転写領域にもヒストンH3のLys4のトリメチル化修飾が認められるようになった.これにともない,Pparg遺伝子およびCebpa遺伝子において遺伝子の転写領域にRNAポリメラーゼIIが検出された.
C/EBPβは脂肪細胞分化を誘導する初期の転写因子であり,Cebpa遺伝子の発現を活性化することが知られている.通常,C/EBPβは分化を誘導する刺激ののち14時間まではほとんど標的となる遺伝子には結合しないことから,Cebpa遺伝子におけるヒストンH3のLys9のトリメチル化修飾がC/EBPβのリクルートをさまたげている可能性について検証した.SETDB1のノックダウンにより,通常はほとんどCebpa遺伝子に結合しないC/EBPβの結合が増加した.さらに,SETDB1のノックダウンにより,クロマチンの開いた構造の特徴であるヒストンH3のLys27のアセチル化修飾が増加した.これらの結果から,前駆脂肪細胞におけるSETDB1によるCebpa遺伝子のヒストンH3のLys9のトリメチル化修飾は,C/EBPβの結合,ヒストンのアセチル化,ヒストンH3のLys4のトリメチル化修飾の遺伝子の転写領域への広がりをさまたげ,RNAポリメラーゼIIを転写開始点に停滞させることが示された.
SETDB1結合領域のモチーフを解析したところCGを含む塩基配列の濃縮が認められ,SETDB1のリクルートとCpGメチル化にはなんらかの関係があると考えられた.前駆脂肪細胞においてCpGメチル化について解析したところ,転写開始点の前後2 kbに存在するSETDB1結合領域の約60%がCpGメチル化されていた.ES細胞では,Cebpa遺伝子において遺伝子の転写領域はCpGメチル化されておらず,SETDB1はリクルートされていなかった.一方,間葉系幹細胞および前駆脂肪細胞において,Cebpa遺伝子の遺伝子の転写領域はCpGメチル化されており,SETDB1がリクルートされていた.これらの結果から,SETDB1のリクルートと細胞系譜に特異的なDNAメチル化は同時に起こることが示唆された.
SETDB1はCpGメチル化部位の結合タンパク質であるMBD1と相互作用することが知られている11).前駆脂肪細胞において,MBD1の結合についてクロマチン免疫沈降-シークエンシング法により解析したところ,SETDB1結合領域の約80%はMBD1結合領域と一致した.Cebpa遺伝子において,CpGメチル化,MBD1,SETDB1は共局在していた.MBD1のノックダウンにより,Cebpa遺伝子の発現は2~3倍にも増強され,脂肪滴の蓄積が促進された.また,MBD1のノックダウンにより,Cebpa遺伝子へのSETDB1のリクルートおよびヒストンH3のLys9のトリメチル化修飾は減少した.SETDB1のリクルートおよびヒストンH3のLys9のトリメチル化修飾の減少は野生型のMBD1の発現により回復したが,CpGメチル化部位との結合ドメインに変異をもつMBD1の発現では回復しなかった.これらの結果から,MBD1はCebpa遺伝子のCpGメチル化部位へSETDB1をリクルートし,ヒストンH3のLys9のメチル化修飾および遺伝子の発現を抑制することが示された.
この研究において,筆者らは,運命決定された前駆脂肪細胞の約200の遺伝子において,転写開始点の上流にヒストンH3のLys4のトリメチル化修飾,転写開始点の下流にヒストンH3のLys9のトリメチル化修飾をもつクロマチンドメインを見い出した.細胞系譜に特異的な遺伝子の転写領域のDNAメチル化およびヒストンH3のLys9のメチル化酵素であるSETDB1のリクルートにより,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインが形成された.このクロマチンドメインはRNAポリメラーゼIIを転写開始点の上流に停滞させ遺伝子の発現を低く維持する一方で,分化を誘導する刺激によりクロマチンを開いた構造へと変化させ,転写因子やクロマチンタンパク質と共役して転写をすみやかに誘導する役割をもっていた(図1).
ヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインはES細胞において発生にかかわる約2500の遺伝子の発現を低く保ち多分化能の維持にかかわる.このクロマチンドメインにおいて,転写を活性化するヒストンH3のLys4のトリメチル化修飾と転写を抑制するヒストンH3のLys27のトリメチル化修飾は転写開始点の近傍の同じあるいは隣接するヌクレオソームに存在する.一方,この研究において見い出されたヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインは,間葉系幹細胞あるいは前駆脂肪細胞においてわずか約200の遺伝子にて形成されていた.このクロマチンドメインにおいては,転写開始点の上流にヒストンH3のLys4のトリメチル化修飾,転写開始点の下流にヒストンH3のLys9のトリメチル化修飾が直列して存在し,ひとつの遺伝子において開いた構造のクロマチンと閉じた構造のクロマチンが共存していると考えられた.このクロマチンドメインは転写開始点の下流における細胞系譜に特異的なDNAメチル化とヒストンH3のLys9のメチル化酵素であるSETDB1のリクルートとが組み合わさることにより形成されたことから,発生にかかわる特定の遺伝子を特定の時期だけ制御するためのしくみと考えられた.
脂肪細胞への運命決定の過程において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインが前駆細胞にふみとどまらせる役割をもつことが見い出されたが,同様のクロマチンドメインが神経細胞,筋細胞,骨細胞といったさまざまな細胞の運命決定の過程において形成され終末分化が必要なときまで前駆細胞を維持している可能性が考えられる.今後,細胞系譜に特異的なDNAメチル化のパターンがどのように形成され,クロマチンの局所的な構造が遺伝子の発現をどのように動的に制御するかについて理解がもとめられる.
略歴:2004年 東北大学大学院理学研究科 修了,同年 秋田大学医学部 研究員,2008年 米国Oregon Health & Science大学 研究員を経て,2011年より東京大学先端科学技術研究センター 特任助教(現 助教).
研究テーマ:肥満および生活習慣病におけるエピゲノムの役割.
抱負:DNAとタンパク質との複合体の解析から複雑な生命現象を解明したい.
酒井 寿郎(Juro Sakai)
東京大学先端科学技術研究センター 教授.
研究室URL:http://www.mm.rcast.u-tokyo.ac.jp
© 2015 松村欣宏・酒井寿郎 Licensed under CC 表示 2.1 日本
(東京大学先端科学技術研究センター 代謝医学分野)
email:松村欣宏,酒井寿郎
DOI: 10.7875/first.author.2015.138
H3K4/H3K9me3 bivalent chromatin domains targeted by lineage-specific DNA methylation pauses adipocyte differentiation.
Yoshihiro Matsumura, Ryo Nakaki, Takeshi Inagaki, Ayano Yoshida, Yuka Kano, Hiroshi Kimura, Toshiya Tanaka, Shuichi Tsutsumi, Mitsuyoshi Nakao, Takefumi Doi, Kiyoko Fukami, Timothy F. Osborne, Tatsuhiko Kodama, Hiroyuki Aburatani, Juro Sakai
Molecular Cell, 60, 584-596 (2015)
要 約
ヒストンH3のLys4のトリメチル化修飾は転写の活性化,ヒストンH3のLys27のトリメチル化修飾およびヒストンH3のLys9のトリメチル化修飾は転写の抑制に機能する.初期胚の幹細胞において,ヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインは,発生にかかわる遺伝子の発現を低く維持している.筆者らは,間葉系幹細胞および前駆脂肪細胞において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインを見い出した.このクロマチンドメインは脂肪細胞の分化におけるマスター遺伝子であるPparg遺伝子およびCebpa遺伝子の発現を分化が必要なときまで低く維持していた.細胞系譜に特異的なDNAメチル化によりヒストンH3のLys9のメチル化酵素であるSETDB1がリクルートされ,このSETDB1はヒストンH3のLys4がトリメチル化修飾された転写開始点のすぐ下流のヒストンH3のLys9をメチル化し,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインを形成した.Cebpa遺伝子において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインは転写因子C/EBPβの結合,ヒストンのアセチル化,さらに,ヒストンH3のLys4のトリメチル化修飾が広がることをさまたげていた.これにより,RNAポリメラーゼIIを転写開始点に停滞させ,Cebpa遺伝子の発現および脂肪細胞の分化を抑制していた.
はじめに
発生にともない,多分化能をもつ初期胚の幹細胞は多分化性幹細胞や前駆細胞へと運命づけられ,最終的にそれぞれの機能をもつ成熟した細胞へと分化する.自己の再生や傷害からの修復のため,成熟した組織は運命決定された前駆細胞を維持する必要がある.これまで,ES細胞が多分化能を維持するエピゲノム機構については広く研究されてきたが1),運命決定された前駆細胞が未分化な状態を維持する機構については明らかではなかった.
DNAのメチル化やヒストンの修飾は細胞の分化にともない変化し,遺伝子の発現を制御する.ヒストンの修飾はヌクレオソームの構造を変化させクロマチンの動態を制御する.ヒストンのメチル化はそのアミノ酸残基の位置により転写の活性化あるいは転写の抑制に機能する.ヒストンH3のLys4のトリメチル化修飾は転写の活性化に,ヒストンH3のLys9のトリメチル化修飾およびヒストンH3のLys27のトリメチル化修飾は転写の抑制に機能することが知られている2).
ES細胞において発生にかかわる約2500の遺伝子においては,転写を活性化するヒストンH3のLys4のトリメチル化修飾,および,転写を抑制するヒストンH3のLys27のトリメチル化修飾をもつクロマチンドメインが形成される3).このクロマチンドメインは発生にかかわる遺伝子の発現を低く保つことによりES細胞の多分化能を維持する一方,必要なときに遺伝子の発現を活性化する役割をもつ.細胞の分化にともない,このクロマチンドメインの多くはヒストンH3のLys4あるいはLys27にのみトリメチル化修飾をもつクロマチンドメインへと変化する4).活性化される遺伝子においては転写を抑制するヒストンH3のLys27のトリメチル化修飾が消失し,転写を活性化するヒストンH3のLys4のトリメチル化修飾が転写開始点の下流に広がる.これにともないRNAポリメラーゼIIが伸長する.しかし,細胞の分化にともないヒストンH3のLys27のトリメチル化修飾を消失した遺伝子のすべてが活性化されるわけではない.
ヒストンH3のLys9のトリメチル化修飾も転写を抑制するヒストン修飾であり,ヘテロクロマチンの形成と関係する5).培養条件にあるES細胞とは異なり,生体における初期胚の幹細胞は一時的に多分化能をもつ.マウスの原外胚葉においてはヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインが存在するが,一部の細胞運命が決定した栄養芽層や胚外外胚葉においてはヒストンH3のLys27のトリメチル化修飾の大部分が消失し,代わりに,ヒストンH3のLys9のトリメチル化修飾が存在することが報告されている6).このことから,細胞運命が決定する過程において,ヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインはヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインへと変化する可能性が示唆されるが,どのような機構によりエピゲノムが変化するかは不明である.
脂肪細胞の分化は前駆脂肪細胞への運命決定,終末分化などいくつかの段階をへて進行する.その過程において,脂肪細胞の分化のマスター転写因子であるPPARγおよびC/EBPαが一連の転写プログラムを制御する7).ES細胞においてはPPARγをコードするPparg遺伝子にヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインが形成されているが,前駆脂肪細胞株である3T3-L1細胞においてはPparg遺伝子に転写を抑制するヒストンH3のLys27のトリメチル化修飾が存在しないにもかかわらず遺伝子の発現は低く維持されている8).分化を誘導する刺激により初期の転写因子C/EBPβの発現が誘導され,Pparg遺伝子およびC/EBPαをコードするCebpa遺伝子の発現が活性化される.C/EBPβの発現は分化を誘導する刺激ののち2~4時間で誘導されるが,14~24時間までPparg遺伝子およびCebpa遺伝子には結合できない9).これはC/EBPβがDNAに結合するにはリン酸化が必要なためであるが,クロマチンの構造のレベルにおいてDNAへの結合が制御されている可能性も考えられる.
今回,筆者らは,間葉系幹細胞および前駆脂肪細胞においてエピゲノムを解析し,脂肪細胞の分化の鍵となる遺伝子においてヒストンH3のLys4およびLys9にトリメチル化修飾をもつ新規のクロマチンドメインを見い出した.
1.運命決定された細胞におけるヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインの存在
脂肪細胞へと運命決定された細胞において未分化性を維持するエピゲノム機構について明らかにするため,TT2 ES細胞株,10T1/2間葉系幹細胞株,3T3-L1前駆脂肪細胞株において,転写を活性化するヒストンH3のLys4のトリメチル化修飾,および,転写を抑制するヒストンH3のLys27のトリメチル化修飾について,クロマチン免疫沈降-シークエンシング法により解析した.以前に報告されたとおり,ES細胞においてはPparg遺伝子およびCebpa遺伝子にヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインが存在した.しかし,運命決定された間葉系幹細胞と前駆脂肪細胞においては,これらの遺伝子にヒストンH3のLys4のトリメチル化修飾は存在したがヒストンH3のLys27のトリメチル化修飾は存在しなかった.転写を抑制するヒストンH3のLys27のトリメチル化修飾が存在しないにもかかわらずPparg遺伝子およびCebpa遺伝子の発現は低く維持されていたことから,これらの細胞においては別のエピゲノム機構がこれらマスター遺伝子の発現を抑制している可能性が示唆された.
以前に筆者らは,ヒストンH3のLys9のメチル化酵素であるSETDB1が前駆脂肪細胞において脂肪細胞の分化を抑制することを報告した10).間葉系幹細胞および前駆脂肪細胞においてSETDB1をノックダウンすると脂肪滴の蓄積が促進された.SETDB1の標的となる遺伝子について明らかにするため,SETDB1およびヒストンH3のLys9のトリメチル化修飾についてクロマチン免疫沈降-シークエンシング法により解析した.その結果,前駆脂肪細胞において7005のSETDB1結合領域が見い出された.その約半分は遺伝子の転写領域に存在し,多くは転写開始点の下流1 kb以内に存在した.転写を活性化するヒストンH3のLys4のトリメチル化修飾と転写を抑制するヒストンH3のLys9のトリメチル化修飾は同じヌクレオソームには存在しないと考えられている4).しかし,前駆脂肪細胞においてヒストンH3のLys4のトリメチル化修飾をもつ約1万の遺伝子のうち,約400の遺伝子はヒストンH3のLys9のトリメチル化修飾ももっていた.ヒストンH3のLys4のトリメチル化修飾およびヒストンH3のLys9のトリメチル化修飾をもつ遺伝子のうち,約200の遺伝子においてSETDB1との結合も認められた.このなかには発生に関与する遺伝子が濃縮されており,脂肪細胞の分化のマスター遺伝子であるPparg遺伝子およびCebpa遺伝子も含まれていた.Pparg遺伝子およびCebpa遺伝子は特有のヒストン修飾のパターンをもち,転写開始点の上流においてヒストンH3のLys4のトリメチル化修飾,転写開始点の下流においてヒストンH3のLys9のトリメチル化修飾をもち,ヒストンH3のLys9のトリメチル化修飾部位にはSETDB1が局在していた.間葉系幹細胞においても,ヒストンH3のLys4のトリメチル化修飾およびヒストンH3のLys9のトリメチル化修飾をもつ遺伝子のうち,Cebpa遺伝子を含む約200の遺伝子においてSETDB1との結合が認められた.これらの結果から,運命決定された間葉系幹細胞および前駆脂肪細胞において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインが存在することが示された.
2.SETDB1は間葉系幹細胞および前駆脂肪細胞においてヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインの形成に関与する
マイクロアレイ解析の結果,SETDB1のノックダウンにより129の遺伝子の発現が誘導され,そのうち,Pparg遺伝子およびCebpa遺伝子を含む10の遺伝子にはSETDB1が結合することが明らかにされた.SETDB1のノックダウンによりSETDB1の結合する遺伝子の一部のみで発現が誘導されたことから,転写を抑制するヒストンH3のLys9のトリメチル化修飾の消失だけではなく,転写因子やクロマチンタンパク質のはたらきも遺伝子発現の誘導には必要であると考えられた.成熟した脂肪細胞においてはSETDB1の発現が低下し,Pparg遺伝子およびCebpa遺伝子の転写開始点の下流にはヒストンH3のLys9のトリメチル化修飾は存在しなかった.また,前駆脂肪細胞においてSETDB1をノックダウンすると,Pparg遺伝子およびCebpa遺伝子の転写開始点の下流のヒストンH3のLys9のトリメチル化修飾は消失した.これらの結果から,SETDB1はヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインの形成にかかわることが示唆された.
3.ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインはプロモーターの近傍においてRNAポリメラーゼIIの停滞を維持する
ES細胞においてヒストンH3のLys27のトリメチル化修飾はRNAポリメラーゼIIの停滞に関与することが知られている.前駆脂肪細胞においては,ヒストンH3のLys27のトリメチル化修飾の代わりに,ヒストンH3のLys9のトリメチル化修飾がPparg遺伝子およびCebpa遺伝子においてRNAポリメラーゼIIの停滞に関与している可能性が考えられた.前駆脂肪細胞のCebpa遺伝子においては転写を活性化するヒストンH3のLys4のトリメチル化修飾は転写開始点の上流にのみにみられ,遺伝子の転写領域には認められなかった.一方,Pparg遺伝子およびCebpa遺伝子の発現している成熟した脂肪細胞においては,ヒストンH3のLys4のトリメチル化修飾は転写開始点の上流だけでなく遺伝子の転写領域にも認められた.SETDB1のノックダウンにより遺伝子の転写領域におけるヒストンH3のLys9のトリメチル化修飾が消失すると,Pparg遺伝子およびCebpa遺伝子において遺伝子の転写領域にもヒストンH3のLys4のトリメチル化修飾が認められるようになった.これにともない,Pparg遺伝子およびCebpa遺伝子において遺伝子の転写領域にRNAポリメラーゼIIが検出された.
C/EBPβは脂肪細胞分化を誘導する初期の転写因子であり,Cebpa遺伝子の発現を活性化することが知られている.通常,C/EBPβは分化を誘導する刺激ののち14時間まではほとんど標的となる遺伝子には結合しないことから,Cebpa遺伝子におけるヒストンH3のLys9のトリメチル化修飾がC/EBPβのリクルートをさまたげている可能性について検証した.SETDB1のノックダウンにより,通常はほとんどCebpa遺伝子に結合しないC/EBPβの結合が増加した.さらに,SETDB1のノックダウンにより,クロマチンの開いた構造の特徴であるヒストンH3のLys27のアセチル化修飾が増加した.これらの結果から,前駆脂肪細胞におけるSETDB1によるCebpa遺伝子のヒストンH3のLys9のトリメチル化修飾は,C/EBPβの結合,ヒストンのアセチル化,ヒストンH3のLys4のトリメチル化修飾の遺伝子の転写領域への広がりをさまたげ,RNAポリメラーゼIIを転写開始点に停滞させることが示された.
4.SETDB1のリクルートは細胞系譜に特異的なDNAメチル化と同時に起こる
SETDB1結合領域のモチーフを解析したところCGを含む塩基配列の濃縮が認められ,SETDB1のリクルートとCpGメチル化にはなんらかの関係があると考えられた.前駆脂肪細胞においてCpGメチル化について解析したところ,転写開始点の前後2 kbに存在するSETDB1結合領域の約60%がCpGメチル化されていた.ES細胞では,Cebpa遺伝子において遺伝子の転写領域はCpGメチル化されておらず,SETDB1はリクルートされていなかった.一方,間葉系幹細胞および前駆脂肪細胞において,Cebpa遺伝子の遺伝子の転写領域はCpGメチル化されており,SETDB1がリクルートされていた.これらの結果から,SETDB1のリクルートと細胞系譜に特異的なDNAメチル化は同時に起こることが示唆された.
5.Cebpa遺伝子のDNAメチル化はSETDB1-MBD1複合体をリクルートする
SETDB1はCpGメチル化部位の結合タンパク質であるMBD1と相互作用することが知られている11).前駆脂肪細胞において,MBD1の結合についてクロマチン免疫沈降-シークエンシング法により解析したところ,SETDB1結合領域の約80%はMBD1結合領域と一致した.Cebpa遺伝子において,CpGメチル化,MBD1,SETDB1は共局在していた.MBD1のノックダウンにより,Cebpa遺伝子の発現は2~3倍にも増強され,脂肪滴の蓄積が促進された.また,MBD1のノックダウンにより,Cebpa遺伝子へのSETDB1のリクルートおよびヒストンH3のLys9のトリメチル化修飾は減少した.SETDB1のリクルートおよびヒストンH3のLys9のトリメチル化修飾の減少は野生型のMBD1の発現により回復したが,CpGメチル化部位との結合ドメインに変異をもつMBD1の発現では回復しなかった.これらの結果から,MBD1はCebpa遺伝子のCpGメチル化部位へSETDB1をリクルートし,ヒストンH3のLys9のメチル化修飾および遺伝子の発現を抑制することが示された.
おわりに
この研究において,筆者らは,運命決定された前駆脂肪細胞の約200の遺伝子において,転写開始点の上流にヒストンH3のLys4のトリメチル化修飾,転写開始点の下流にヒストンH3のLys9のトリメチル化修飾をもつクロマチンドメインを見い出した.細胞系譜に特異的な遺伝子の転写領域のDNAメチル化およびヒストンH3のLys9のメチル化酵素であるSETDB1のリクルートにより,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインが形成された.このクロマチンドメインはRNAポリメラーゼIIを転写開始点の上流に停滞させ遺伝子の発現を低く維持する一方で,分化を誘導する刺激によりクロマチンを開いた構造へと変化させ,転写因子やクロマチンタンパク質と共役して転写をすみやかに誘導する役割をもっていた(図1).
ヒストンH3のLys4およびLys27にトリメチル化修飾をもつクロマチンドメインはES細胞において発生にかかわる約2500の遺伝子の発現を低く保ち多分化能の維持にかかわる.このクロマチンドメインにおいて,転写を活性化するヒストンH3のLys4のトリメチル化修飾と転写を抑制するヒストンH3のLys27のトリメチル化修飾は転写開始点の近傍の同じあるいは隣接するヌクレオソームに存在する.一方,この研究において見い出されたヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインは,間葉系幹細胞あるいは前駆脂肪細胞においてわずか約200の遺伝子にて形成されていた.このクロマチンドメインにおいては,転写開始点の上流にヒストンH3のLys4のトリメチル化修飾,転写開始点の下流にヒストンH3のLys9のトリメチル化修飾が直列して存在し,ひとつの遺伝子において開いた構造のクロマチンと閉じた構造のクロマチンが共存していると考えられた.このクロマチンドメインは転写開始点の下流における細胞系譜に特異的なDNAメチル化とヒストンH3のLys9のメチル化酵素であるSETDB1のリクルートとが組み合わさることにより形成されたことから,発生にかかわる特定の遺伝子を特定の時期だけ制御するためのしくみと考えられた.
脂肪細胞への運命決定の過程において,ヒストンH3のLys4およびLys9にトリメチル化修飾をもつクロマチンドメインが前駆細胞にふみとどまらせる役割をもつことが見い出されたが,同様のクロマチンドメインが神経細胞,筋細胞,骨細胞といったさまざまな細胞の運命決定の過程において形成され終末分化が必要なときまで前駆細胞を維持している可能性が考えられる.今後,細胞系譜に特異的なDNAメチル化のパターンがどのように形成され,クロマチンの局所的な構造が遺伝子の発現をどのように動的に制御するかについて理解がもとめられる.
文 献
- Tee, W. W. & Reinberg, D.: Chromatin features and the epigenetic regulation of pluripotency states in ESCs. Development, 141, 2376-2390 (2014)[PubMed]
- Margueron, R., Trojer, P. & Reinberg, D.: The key to development: interpreting the histone code? Curr. Opin. Genet. Dev., 15, 163-176 (2005)[PubMed]
- Bernstein, B. E., Mikkelsen, T. S., Xie, X. et al.: A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell, 125, 315-326 (2006)[PubMed]
- Mikkelsen, T. S., Ku, M., Jaffe, D. B. et al.: Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature, 448, 553-560 (2007)[PubMed]
- Grewal, S. I. & Elgin, S. C.: Heterochromatin: new possibilities for the inheritance of structure. Curr. Opin. Genet. Dev., 12, 178-187 (2002)[PubMed]
- Rugg-Gunn, P. J., Cox, B. J., Ralston, A. et al.: Distinct histone modifications in stem cell lines and tissue lineages from the early mouse embryo. Proc. Natl. Acad. Sci. USA, 107, 10783-10790 (2010)[PubMed]
- Farmer, S. R.: Transcriptional control of adipocyte formation. Cell Metab., 4, 263-273 (2006)[PubMed]
- Mikkelsen, T. S., Xu, Z., Zhang, X. et al.: Comparative epigenomic analysis of murine and human adipogenesis. Cell, 143, 156-169 (2010)[PubMed]
- Tang, Q. Q. & Lane, M. D.: Activation and centromeric localization of CCAAT/enhancer-binding proteins during the mitotic clonal expansion of adipocyte differentiation. Genes Dev., 13, 2231-2241 (1999)[PubMed]
- Wakabayashi, K., Okamura, M., Tsutsumi, S. et al.: The peroxisome proliferator-activated receptor γ/retinoid X receptor α heterodimer targets the histone modification enzyme PR-Set7/Setd8 gene and regulates adipogenesis through a positive feedback loop. Mol. Cell. Biol., 29, 3544-3555 (2009)[PubMed]
- Sarraf, S. A. & Stancheva, I.: Methyl-CpG binding protein MBD1 couples histone H3 methylation at lysine 9 by SETDB1 to DNA replication and chromatin assembly. Mol. Cell, 15, 595-605 (2004)[PubMed]
著者プロフィール
略歴:2004年 東北大学大学院理学研究科 修了,同年 秋田大学医学部 研究員,2008年 米国Oregon Health & Science大学 研究員を経て,2011年より東京大学先端科学技術研究センター 特任助教(現 助教).
研究テーマ:肥満および生活習慣病におけるエピゲノムの役割.
抱負:DNAとタンパク質との複合体の解析から複雑な生命現象を解明したい.
酒井 寿郎(Juro Sakai)
東京大学先端科学技術研究センター 教授.
研究室URL:http://www.mm.rcast.u-tokyo.ac.jp
© 2015 松村欣宏・酒井寿郎 Licensed under CC 表示 2.1 日本