Rif1はグアニン4重鎖構造を形成するDNAを介して染色体へ結合し複製の開始を広範囲にわたり抑制する
加納 豊・松本清治・正井久雄
(東京都医学総合研究所 ゲノム動態プロジェクト)
email:正井久雄
DOI: 10.7875/first.author.2015.127
Rif1 binds to G quadruplexes and suppresses replication over long distances.
Yutaka Kanoh, Seiji Matsumoto, Rino Fukatsu, Naoko Kakusho, Nobuaki Kono, Claire Renard-Guillet, Koji Masuda, Keisuke Iida, Kazuo Nagasawa, Katsuhiko Shirahige, Hisao Masai
Nature Structural & Molecular Biology, 22, 889-897 (2015)
真核生物のゲノムは効率のよい複製のために複数の複製起点をもち,これら多くの複製起点はS期をつうじて秩序正しく活性化される.複製タイミングとよばれるこの制御にはさまざまなタンパク質が関与する.以前に,筆者らは,Rif1という進化的に保存されているタンパク質が複製タイミングの制御に中心的な役割をはたすことを見い出した.今回,Rif1結合部位をゲノムワイドに決定したところ,コンセンサス配列が見い出された.そこには複数のグアニンリピートが含まれており,Rif1はこのグアニンリピートにより形成されるグアニン4重鎖構造に結合することがわかった.そして,染色体に結合したRif1はその周辺の約100 kbにわたり複製の開始を抑制することが示された.これらの結果から,Rif1とグアニン4重鎖構造を形成するDNAとの相互作用により,複製の開始に抑制的な複製ドメインが構築されることが示唆された.
細胞はその設計図である染色体DNAを次世代に伝達するため,その正確なコピーを作製しなくてはならない.ヒトにおいては,60億塩基対という膨大な量のDNAを約8時間で複製する.ゲノムの大きい真核生物は複数の複製起点を使用し,秩序正しく複製を完遂する.発生の段階では複製起点が一斉に活性化され,より短時間に複製を完了する場合もある.分化が進み転写やさまざまな染色体の機能が稼働しはじめると,異なる複製起点からの複製開始のタイミングにずれが生じはじめ,細胞種ごとに既定されたタイミングのもと,特定のゲノムの領域が複製されるようになる.筆者らは,この制御を“複製タイミング”とよんでいる.複製開始のタイミングの異なる複製起点のセットを得ることにより,複製の途中にDNA損傷などの問題が発生したとき,未開始の複製起点を抑制することより複製の進行を遅らせ,問題の解消までの時間をかせぐことができる.このシステムを複製チェックポイントとよぶ1).
筆者らは,分裂酵母のスクリーニングにより,複製タイミングの制御において中心的な役割をはたすタンパク質としてRif1を発見した2,3).Rif1の欠損株においては後期の複製起点の広範な活性化および初期の複製起点の活性化の低下が観察された.さらに,Rif1は動物細胞においても複製タイミングを制御することが見い出された.動物細胞においてRif1をノックダウンしたところクロマチンループが伸長したことから,Rif1は核において染色体と結合し,その折りたたみ構造に影響をおよぼしていることが想像された(図1).今回の研究においては,Rif1の染色体への結合について詳細に解析した.
分裂酵母において複製タイミングを制御するタンパク質をスクリーニングすることにより,テロメア長の制御タンパク質として発見されたRif1が,ゲノムワイドにおける複製タイミングの制御に必要であることが見い出された.ChIP-chip法により決定された1100個の複製起点のうち,500個はS期の初期には抑制されているが,Rif1の欠損株では,この500個の複製起点のうち100個以上が脱抑制されており,ヌクレオチド合成酵素の阻害剤の存在のもとで活性化されることが判明した.この複製タイミングの変化は,これまで報告されてきた複製タイミングを制御するタンパク質の変異体と比べ,非常に強く一線を画すものであった.また,Rif1は複数の機能をもつタンパク質であるが,複製を制御する機能は酵母からヒトまで保存されており,分裂酵母やヒトを含め,さまざまな生物においてRif1の欠損により複製タイミングの制御が喪失することが報告されている4).さらに,Rif1は2本鎖DNA切断修復の制御にも関与することが報告されている5).Rif1の染色体における結合部位をChIP-seq法により決定した結果,90個の比較的強い結合配列の同定に成功した.この結合配列からコンセンサス配列を探索したところ,よく保存された配列としてCNWWGTGGGGGが見い出された.さらにくわしく解析してみたところ,90個の結合配列のうち35個においては,複数のコンセンサス配列が含まれており,さらに,この35個の結合配列へのRif1の結合はほかの結合配列への結合よりも強いことが判明した.のちの解析から,この35個の結合配列がRif1の主要な結合部位であろうと考えた.分裂酵母の染色体サイズは12.5 Mbであり,そこに存在する35個の結合配列に結合したRif1が100個以上の複製起点に影響をおよぼしている点は興味深い.35個の結合配列から2個を選択してコンセンサス配列に変異を導入し,Rif1の染色体への結合を解析した結果,この2個の結合配列とRif1との結合は消失していた.つまり,このコンセンサス配列がRif1との結合に必須であることが証明された.
in vitroにおいて,Rif1結合配列からなる2本鎖DNAと精製した分裂酵母のRif1が相互作用するかどうか検証したが,特異的な結合は観察されなかった.しかし,Rif1結合配列をより詳細に解析した結果,コンセンサス配列以外にも,グアニンからなるリピートが存在することが判明し,このことから,Rif1結合配列はグアニン4重鎖構造とよばれる特殊な構造をとる可能性が想定された.そこで,Rif1結合配列を熱変性し再アニールした結果,アクリルアミドゲルにおいてグアニン4重鎖構造様の構造を形成することが確認された.さらに,このグアニン4重鎖様の構造に対し,精製Rif1が特異的に結合することもわかった.Rif1結合配列により形成されるこの構造を検証するため,グアニン4重鎖構造をとるDNAに特異的にインターカレートして結合しその構造を安定化するリガンド,L1BOD-7OTD 6) との相互作用について検討した.その結果,このリガンドはRif1結合配列から形成されたDNA構造と結合しこれを安定化した.この結果は,Rif1結合配列はグアニン4重鎖構造を形成していることを支持した.そして,この安定化されたグアニン4重鎖構造を形成したRif1結合配列に対し,精製Rif1が結合した.一方で,コンセンサス配列に変異を導入したRif1結合配列はグアニン4重鎖構造を形成せず,精製Rif1とも相互作用しなかった.また,動物細胞のRif1は十文字構造を形成するDNAと結合するという報告があったが7),競合実験の結果,Rif1はグアニン4重鎖構造に対し,より高い親和性にて結合した.
in vitroにおいて,Rif1結合配列に含まれるグアニンのリピートはグアニン4重鎖構造の形成に必要であることが確認された.さらに,このグアニンリピートに変異をもつRif1結合配列は細胞内においてRif1との結合が減少していた.以上の結果から,Rif1はグアニン4重鎖構造を形成したDNAに結合している可能性が強く支持された.一方で,情報科学的な解析にもとづき分裂酵母においてグアニン4重鎖構造をとりうる可能性のあるDNA配列のリストが報告されているが8),それらとは35個のRif1結合配列のうち4個しか一致しなかった.これは,Rif1結合配列が典型的なグアニン4重鎖構造を形成しないからかもしれないが,塩基配列からグアニン4重鎖構造を予測することの困難さを示しているとも思われた.
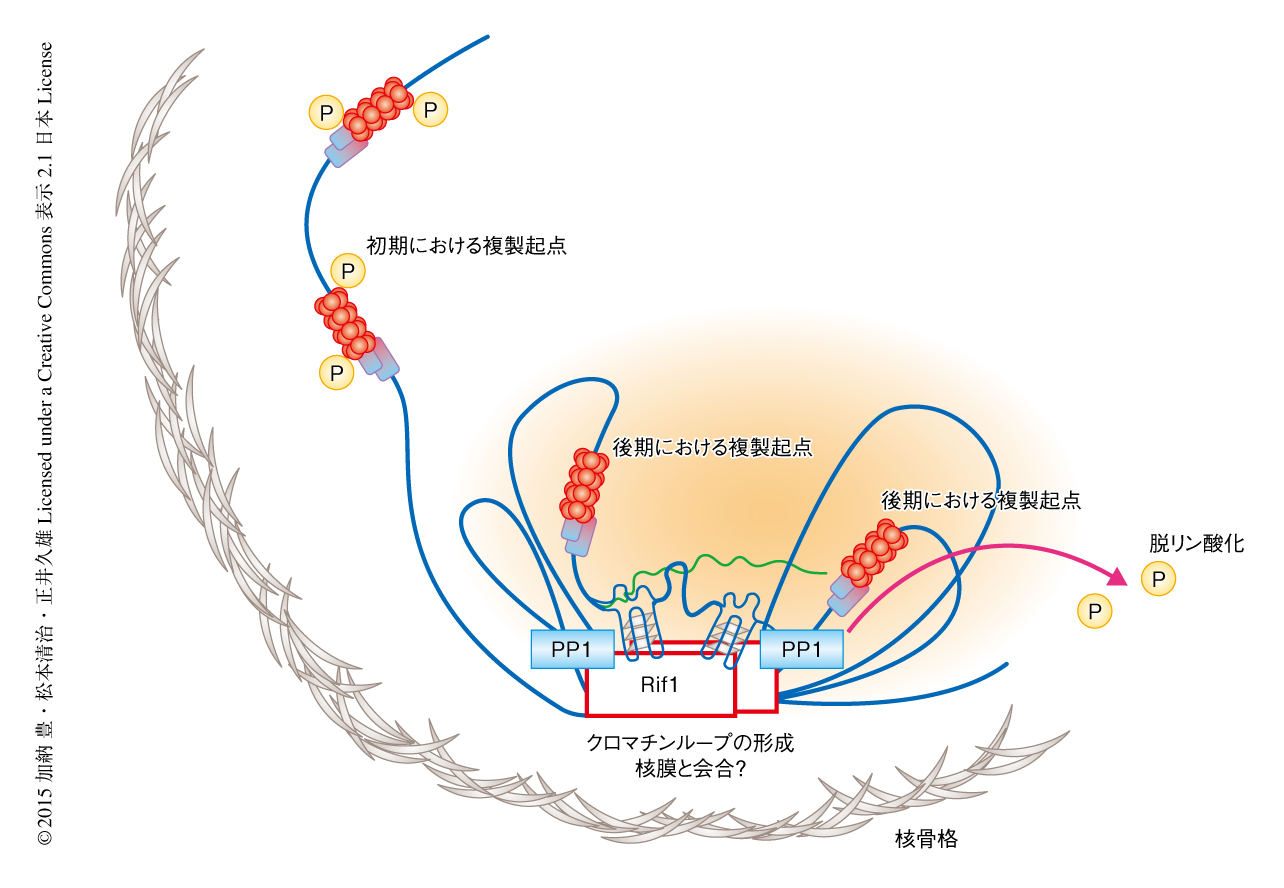
Rif1結合配列に変異をもつ株において染色体とRif1との結合は消失したが,これが複製タイミングに影響をおよぼすかどうか検証した.Rif1結合配列の変異株において,その周辺における複製タイミングの変化について解析したところ,Rif1結合部位から約50 kbも離れた複製起点の活性にも変化がみられた.つまり,Rif1は最大でその周辺の約100 kbまで影響をおよぼす可能性が示唆された.Rif1は核膜の近傍において染色体ファイバー構造をたばねてクロマチンドメインを形成することにより,複製に阻害的な複製ドメインを形成するのかもしれない(図2).実際に,精製Rif1は多量体を形成し,複数のDNA鎖に同時に結合しうることが見い出された.
グアニン4重鎖構造が形成されるためには,少なくとも一過的に2本鎖DNAがほどかれる必要があるが,これまでの解析からRif1はM期の後期からG1期までのあいだにDNAと結合することがわかっていることから,複製によりグアニン4重鎖構造が形成される可能性は除外された.ゲノムのアノテーションから,Rif1結合部位は非コードRNAをコードする領域や遺伝子の3’側非翻訳領域および5’側非翻訳領域に存在することが明らかにされた.このことから,細胞内におけるグアニン4重鎖構造の形成には転写が関与する可能性が示唆された.実際に,in vitroにおいてRif1結合部位を含む2本鎖DNAを転写するとグアニン4重鎖様の構造が形成され,Rif1はこの構造に結合した.このことは,細胞が分化する過程において転写が活性化されるにしたがい,複製ドメインが構築されることと関連しているのかもしれない.今後,分裂酵母やヒトの細胞において核におけるRif1の局在および動態を調べることにより,Rif1による染色体の高次構造の構築についてその詳細が明らかにされることが期待される.そして,それらの染色体の高次構造が複製,転写,組換え・修復などを協調的に制御する可能性についても研究が進むと期待される.
グアニン4重鎖構造はヒトのゲノムに37万個以上も存在すると推定されるにもかかわらず9),その普遍的な生物学的な意義はほとんど不明であった.今回の発見により,グアニン4重鎖構造が遺伝子間領域に存在し,Rif1により認識されて染色体の高次構造の形成に関与する可能性が強く示唆された.すでに,動物細胞のRif1も同様にグアニン4重鎖構造と特異的に結合することが見い出されており,Rif1によるグアニン4重鎖の認識と高次構造の高次構造の形成は種をこえて保存されているようである.したがって,この研究はグアニン4重鎖構造の普遍的な生物学的な役割の一端にせまったものといえる.
略歴:2006年 横浜市立大学大学院総合理学研究科 修了,同年 東京都臨床医学総合研究所 研究員を経て,2012年より東京都医学総合研究所 主任研究員.
研究テーマ:Rif1による真核生物における複製の制御機構.
関心事:次世代シークエンサーのデータの解析手法を習得して,思いどおりの解析がしたい.
松本 清治(Seiji Matsumoto)
東京都医学総合研究所 主任研究員.
正井 久雄(Hisao Masai)
東京都医学総合研究所 プロジェクトリーダー.
研究室URL:http://www.igakuken.or.jp/genome/
© 2015 加納 豊・松本清治・正井久雄 Licensed under CC 表示 2.1 日本
(東京都医学総合研究所 ゲノム動態プロジェクト)
email:正井久雄
DOI: 10.7875/first.author.2015.127
Rif1 binds to G quadruplexes and suppresses replication over long distances.
Yutaka Kanoh, Seiji Matsumoto, Rino Fukatsu, Naoko Kakusho, Nobuaki Kono, Claire Renard-Guillet, Koji Masuda, Keisuke Iida, Kazuo Nagasawa, Katsuhiko Shirahige, Hisao Masai
Nature Structural & Molecular Biology, 22, 889-897 (2015)
要 約
真核生物のゲノムは効率のよい複製のために複数の複製起点をもち,これら多くの複製起点はS期をつうじて秩序正しく活性化される.複製タイミングとよばれるこの制御にはさまざまなタンパク質が関与する.以前に,筆者らは,Rif1という進化的に保存されているタンパク質が複製タイミングの制御に中心的な役割をはたすことを見い出した.今回,Rif1結合部位をゲノムワイドに決定したところ,コンセンサス配列が見い出された.そこには複数のグアニンリピートが含まれており,Rif1はこのグアニンリピートにより形成されるグアニン4重鎖構造に結合することがわかった.そして,染色体に結合したRif1はその周辺の約100 kbにわたり複製の開始を抑制することが示された.これらの結果から,Rif1とグアニン4重鎖構造を形成するDNAとの相互作用により,複製の開始に抑制的な複製ドメインが構築されることが示唆された.
はじめに
細胞はその設計図である染色体DNAを次世代に伝達するため,その正確なコピーを作製しなくてはならない.ヒトにおいては,60億塩基対という膨大な量のDNAを約8時間で複製する.ゲノムの大きい真核生物は複数の複製起点を使用し,秩序正しく複製を完遂する.発生の段階では複製起点が一斉に活性化され,より短時間に複製を完了する場合もある.分化が進み転写やさまざまな染色体の機能が稼働しはじめると,異なる複製起点からの複製開始のタイミングにずれが生じはじめ,細胞種ごとに既定されたタイミングのもと,特定のゲノムの領域が複製されるようになる.筆者らは,この制御を“複製タイミング”とよんでいる.複製開始のタイミングの異なる複製起点のセットを得ることにより,複製の途中にDNA損傷などの問題が発生したとき,未開始の複製起点を抑制することより複製の進行を遅らせ,問題の解消までの時間をかせぐことができる.このシステムを複製チェックポイントとよぶ1).
筆者らは,分裂酵母のスクリーニングにより,複製タイミングの制御において中心的な役割をはたすタンパク質としてRif1を発見した2,3).Rif1の欠損株においては後期の複製起点の広範な活性化および初期の複製起点の活性化の低下が観察された.さらに,Rif1は動物細胞においても複製タイミングを制御することが見い出された.動物細胞においてRif1をノックダウンしたところクロマチンループが伸長したことから,Rif1は核において染色体と結合し,その折りたたみ構造に影響をおよぼしていることが想像された(図1).今回の研究においては,Rif1の染色体への結合について詳細に解析した.
1.Rif1は保存された配列に依存して染色体と結合する
分裂酵母において複製タイミングを制御するタンパク質をスクリーニングすることにより,テロメア長の制御タンパク質として発見されたRif1が,ゲノムワイドにおける複製タイミングの制御に必要であることが見い出された.ChIP-chip法により決定された1100個の複製起点のうち,500個はS期の初期には抑制されているが,Rif1の欠損株では,この500個の複製起点のうち100個以上が脱抑制されており,ヌクレオチド合成酵素の阻害剤の存在のもとで活性化されることが判明した.この複製タイミングの変化は,これまで報告されてきた複製タイミングを制御するタンパク質の変異体と比べ,非常に強く一線を画すものであった.また,Rif1は複数の機能をもつタンパク質であるが,複製を制御する機能は酵母からヒトまで保存されており,分裂酵母やヒトを含め,さまざまな生物においてRif1の欠損により複製タイミングの制御が喪失することが報告されている4).さらに,Rif1は2本鎖DNA切断修復の制御にも関与することが報告されている5).Rif1の染色体における結合部位をChIP-seq法により決定した結果,90個の比較的強い結合配列の同定に成功した.この結合配列からコンセンサス配列を探索したところ,よく保存された配列としてCNWWGTGGGGGが見い出された.さらにくわしく解析してみたところ,90個の結合配列のうち35個においては,複数のコンセンサス配列が含まれており,さらに,この35個の結合配列へのRif1の結合はほかの結合配列への結合よりも強いことが判明した.のちの解析から,この35個の結合配列がRif1の主要な結合部位であろうと考えた.分裂酵母の染色体サイズは12.5 Mbであり,そこに存在する35個の結合配列に結合したRif1が100個以上の複製起点に影響をおよぼしている点は興味深い.35個の結合配列から2個を選択してコンセンサス配列に変異を導入し,Rif1の染色体への結合を解析した結果,この2個の結合配列とRif1との結合は消失していた.つまり,このコンセンサス配列がRif1との結合に必須であることが証明された.
2.Rif1結合配列はグアニン4重鎖構造を形成しRif1はこの構造に特異的に結合する
in vitroにおいて,Rif1結合配列からなる2本鎖DNAと精製した分裂酵母のRif1が相互作用するかどうか検証したが,特異的な結合は観察されなかった.しかし,Rif1結合配列をより詳細に解析した結果,コンセンサス配列以外にも,グアニンからなるリピートが存在することが判明し,このことから,Rif1結合配列はグアニン4重鎖構造とよばれる特殊な構造をとる可能性が想定された.そこで,Rif1結合配列を熱変性し再アニールした結果,アクリルアミドゲルにおいてグアニン4重鎖構造様の構造を形成することが確認された.さらに,このグアニン4重鎖様の構造に対し,精製Rif1が特異的に結合することもわかった.Rif1結合配列により形成されるこの構造を検証するため,グアニン4重鎖構造をとるDNAに特異的にインターカレートして結合しその構造を安定化するリガンド,L1BOD-7OTD 6) との相互作用について検討した.その結果,このリガンドはRif1結合配列から形成されたDNA構造と結合しこれを安定化した.この結果は,Rif1結合配列はグアニン4重鎖構造を形成していることを支持した.そして,この安定化されたグアニン4重鎖構造を形成したRif1結合配列に対し,精製Rif1が結合した.一方で,コンセンサス配列に変異を導入したRif1結合配列はグアニン4重鎖構造を形成せず,精製Rif1とも相互作用しなかった.また,動物細胞のRif1は十文字構造を形成するDNAと結合するという報告があったが7),競合実験の結果,Rif1はグアニン4重鎖構造に対し,より高い親和性にて結合した.
in vitroにおいて,Rif1結合配列に含まれるグアニンのリピートはグアニン4重鎖構造の形成に必要であることが確認された.さらに,このグアニンリピートに変異をもつRif1結合配列は細胞内においてRif1との結合が減少していた.以上の結果から,Rif1はグアニン4重鎖構造を形成したDNAに結合している可能性が強く支持された.一方で,情報科学的な解析にもとづき分裂酵母においてグアニン4重鎖構造をとりうる可能性のあるDNA配列のリストが報告されているが8),それらとは35個のRif1結合配列のうち4個しか一致しなかった.これは,Rif1結合配列が典型的なグアニン4重鎖構造を形成しないからかもしれないが,塩基配列からグアニン4重鎖構造を予測することの困難さを示しているとも思われた.
3.Rif1はその結合部位により広範囲な領域にわたり複製の開始を抑制する
Rif1結合配列に変異をもつ株において染色体とRif1との結合は消失したが,これが複製タイミングに影響をおよぼすかどうか検証した.Rif1結合配列の変異株において,その周辺における複製タイミングの変化について解析したところ,Rif1結合部位から約50 kbも離れた複製起点の活性にも変化がみられた.つまり,Rif1は最大でその周辺の約100 kbまで影響をおよぼす可能性が示唆された.Rif1は核膜の近傍において染色体ファイバー構造をたばねてクロマチンドメインを形成することにより,複製に阻害的な複製ドメインを形成するのかもしれない(図2).実際に,精製Rif1は多量体を形成し,複数のDNA鎖に同時に結合しうることが見い出された.
おわりに
グアニン4重鎖構造が形成されるためには,少なくとも一過的に2本鎖DNAがほどかれる必要があるが,これまでの解析からRif1はM期の後期からG1期までのあいだにDNAと結合することがわかっていることから,複製によりグアニン4重鎖構造が形成される可能性は除外された.ゲノムのアノテーションから,Rif1結合部位は非コードRNAをコードする領域や遺伝子の3’側非翻訳領域および5’側非翻訳領域に存在することが明らかにされた.このことから,細胞内におけるグアニン4重鎖構造の形成には転写が関与する可能性が示唆された.実際に,in vitroにおいてRif1結合部位を含む2本鎖DNAを転写するとグアニン4重鎖様の構造が形成され,Rif1はこの構造に結合した.このことは,細胞が分化する過程において転写が活性化されるにしたがい,複製ドメインが構築されることと関連しているのかもしれない.今後,分裂酵母やヒトの細胞において核におけるRif1の局在および動態を調べることにより,Rif1による染色体の高次構造の構築についてその詳細が明らかにされることが期待される.そして,それらの染色体の高次構造が複製,転写,組換え・修復などを協調的に制御する可能性についても研究が進むと期待される.
グアニン4重鎖構造はヒトのゲノムに37万個以上も存在すると推定されるにもかかわらず9),その普遍的な生物学的な意義はほとんど不明であった.今回の発見により,グアニン4重鎖構造が遺伝子間領域に存在し,Rif1により認識されて染色体の高次構造の形成に関与する可能性が強く示唆された.すでに,動物細胞のRif1も同様にグアニン4重鎖構造と特異的に結合することが見い出されており,Rif1によるグアニン4重鎖の認識と高次構造の高次構造の形成は種をこえて保存されているようである.したがって,この研究はグアニン4重鎖構造の普遍的な生物学的な役割の一端にせまったものといえる.
文 献
- Masai, H., Matsumoto, S., You, Z. et al.: Eukaryotic chromosome DNA replication: where, when, and how? Annu. Rev. Biochem., 79, 89-130 (2010)[PubMed]
- Hayano, M., Kanoh, Y., Matsumoto, S. et al.: Rif1 is a global regulator of timing of replication origin firing in fission yeast. Genes Dev., 26, 137-150 (2012)[PubMed]
- Yamazaki, S., Ishii, A., Kanoh, Y. et al.: Rif1 regulates the replication timing domains on the human genome. EMBO J., 31, 3667-3677 (2012)[PubMed]
- Peace, J. M., Ter-Zakarian, A. & Aparicio, O. M.: Rif1 regulates initiation timing of late replication origins throughout the S. cerevisiae genome. PLoS One, 9, e98501 (2014)[PubMed]
- Zimmermann, M., Lottersberger, F., Buonomo, S. B. et al.: 53BP1 regulates DSB repair using Rif1 to control 5’ end resection. Science, 339, 700-704 (2013)[PubMed]
- Iida, K., Nakamura, T., Yoshida, W. et al.: Fluorescent-ligand-mediated screening of G-quadruplex structures using a DNA microarray. Angew. Chem. Int. Ed. Engl., 52, 12052-12055 (2013)[PubMed]
- Sukackaite, R., Jensen, M. R., Mas, P. J. et al.: Structural and biophysical characterization of murine Rif1 C terminus reveals high specificity for DNA cruciform structures. J. Biol. Chem., 289, 13903-13911 (2014)[PubMed]
- Sabouri, N., Capra, J. A. & Zakian, V. A.: The essential Schizosaccharomyces pombe Pfh1 DNA helicase promotes fork movement past G-quadruplex motifs to prevent DNA damage. BMC Biol., 12, 101 (2014)[PubMed]
- Huppert, J. L. & Balasubramanian, S.: Prevalence of quadruplexes in the human genome. Nucleic Acids Res., 33, 2908-2916 (2005)[PubMed]
著者プロフィール
略歴:2006年 横浜市立大学大学院総合理学研究科 修了,同年 東京都臨床医学総合研究所 研究員を経て,2012年より東京都医学総合研究所 主任研究員.
研究テーマ:Rif1による真核生物における複製の制御機構.
関心事:次世代シークエンサーのデータの解析手法を習得して,思いどおりの解析がしたい.
松本 清治(Seiji Matsumoto)
東京都医学総合研究所 主任研究員.
正井 久雄(Hisao Masai)
東京都医学総合研究所 プロジェクトリーダー.
研究室URL:http://www.igakuken.or.jp/genome/
© 2015 加納 豊・松本清治・正井久雄 Licensed under CC 表示 2.1 日本