テトラヒメナにおけるDNAの削減は小分子RNAによるトランス認識ネットワークにより制御されている
能登朋子・片岡研介・望月一史
(オーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciences)
email:望月一史
DOI: 10.7875/first.author.2015.093
Small-RNA-mediated genome-wide trans-recognition network in Tetrahymena DNA elimination.
Tomoko Noto, Kensuke Kataoka, Jan H. Suhren, Azusa Hayashi, Katrina J. Woolcock, Martin A. Gorovsky, Kazufumi Mochizuki
Molecular Cell, 59, 229-242 (2015)
多くの真核生物は小分子RNAを共通してトランスポゾンの抑制に用いている.しかし,小分子RNAをどのように宿主のゲノムとトランスポゾンとの識別に用いるかについては多様性がある.繊毛虫類に属するテトラヒメナは小分子RNAを用いて小核と大核のゲノムを比較することによりトランスポゾンに由来する配列を認識し,DNAの削減によりそのDNA自体を大核から取り除くというユニークなトランスポゾンの抑制の戦略をとる.この論文では,この小核と大核の比較にくわえ,一部のかぎられたIESから形成された小分子RNAがほかのIESをトランスに認識し,その認識が新奇の小分子RNAの合成の連鎖反応をひき起こすことによりDNAの削減の堅牢性が確保されていることを示した.
トランスポゾンは宿主のゲノムの安定性をみだす脅威であり,多くの真核生物は共通してRNAiやそれに類する機構により形成される20~30塩基の小分子RNAをトランスポゾンの抑制に用いている.しかし,トランスポゾンのみに小分子RNAをはたらかせる方法については生物種のあいだで多様性がみられる.なかでも,テトラヒメナがトランスポゾンを識別する戦略はひときわユニークである.
繊毛虫類に属する単細胞真核生物であるテトラヒメナは同一の細胞に小核および大核とよばれる異なる2つの核をもつ1).小核は転写活性を欠く生殖核で,大核は転写活性をもつ栄養核である.有性生殖の過程において大核は小核から分化し,その際にDNAの削減によりゲノムの約1/3(約50 Mb)が除去される.この除去される配列は0.5~40 kbの約1万箇所の領域に分かれて小核のゲノムに散在しており,IES(internal eliminated sequence)とよばれる.既知のトランスポゾンはすべてIESに存在することから,DNAの削減は転写活性をもつ大核からトランスポゾンをDNA配列ごと除去する過程と考えられる.
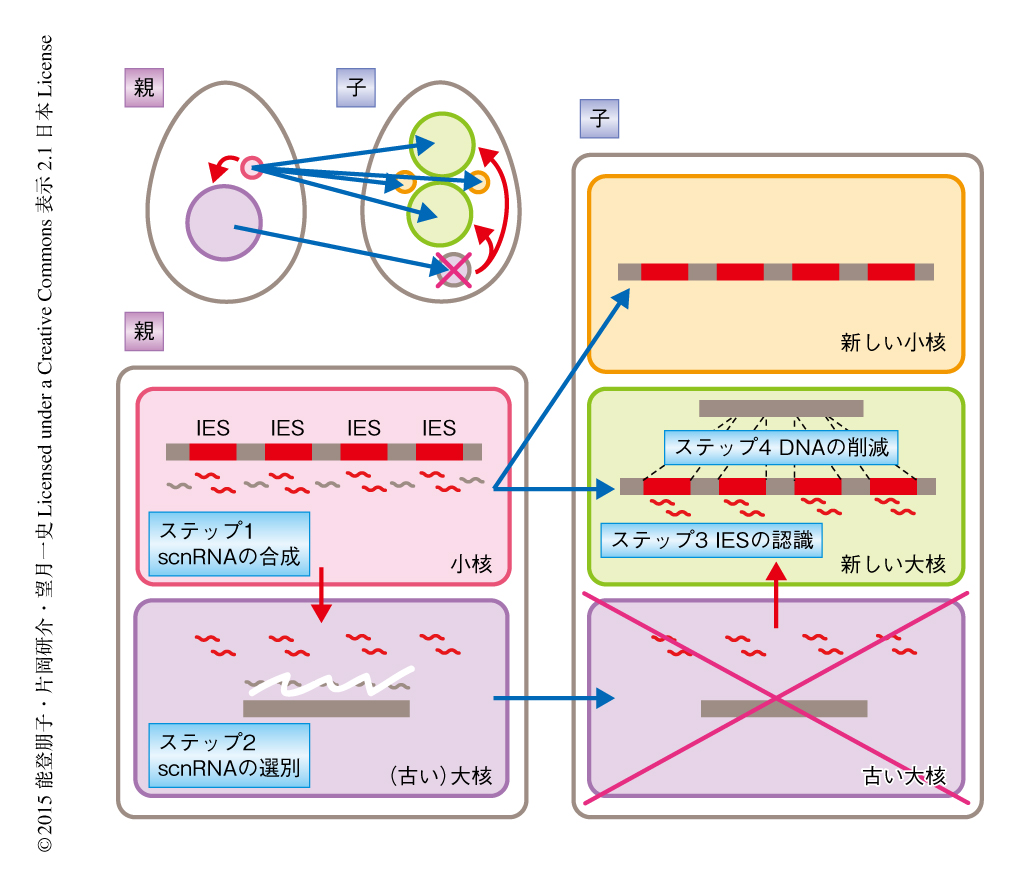
以前に,筆者らは,少なくとも一部のIESは小分子RNAを用いた大核と小核のゲノムの差し引きにより小核にのみ存在する配列として認識されていることを報告した2,3).それにもとづいたモデル(図1)では,まず,ふだんは転写活性をもたない小核が有性生殖の初期にのみ転写され,そこから小核のゲノム全体を写し取った約29塩基の小分子RNA(scnRNA)が形成される(ステップ1:scnRNAの合成).つぎに,scnRNAは大核へと移動し,大核のゲノムに相補的なscnRNAは分解される(ステップ2:scnRNAの選別).分解からのがれたscnRNAは新たに形成された大核へと移動し,それに相補的な配列をIESとして認識する(ステップ3:IESの認識).IESはヘテロクロマチン化され最終的に除去される(ステップ4:DNAの削減).このような2つのゲノムの差し引きのみによりすべてのIESを特定するには,小核のゲノムが一様に転写されscnRNAに写し取られる必要がある.しかし,実際にはscnRNAは小核のゲノムの一部からしか形成されず4),scnRNAを形成しないIESの削減がどのように制御されているのかは不明であった.
実験条件のもとでは,テトラヒメナの有性生殖の開始から新しい大核の形成まで約7 hpm(hpm:hours post mixing),新しい大核におけるDNAの削減の開始まで約14 hpmを要する.また,筆者らによる以前の研究から,scnRNAの合成は3 hpm前後に起こり,scnRNAの選別は新しい大核の形成までに完了することがわかっている3,4).今回,新しい大核の形成ののち,scnRNAとほぼ同じ約29塩基の小分子RNAが合成されていることが見い出された.この有性生殖の後期に発現する新奇の小分子RNAを後期scnRNA,以前から知られていた有性生殖の前期から発現するscnRNAを初期scnRNAとよぶことにした.以前に,初期scnRNAは12個あるArgonauteのうちTwi1pと結合して作用することが報告されている3).一方で,後期scnRNAはTwi1pにくわえ,有性生殖の後期において特異的に発現するTwi11pとも結合していた.初期scnRNAが結合しているTwi1pは有性生殖の前期において古い(有性生殖ののちに分解される)大核から発現していたのに対し,後期scnRNAの結合しているTwi1pおよびTwi11pはともに新しい大核から発現していた.
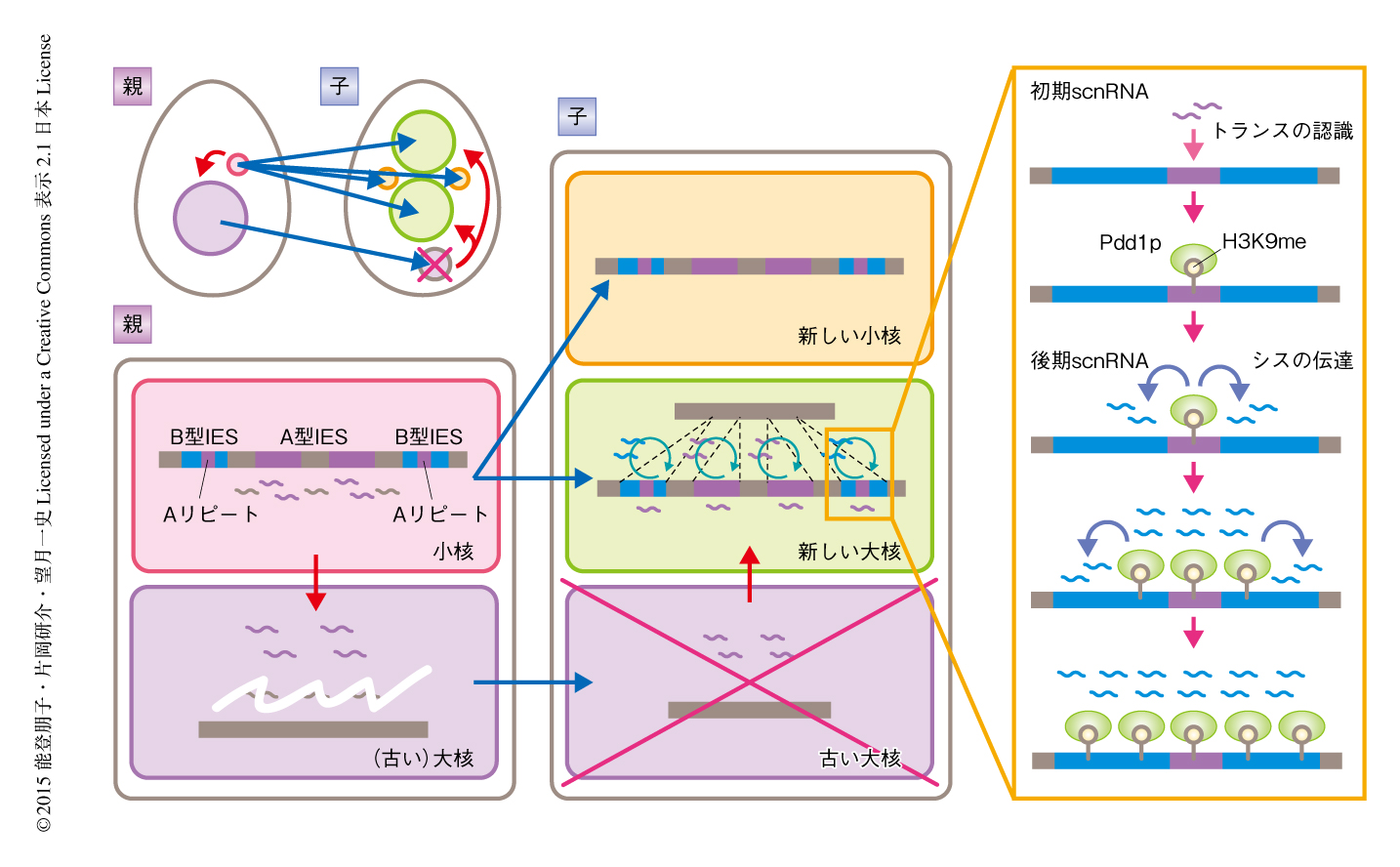
初期scnRNAが一部のIESから形成されIESへの特異性がscnRNAの選別により獲得されるのに対し,後期scnRNAはほぼすべてのIESから形成され合成の段階からIESに対する高い特異性をもつことがわかった.つまり,IESには,初期scnRNAおよび後期scnRNAを発現するものと,後期scnRNAのみを発現するものが存在した.初期scnRNAおよび後期scnRNAを発現するIESをA型IES,後期scnRNAのみを発現するIESをB型IESとよぶことにした.また,ごく少数ではあるが初期scnRNAあるいは後期scnRNAのどちらの発現も認められないIESが存在し,これらをC型IESとよぶことにした.調べた8105個のIESのうち,A型IESが4695個,B型IESが3293個,C型IESが117個あった.
テトラヒメナにおいてはTlr-1とよばれるDNAトランスポゾンとREPファミリーとよばれるレトロトランスポゾンの存在が報告されている.これらのトランスポゾンは転移や逆転写などにかかわる遺伝子が保存されていることから,現在でも転移活性をもつと考えられている.これらのトランスポゾンと相補的な配列はほぼすべてA型IESのみに見い出された.また,小核のゲノム全体のGC含量が22%なのに対し,A型IES,B型IES,C型IESのGC含量は,それぞれ,31%,28%,26%であった.これらの結果,および,後述するB型IESに存在するAリピートの存在は,A型IESは活性をもつトランスポゾンを含む進化的に新しいIESであり,B型IESおよびC型IESはトランスポゾン配列が退化しより“テトラヒメナ化”した進化的に古いIESであることを示唆した.
後期scnRNAのDNAの削減における役割について調べるため,小核のTwi1pをコードする遺伝子およびTwi11pをコードする遺伝子を破壊し,この遺伝子破壊株において有性生殖が終了したのちのDNAの削減について解析した.この遺伝子破壊株では古い大核からはTwi1pが発現するため初期scnRNAは正常に機能したが,小核から形成される新しい大核からはTwi1pおよびTwi11pは発現せず後期scnRNAは不安定かつ機能しなかった.この遺伝子破壊株の子においては多くのB型IESの削減が抑制され,また,A型IESおよびC型IESの削減も部分的に影響をうけていた.よって,後期scnRNAはDNAの削減に必須ではないが,一部のIES,とくにB型IESはDNAの削減において重要な役割をはたすことがわかった.
一方で,古い大核のTwi1pをコードする遺伝子を破壊した場合,初期scnRNAおよび後期scnRNAが失われ,この条件ではIESのタイプにかかわらずすべてのDNAの削減が完全に抑制された.よって,初期scnRNAはすべてのDNAの削減に必須であり,また,後期scnRNAの発現は初期scnRNAに依存することが示された.
ほぼすべてのB型IESにはA型IESに相補的な少なくとも約100塩基の配列が存在していた.このB型IESに存在する配列をAリピートとよぶことにした.A型IESからは初期scnRNAが形成されるため,Aリピートを介しB型IESも初期scnRNAによりトランスに認識されることが予想された.実際に,GFPをコードする遺伝子はIESとは認識されずDNAの削減をうけないが,その真ん中にB型IESのもつAリピートを挿入することによりDNAの削減がひき起こされた.よって,AリピートにはDNAの削減を誘導するシグナル(おそらくは,初期scnRNA)をうけとる能力があると考えられた.
時系列にそって詳細に解析したところ,後期scnRNAの発現はAリピートの付近からはじまり,それがシスに広がって,最終的にB型IESの全体をカバーしていくようすが観察された.任意のA型IESをテトラヒメナの形質転換ベクターにクローニングし,さらに,その中央にIESではない(DNAの削減をうけない)0.5~1 kbほどのテトラヒメナのゲノム配列(標的配列)を挿入した.つぎに,この形質転換ベクターを有性生殖している新しい大核に導入した.すると,標的配列に相補的なゲノム配列がDNAの削減をうけるのが観察され,この現象を共削減(co-deletion)とよぶことにした.共削減は形質転換ベクターに導入されたA型IESや標的配列の種類にかかわらず高い頻度で起こった.また,Twi11pの発現を欠損した変異体では抑制されたことから,後期scnRNAに依存的な現象であると考えられた.共削減は,初期scnRNAが形質転換ベクターに導入されたA型IESを認識し,それが後期scnRNAの発現をシスに誘導することにより標的配列から後期scnRNAが形成され,それが標的配列と相補的な内在性の配列をトランスに認識しDNAの削減を誘導した結果であると考えられた.
IESは初期scnRNAに依存してヘテロクロマチン化され,ヘテロクロマチンが最終的にDNAの削減を誘導する5,6).また,多くの真核生物において,ヘテロクロマチン化はシスに拡散する性質がある.これらのことから,後期scnRNAの発現はIESをシスに拡散するヘテロクロマチン化に依存している可能性が考えられた.そこで,IESのヘテロクロマチン化に必須なヒストンH3のLys9のメチル化酵素であるEzl1p,あるいは,ヒストンH3のLys9のメチル化に特異的に結合するHP1様タンパク質であるPdd1pを欠損した変異体においてscnRNAの発現を観察したところ,予想どおり,いずれの変異体においても初期scnRNAの発現は正常であったが後期scnRNAの発現は消失していた.よって,後期scnRNAの合成にはヘテロクロマチン化が必須であると結論した.
scnRNAによるIESのトランス認識および後期scnRNAの発現のシスの拡散は後期scnRNAの合成の連鎖反応をひき起こすと考えられた.したがって,ゲノムに散在するIESがネットワークを形成し,一部のIESからの初期scnRNAの発現がみだされたとしても,ほかのIESから発現した初期scnRNAによるトランス認識によりDNAの削減は保障されていると予想された.このようなscnRNAによるトランス認識ネットワークのはたらきについて調べるため,初期scnRNAの発現のみだれ,および,それによるIESの認識についてシミュレーションした.まず,ランダムに選んだ一部のA型IESのみが初期scnRNAを発現する状況を想定し,このかぎられた初期scnRNAが残りのどれだけのIESを認識できるか計算した.この結果,1%のA型IESしか初期scnRNAを形成しなかったとしても,これらはすべてのタイプのIESを含む33%のIESをトランスに認識することができ,この33%のIESが後期scnRNAを発現しそれがさらにほかのIESをトランスに認識したとすれば,あわせて84%のIESがscnRNAにより認識されることが示唆された.一方,すべてのIESが一様にscnRNAを発現したとしても,それらは1.3%の遺伝子をコードする領域しか認識しなかった.よって,DNAの削減はscnRNAによるIESのトランス認識ネットワークによりその堅牢性が保障されており,そのネットワークはIES以外の領域には伝達されないよう進化的にデザインされていると考えられた.
この研究,および,これまでの報告を総括すると以下のように考えられる.初期scnRNAはA型IESとその周辺のかぎられた小核のゲノムから発現し,古い大核においてscnRNAの選別をうけたのち,新しい大核へと移動する.そこで,初期scnRNAはA型IESを認識するとともに,Aリピートを介しB型IESを認識する.そののち,A型IESおよびB型IESから後期scnRNAが発現し,最終的にDNAの削減を誘導する(図2).
哺乳類やショウジョウバエなどの後生動物においてはPIWIに結合する小分子RNAであるpiRNAがトランスポゾンの抑制において重要なはたらきをもつ7,8).これらの生物にはpiRNAクラスターとよばれる,トランスポゾンや退化したトランスポゾンが蓄積したゲノム領域が存在し,そこから形成されたpiRNAがゲノムに散在するトランスポゾンを認識し抑制する.また,piRNAがトランスポゾンに由来するRNAに出会うと,ping-pongサイクルとよばれる生合成経路によりトランスポゾンに由来するRNAに相補的なpiRNAが増幅される8,9).今回の研究におけるテトラヒメナのscnRNAによるトランス認識ネットワークは,このpiRNA機構を想起させた.すなわち,A型IESはpiRNAクラスターに,また,後期scnRNAの合成の連鎖反応はping-pongサイクルに相当する役割をもつと考えることができる.後生動物とテトラヒメナという進化的に遠く離れた生物が,piRNAとscnRNAという合成経路の異なる小分子RNAを使い,結果的に戦略的に類似したトランスポゾンの認識の機構を獲得していることは,トランスポゾンという生物に共通する脅威に対する機構の進化を考えるうえで興味深い.
略歴:2003年 金沢大学大学院自然科学研究科 修了,2004年 米国Rochester大学 博士研究員を経て,2006年よりオーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciencesリサーチアソシエイト.
研究テーマ:テトラヒメナでのDNAの削減における小分子RNAの役割.
片岡 研介(Kensuke Kataoka)
略歴:2007年 兵庫県立大学大学院生命理学研究科 修了,同年よりオーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciences博士研究員.
研究テーマ:テトラヒメナでのDNAの削減におけるヘテロクロマチンの役割.
望月 一史(Kazufumi Mochizuki)
オーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciencesグループリーダー.
研究室URL:http://www.imba.oeaw.ac.at/research/kazufumi-mochizuki/
© 2015 能登朋子・片岡研介・望月一史 Licensed under CC 表示 2.1 日本
(オーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciences)
email:望月一史
DOI: 10.7875/first.author.2015.093
Small-RNA-mediated genome-wide trans-recognition network in Tetrahymena DNA elimination.
Tomoko Noto, Kensuke Kataoka, Jan H. Suhren, Azusa Hayashi, Katrina J. Woolcock, Martin A. Gorovsky, Kazufumi Mochizuki
Molecular Cell, 59, 229-242 (2015)
要 約
多くの真核生物は小分子RNAを共通してトランスポゾンの抑制に用いている.しかし,小分子RNAをどのように宿主のゲノムとトランスポゾンとの識別に用いるかについては多様性がある.繊毛虫類に属するテトラヒメナは小分子RNAを用いて小核と大核のゲノムを比較することによりトランスポゾンに由来する配列を認識し,DNAの削減によりそのDNA自体を大核から取り除くというユニークなトランスポゾンの抑制の戦略をとる.この論文では,この小核と大核の比較にくわえ,一部のかぎられたIESから形成された小分子RNAがほかのIESをトランスに認識し,その認識が新奇の小分子RNAの合成の連鎖反応をひき起こすことによりDNAの削減の堅牢性が確保されていることを示した.
はじめに
トランスポゾンは宿主のゲノムの安定性をみだす脅威であり,多くの真核生物は共通してRNAiやそれに類する機構により形成される20~30塩基の小分子RNAをトランスポゾンの抑制に用いている.しかし,トランスポゾンのみに小分子RNAをはたらかせる方法については生物種のあいだで多様性がみられる.なかでも,テトラヒメナがトランスポゾンを識別する戦略はひときわユニークである.
繊毛虫類に属する単細胞真核生物であるテトラヒメナは同一の細胞に小核および大核とよばれる異なる2つの核をもつ1).小核は転写活性を欠く生殖核で,大核は転写活性をもつ栄養核である.有性生殖の過程において大核は小核から分化し,その際にDNAの削減によりゲノムの約1/3(約50 Mb)が除去される.この除去される配列は0.5~40 kbの約1万箇所の領域に分かれて小核のゲノムに散在しており,IES(internal eliminated sequence)とよばれる.既知のトランスポゾンはすべてIESに存在することから,DNAの削減は転写活性をもつ大核からトランスポゾンをDNA配列ごと除去する過程と考えられる.
以前に,筆者らは,少なくとも一部のIESは小分子RNAを用いた大核と小核のゲノムの差し引きにより小核にのみ存在する配列として認識されていることを報告した2,3).それにもとづいたモデル(図1)では,まず,ふだんは転写活性をもたない小核が有性生殖の初期にのみ転写され,そこから小核のゲノム全体を写し取った約29塩基の小分子RNA(scnRNA)が形成される(ステップ1:scnRNAの合成).つぎに,scnRNAは大核へと移動し,大核のゲノムに相補的なscnRNAは分解される(ステップ2:scnRNAの選別).分解からのがれたscnRNAは新たに形成された大核へと移動し,それに相補的な配列をIESとして認識する(ステップ3:IESの認識).IESはヘテロクロマチン化され最終的に除去される(ステップ4:DNAの削減).このような2つのゲノムの差し引きのみによりすべてのIESを特定するには,小核のゲノムが一様に転写されscnRNAに写し取られる必要がある.しかし,実際にはscnRNAは小核のゲノムの一部からしか形成されず4),scnRNAを形成しないIESの削減がどのように制御されているのかは不明であった.
1.後期scnRNAは有性生殖の後期に発現する新奇の小分子RNAである
実験条件のもとでは,テトラヒメナの有性生殖の開始から新しい大核の形成まで約7 hpm(hpm:hours post mixing),新しい大核におけるDNAの削減の開始まで約14 hpmを要する.また,筆者らによる以前の研究から,scnRNAの合成は3 hpm前後に起こり,scnRNAの選別は新しい大核の形成までに完了することがわかっている3,4).今回,新しい大核の形成ののち,scnRNAとほぼ同じ約29塩基の小分子RNAが合成されていることが見い出された.この有性生殖の後期に発現する新奇の小分子RNAを後期scnRNA,以前から知られていた有性生殖の前期から発現するscnRNAを初期scnRNAとよぶことにした.以前に,初期scnRNAは12個あるArgonauteのうちTwi1pと結合して作用することが報告されている3).一方で,後期scnRNAはTwi1pにくわえ,有性生殖の後期において特異的に発現するTwi11pとも結合していた.初期scnRNAが結合しているTwi1pは有性生殖の前期において古い(有性生殖ののちに分解される)大核から発現していたのに対し,後期scnRNAの結合しているTwi1pおよびTwi11pはともに新しい大核から発現していた.
2.scnRNAの発現パターンから3種類のIESが定義される
初期scnRNAが一部のIESから形成されIESへの特異性がscnRNAの選別により獲得されるのに対し,後期scnRNAはほぼすべてのIESから形成され合成の段階からIESに対する高い特異性をもつことがわかった.つまり,IESには,初期scnRNAおよび後期scnRNAを発現するものと,後期scnRNAのみを発現するものが存在した.初期scnRNAおよび後期scnRNAを発現するIESをA型IES,後期scnRNAのみを発現するIESをB型IESとよぶことにした.また,ごく少数ではあるが初期scnRNAあるいは後期scnRNAのどちらの発現も認められないIESが存在し,これらをC型IESとよぶことにした.調べた8105個のIESのうち,A型IESが4695個,B型IESが3293個,C型IESが117個あった.
テトラヒメナにおいてはTlr-1とよばれるDNAトランスポゾンとREPファミリーとよばれるレトロトランスポゾンの存在が報告されている.これらのトランスポゾンは転移や逆転写などにかかわる遺伝子が保存されていることから,現在でも転移活性をもつと考えられている.これらのトランスポゾンと相補的な配列はほぼすべてA型IESのみに見い出された.また,小核のゲノム全体のGC含量が22%なのに対し,A型IES,B型IES,C型IESのGC含量は,それぞれ,31%,28%,26%であった.これらの結果,および,後述するB型IESに存在するAリピートの存在は,A型IESは活性をもつトランスポゾンを含む進化的に新しいIESであり,B型IESおよびC型IESはトランスポゾン配列が退化しより“テトラヒメナ化”した進化的に古いIESであることを示唆した.
3.後期scnRNAは一部のIESの削減において重要である
後期scnRNAのDNAの削減における役割について調べるため,小核のTwi1pをコードする遺伝子およびTwi11pをコードする遺伝子を破壊し,この遺伝子破壊株において有性生殖が終了したのちのDNAの削減について解析した.この遺伝子破壊株では古い大核からはTwi1pが発現するため初期scnRNAは正常に機能したが,小核から形成される新しい大核からはTwi1pおよびTwi11pは発現せず後期scnRNAは不安定かつ機能しなかった.この遺伝子破壊株の子においては多くのB型IESの削減が抑制され,また,A型IESおよびC型IESの削減も部分的に影響をうけていた.よって,後期scnRNAはDNAの削減に必須ではないが,一部のIES,とくにB型IESはDNAの削減において重要な役割をはたすことがわかった.
一方で,古い大核のTwi1pをコードする遺伝子を破壊した場合,初期scnRNAおよび後期scnRNAが失われ,この条件ではIESのタイプにかかわらずすべてのDNAの削減が完全に抑制された.よって,初期scnRNAはすべてのDNAの削減に必須であり,また,後期scnRNAの発現は初期scnRNAに依存することが示された.
4.B型IESはAリピートを介しA型IESに由来する初期scnRNAにより認識される
ほぼすべてのB型IESにはA型IESに相補的な少なくとも約100塩基の配列が存在していた.このB型IESに存在する配列をAリピートとよぶことにした.A型IESからは初期scnRNAが形成されるため,Aリピートを介しB型IESも初期scnRNAによりトランスに認識されることが予想された.実際に,GFPをコードする遺伝子はIESとは認識されずDNAの削減をうけないが,その真ん中にB型IESのもつAリピートを挿入することによりDNAの削減がひき起こされた.よって,AリピートにはDNAの削減を誘導するシグナル(おそらくは,初期scnRNA)をうけとる能力があると考えられた.
5.後期scnRNAの合成は初期scnRNAにより誘導されヘテロクロマチン化に依存してシスに伝達される
時系列にそって詳細に解析したところ,後期scnRNAの発現はAリピートの付近からはじまり,それがシスに広がって,最終的にB型IESの全体をカバーしていくようすが観察された.任意のA型IESをテトラヒメナの形質転換ベクターにクローニングし,さらに,その中央にIESではない(DNAの削減をうけない)0.5~1 kbほどのテトラヒメナのゲノム配列(標的配列)を挿入した.つぎに,この形質転換ベクターを有性生殖している新しい大核に導入した.すると,標的配列に相補的なゲノム配列がDNAの削減をうけるのが観察され,この現象を共削減(co-deletion)とよぶことにした.共削減は形質転換ベクターに導入されたA型IESや標的配列の種類にかかわらず高い頻度で起こった.また,Twi11pの発現を欠損した変異体では抑制されたことから,後期scnRNAに依存的な現象であると考えられた.共削減は,初期scnRNAが形質転換ベクターに導入されたA型IESを認識し,それが後期scnRNAの発現をシスに誘導することにより標的配列から後期scnRNAが形成され,それが標的配列と相補的な内在性の配列をトランスに認識しDNAの削減を誘導した結果であると考えられた.
IESは初期scnRNAに依存してヘテロクロマチン化され,ヘテロクロマチンが最終的にDNAの削減を誘導する5,6).また,多くの真核生物において,ヘテロクロマチン化はシスに拡散する性質がある.これらのことから,後期scnRNAの発現はIESをシスに拡散するヘテロクロマチン化に依存している可能性が考えられた.そこで,IESのヘテロクロマチン化に必須なヒストンH3のLys9のメチル化酵素であるEzl1p,あるいは,ヒストンH3のLys9のメチル化に特異的に結合するHP1様タンパク質であるPdd1pを欠損した変異体においてscnRNAの発現を観察したところ,予想どおり,いずれの変異体においても初期scnRNAの発現は正常であったが後期scnRNAの発現は消失していた.よって,後期scnRNAの合成にはヘテロクロマチン化が必須であると結論した.
6.scnRNAによるトランス認識ネットワークがDNAの削減の堅牢性を保障する
scnRNAによるIESのトランス認識および後期scnRNAの発現のシスの拡散は後期scnRNAの合成の連鎖反応をひき起こすと考えられた.したがって,ゲノムに散在するIESがネットワークを形成し,一部のIESからの初期scnRNAの発現がみだされたとしても,ほかのIESから発現した初期scnRNAによるトランス認識によりDNAの削減は保障されていると予想された.このようなscnRNAによるトランス認識ネットワークのはたらきについて調べるため,初期scnRNAの発現のみだれ,および,それによるIESの認識についてシミュレーションした.まず,ランダムに選んだ一部のA型IESのみが初期scnRNAを発現する状況を想定し,このかぎられた初期scnRNAが残りのどれだけのIESを認識できるか計算した.この結果,1%のA型IESしか初期scnRNAを形成しなかったとしても,これらはすべてのタイプのIESを含む33%のIESをトランスに認識することができ,この33%のIESが後期scnRNAを発現しそれがさらにほかのIESをトランスに認識したとすれば,あわせて84%のIESがscnRNAにより認識されることが示唆された.一方,すべてのIESが一様にscnRNAを発現したとしても,それらは1.3%の遺伝子をコードする領域しか認識しなかった.よって,DNAの削減はscnRNAによるIESのトランス認識ネットワークによりその堅牢性が保障されており,そのネットワークはIES以外の領域には伝達されないよう進化的にデザインされていると考えられた.
おわりに
この研究,および,これまでの報告を総括すると以下のように考えられる.初期scnRNAはA型IESとその周辺のかぎられた小核のゲノムから発現し,古い大核においてscnRNAの選別をうけたのち,新しい大核へと移動する.そこで,初期scnRNAはA型IESを認識するとともに,Aリピートを介しB型IESを認識する.そののち,A型IESおよびB型IESから後期scnRNAが発現し,最終的にDNAの削減を誘導する(図2).
哺乳類やショウジョウバエなどの後生動物においてはPIWIに結合する小分子RNAであるpiRNAがトランスポゾンの抑制において重要なはたらきをもつ7,8).これらの生物にはpiRNAクラスターとよばれる,トランスポゾンや退化したトランスポゾンが蓄積したゲノム領域が存在し,そこから形成されたpiRNAがゲノムに散在するトランスポゾンを認識し抑制する.また,piRNAがトランスポゾンに由来するRNAに出会うと,ping-pongサイクルとよばれる生合成経路によりトランスポゾンに由来するRNAに相補的なpiRNAが増幅される8,9).今回の研究におけるテトラヒメナのscnRNAによるトランス認識ネットワークは,このpiRNA機構を想起させた.すなわち,A型IESはpiRNAクラスターに,また,後期scnRNAの合成の連鎖反応はping-pongサイクルに相当する役割をもつと考えることができる.後生動物とテトラヒメナという進化的に遠く離れた生物が,piRNAとscnRNAという合成経路の異なる小分子RNAを使い,結果的に戦略的に類似したトランスポゾンの認識の機構を獲得していることは,トランスポゾンという生物に共通する脅威に対する機構の進化を考えるうえで興味深い.
文 献
- Chalker, D. L., Meyer, E. & Mochizuki, K.: Epigenetics of ciliates. Cold Spring Harb. Perspect. Biol., 5, a017764 (2013)[PubMed]
- Mochizuki, K., Fine, N. A., Fujisawa, T. et al.: Analysis of a piwi-related gene implicates small RNAs in genome rearrangement in Tetrahymena. Cell, 110, 689-699 (2002)[PubMed]
- Mochizuki, K. & Gorovsky, M. A.: Conjugation-specific small RNAs in Tetrahymena have predicted properties of scan (scn) RNAs involved in genome rearrangement. Genes Dev., 18, 2068-2073 (2004)[PubMed]
- Schoeberl, U. E., Kurth, H. M., Noto, T. et al.: Biased transcription and selective degradation of small RNAs shape the pattern of DNA elimination in Tetrahymena. Genes Dev., 26, 1729-1742 (2012)[PubMed]
- Taverna, S. D., Coyne, R. S. & Allis, C. D.: Methylation of histone H3 at lysine 9 targets programmed DNA elimination in Tetrahymena. Cell, 110, 701-711 (2002)[PubMed]
- Liu, Y., Taverna, S. D., Muratore, T. L. et al.: RNAi-dependent H3K27 methylation is required for heterochromatin formation and DNA elimination in Tetrahymena. Genes Dev., 21, 1530-1545 (2007)[PubMed]
- Aravin, A. A., Sachidanandam, R., Girard, A. et al.: Developmentally regulated piRNA clusters implicate MILI in transposon control. Science, 316, 744-747 (2007)[PubMed]
- Brennecke, J., Aravin, A. A., Stark, A. et al.: Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell, 128, 1089-1103 (2007)[PubMed]
- Gunawardane, L. S., Saito, K., Nishida, K. M. et al.: A slicer-mediated mechanism for repeat-associated siRNA 5' end formation in Drosophila. Science, 315, 1587-1590 (2007)[PubMed]
著者プロフィール
略歴:2003年 金沢大学大学院自然科学研究科 修了,2004年 米国Rochester大学 博士研究員を経て,2006年よりオーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciencesリサーチアソシエイト.
研究テーマ:テトラヒメナでのDNAの削減における小分子RNAの役割.
片岡 研介(Kensuke Kataoka)
略歴:2007年 兵庫県立大学大学院生命理学研究科 修了,同年よりオーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciences博士研究員.
研究テーマ:テトラヒメナでのDNAの削減におけるヘテロクロマチンの役割.
望月 一史(Kazufumi Mochizuki)
オーストリアInstitute of Molecular Biotechnology of the Austrian Academy of Sciencesグループリーダー.
研究室URL:http://www.imba.oeaw.ac.at/research/kazufumi-mochizuki/
© 2015 能登朋子・片岡研介・望月一史 Licensed under CC 表示 2.1 日本