タンパク質へのセレンの取り込み:その厳密性の分子基盤
千葉志穂1・伊藤弓弦1・関根俊一1・横山茂之2
(1東京大学大学院理学系研究科 生物化学専攻,2理化学研究所生命分子システム基盤研究領域)
email:横山茂之
DOI: 10.7875/first.author.2010.021
Structural basis for the major role of O-phosphoseryl-tRNA kinase in the UGA-specific encoding of selenocysteine.
Shiho Chiba, Yuzuru Itoh, Shun-ichi Sekine, Shigeyuki Yokoyama
Molecular Cell, 39, 410-420 (2010)
セレンは,われわれヒトを含め多くの生物の生存に必須な微量元素である.生体内では“21番目のアミノ酸”として知られるセレノシステインを構成する元素としてタンパク質に取り込まれ,抗酸化作用をもつ酵素の活性中心などに用いられている.セレノシステインは,古細菌および真核生物の場合,セレノシステイン専用のtRNA(tRNASec)に対して3種類の酵素が順に作用することで合成される.第1段階の反応を触媒するセリルtRNA合成酵素は,tRNASecとセリンに対応するtRNA(tRNASer)の両方を基質とする.したがって,第2段階を担うO-ホスホセリルtRNAキナーゼがtRNASecとtRNASerとを厳密に区別して,tRNASecのみを選択的にセレノシステイン生合成経路に導いているものと考えられるが,その詳細な分子機構は不明であった.今回,筆者らは,古細菌tRNASecとO-ホスホセリルtRNAキナーゼとの複合体の結晶構造を決定した.これにより,O-ホスホセリルtRNAキナーゼがC末端ドメインを介してtRNASecに特有のDアームの立体構造を認識することでtRNASerと見分け,tRNASecを介したセレンの取り込みの厳密性を保証していることが明らかになった.
セレンはヒトから細菌にいたるまで幅広い生物の生存に必須な微量元素である.ヒトにおけるセレン欠乏は,心筋症や高血圧,関節炎の症状と関連するほか,ある種のウイルス感染時には病原性増悪因子となりうる.一方,セレンの過剰摂取は神経系への障害やさまざまな内臓疾患をひき起こし有毒である1).つまり,生物には,本来は有毒なセレンを,毒性を発揮しないようなかたちで取り込み有効に利用するための制御されたしくみが備わっている.
セレノシステイン(Sec)は,その名のとおり,システインの硫黄原子(S)がセレン原子(Se)に置き換わった構造をしたアミノ酸である.セレノシステイン生合成にかかわる酵素は,真正細菌,古細菌,真核生物のいずれにも存在していることから,生体内でのセレノシステインの重要性がうかがえる.セレノシステイン残基はタンパク質のなかで主として酸化還元酵素の活性部位に存在する.セレノシステインの側鎖のセレノール基(-SeH)はシステインのチオール基(-SH)より求核反応性に富んでおり,化学反応を触媒するのに役立つ.たとえば,ヒトにおいてこれまでに同定されている25種類のセレン含有タンパク質には,細胞内外の酸化・抗酸化作用,甲状腺ホルモン代謝,グルコース代謝,精子の成熟にかかわるものが含まれる.また,10残基もセレノシステイン残基をもち,セレンの全身への輸送に役立っていると考えられるタンパク質もある.このように,生物は反応性が高く高濃度では有毒なセレンを,セレノシステインというアミノ酸としてタンパク質に取り込み,安全に活用している.
セレノシステインは,タンパク質合成に用いられる20種類の標準アミノ酸と同様に,リボソームにおける翻訳の過程をへてタンパク質に取り込まれる.そのため“21番目のアミノ酸”と称される.標準アミノ酸と同様に,セレノシステインにも専用のtRNA(tRNASec)が存在し,これがリボソームにセレノシステインを運び込む.しかし,おなじみのコドン表にセレノシステインに対応したコドンなどあっただろうか? じつは,tRNASecは“UGA”コドンに相補的なアンチコドンをもっている.UGAは通常は終止コドンとして機能する.ところが,mRNAにセレノシステイン挿入配列(Sec-insertion sequence:SECIS)とよばれる配列が存在すると,tRNASecとセレノシステインに特異的な翻訳伸長因子EF-Secとのはたらきにより,UGAコドンは終止コドンではなくSecを指定するコドンと解釈されタンパク質にSecが取り込まれるのである.
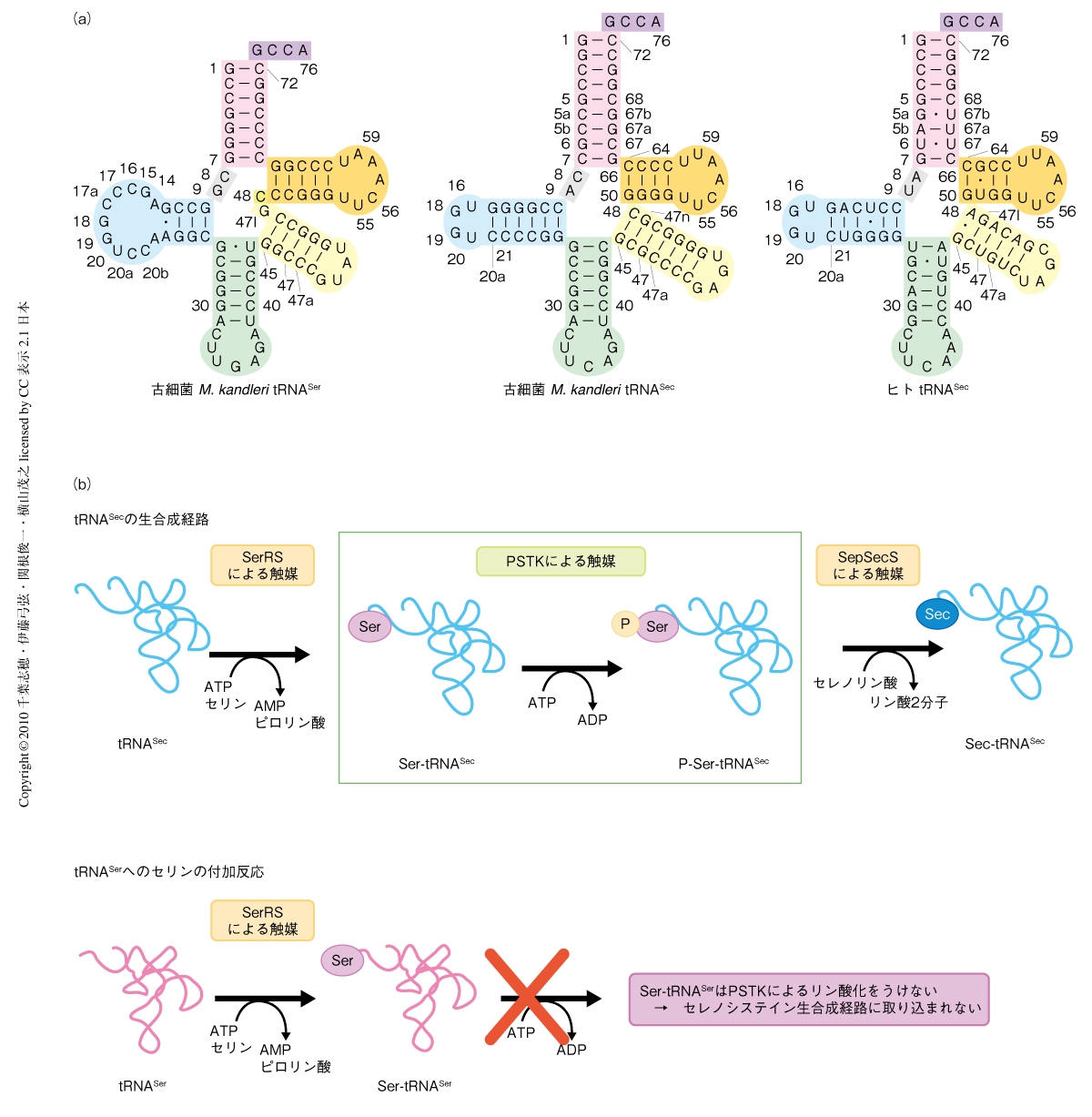
最近,筆者らは,ヒトのtRNASecの結晶構造を明らかにした2)(PDB ID:3A3A).これにより,tRNASecはアンチコドン以外に典型的なtRNAとは異なる特徴をもつことがわかった(図1a).まず,tRNASecは長いエクストラアームをもつ(これは,セリンやロイシンのtRNAと共通する).また,標準的なtRNAではアクセプターステムとTステムとをあわせた長さが12塩基対であるのに対し,tRNASecでは13塩基対である.そして,標準的なtRNAのDアームは3~4塩基対のステムと7~11塩基のループで構成されるのに対し,ヒトのtRNASecは6塩基対の長いステムと4塩基のループからなることもわかった.tRNASecのDアームは,この長いDステムが原因で通常のtRNAよりも立体構造上,大きく外側に張り出している.さらに,tRNASecに固有の3次元的な塩基対(U20:G19:C56)も見い出された.これらのtRNASecに特有の構造的な特徴の一部または全部が,セレノシステインの生合成を担う酵素がtRNASecを認識し,ほかの標準的なtRNAと識別するための目印になっているものと予想された.しかし,実際にどの特徴が決定的な要素であり,それがどのように認識されているかという構造的な基盤はこれまで明らかにされていなかった.
さて,ここで古細菌および真核生物におけるセレノシステイン生合成経路を紹介する(図1b).セレノシステインはtRNASecに結合した状態で複数の酵素の作用をへて合成される.まず,tRNASecはセリルtRNA合成酵素(Seryl-tRNA synthetase:SerRS)によってセリンを付加される(Ser-tRNASec).つぎに,この付加されたセリンの側鎖の水酸基(-OH)がO-ホスホセリルtRNAキナーゼ(O-phosphoseryl-tRNA kinase:PSTK)によってリン酸化される.このリン酸化セリンのリン酸基がO-ホスホセリルtRNA:セレノシステイニルtRNA合成酵素(Sep-tRNA:Sec-tRNA synthase:SepSecS)によりセレノール基(-SeH)に変換され,最終的にセレノシステインが結合したtRNASec(Sec-tRNASec)が完成する.
このセレノシステイン生合成経路は大きな危険ととなりあわせのシステムである.1段階目の反応を触媒するセリルtRNA合成酵素はセリンに対応するtRNA(tRNASer)にセリンを付加する機能を兼ねている.細胞内にはセリンのほうがセレノシステインよりも圧倒的に多く存在することから,tRNASerに対するセリンの付加こそがセリルtRNA合成酵素の主たる仕事である.ここで仮に,セリンを付加されたtRNASer(Ser-tRNASer)がtRNASecと誤認され,のちのセレノシステイン生合成経路に入ってしまったとする.そうすると,セリンに対応するコドンをもちながらセレノシステインと結合したtRNASer(Sec-tRNASer)ができてしまう.これでは,結果的にセリンが挿入されるはずの位置にセレノシステインが取り込まれたタンパク質が合成されてしまい,重篤な問題が生じる.すなわち,セリルtRNA合成酵素による反応産物のうちSer-tRNASecだけが選択的にセレノシステイン生合成経路に取り込まれるための,厳密な見分けの機構が不可欠なのである.
では,その厳密な見分けを保証するのはいったいどの段階の反応であろうか? じつは,O-ホスホセリルtRNAキナーゼはSer-tRNASecのみをリン酸化し,Ser-tRNASerを基質としないことが生化学的に示されている3-5)(図1b).つまり,なんらかの機構で2種類のtRNAを厳密に見分けていると考えられる.近年,報告されたO-ホスホセリルtRNAキナーゼとATP類似体との結晶構造(PDB ID:3A4L )からは,O-ホスホセリルtRNAキナーゼがtRNASecのアクセプターステムの長さを認識することが推測された6).しかし,アクセプターステムの長さを変えたtRNASec変異体でもO-ホスホセリルtRNAキナーゼによってリン酸化されることが知られており5),O-ホスホセリルtRNAキナーゼがtRNASecとtRNASerとを見分ける機構は依然として謎のままであった.
今回,筆者らが解析したtRNASecとO-ホスホセリルtRNAキナーゼの複合体の結晶構造により,tRNASecとO-ホスホセリルtRNAキナーゼとの特異的な相互作用のしくみが明らかになり,O-ホスホセリルtRNAキナーゼによるtRNAの厳密な選択性の謎が解けた.
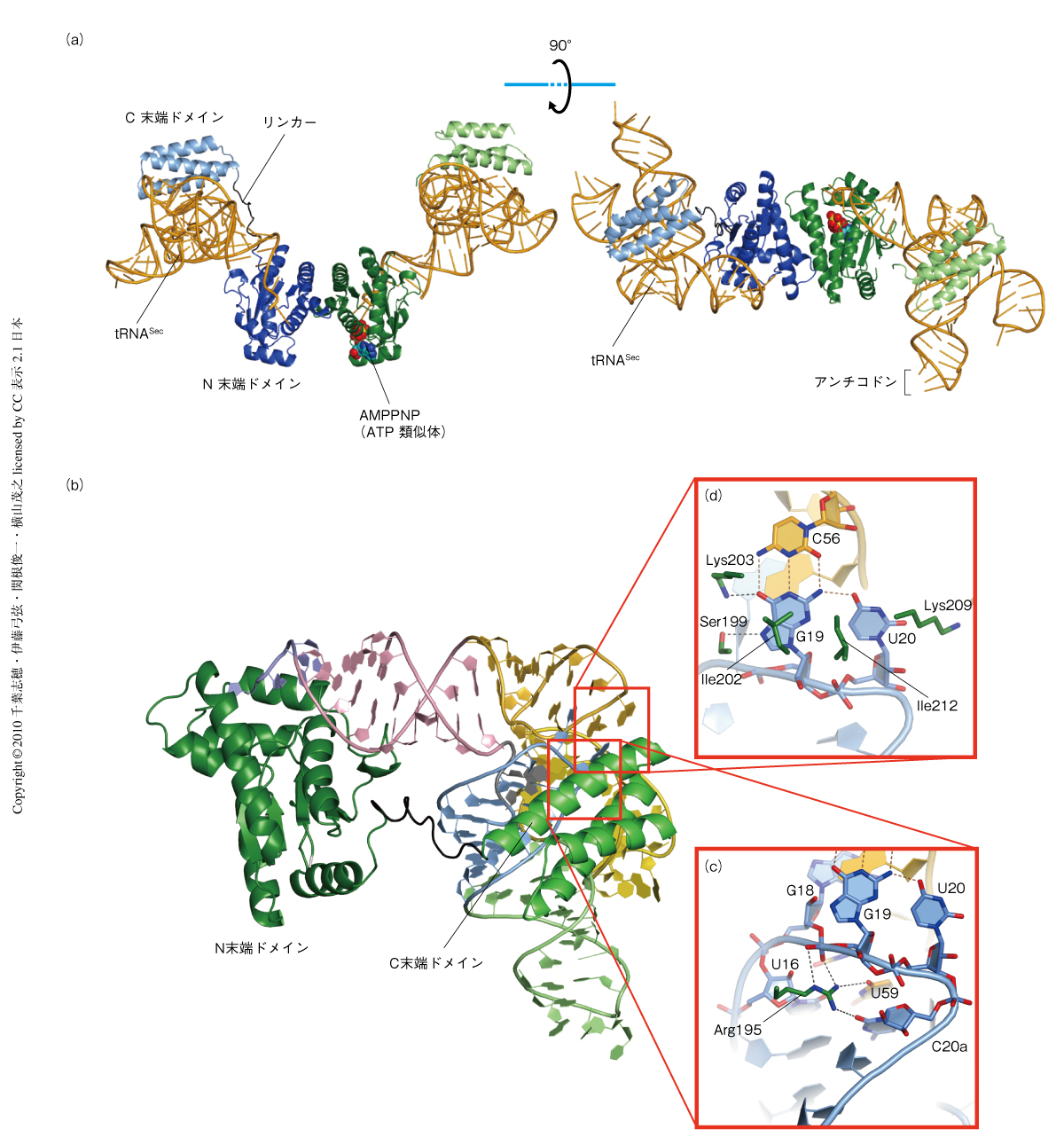
筆者らは,古細菌のtRNASecとO-ホスホセリルtRNAキナーゼの複合体の立体構造をX線結晶構造解析により2.4~2.9Åの分解能で決定した(PDB ID:3ADB ,3ADC ,3ADD ).tRNASecはMethanopyrus kandleri由来のものを,O-ホスホセリルtRNAキナーゼはMethanocaldococcus jannaschii由来のものを用いた.M. jannaschiiのO-ホスホセリルtRNAキナーゼはセリンを結合したM. kandleriのtRNASecを効率よくリン酸化するが,tRNASerには作用しない.O-ホスホセリルtRNAキナーゼはホモ2量体を形成しており,単量体1つにつき1分子のtRNASecと結合していた(図2a).古細菌M. kandleriのtRNASecにはヒトtRNASecにおいてみられた前述の特徴がすべて共通していた2).これらの特徴は,古細菌および真核生物においてtRNASecに特有の特徴として広く保存されているものと考えられる.一方,248アミノ酸残基からなるO-ホスホセリルtRNAキナーゼ単量体は,リン酸化活性部位をもつN末端ドメイン(1~172残基)とC末端ドメイン(184~248残基),および,これらをつなぐリンカー(173~183残基)によって構成されていることがわかった.以前に報告されたM. jannaschiiのO-ホスホセリルtRNAキナーゼ単独での結晶構造(PDB ID:3A4L )では,N末端ドメインとC末端ドメインとが近接しひとつの大きなドメインを形成しているようにみえた6).しかし,今回の複合体の結晶構造では,O-ホスホセリルtRNAキナーゼのN末端ドメインとC末端ドメインとは互いに分離しており,それぞれ,tRNASecのアクセプターアーム,Dアームと結合することが明らかになった.また,N末端ドメイン,C末端ドメインのいずれも,tRNASecに特有のアンチコドンとは相互作用しておらず,O-ホスホセリルtRNAキナーゼにとってアンチコドンはtRNA識別の目印としてははたらいていないことがわかった.
tRNASecのDアームは6塩基対以上のDステムと4塩基のDループからなり,この特徴は古細菌と真核生物とで保存されている.今回明らかにした複合体の構造で,この長いDステムは通常のtRNAの3~4塩基対のステムの場合よりも大きく外側に張り出していた.また,Dループの残基G19とU20はTループ上のC56と塩基の3つ組(U20:G19:C56)を形成し,同じくDループのU16はtRNASecの内部でTループのU59と塩基対をつくっていた.これらは,いずれもヒトtRNASecにも共通してみられた特徴である2).一方,O-ホスホセリルtRNAキナーゼのC末端ドメインは3本のαヘリックスで構成されており,リンカー部分もあわせてtRNASecのDアームにそうように位置していた.
興味深いことに,O-ホスホセリルtRNAキナーゼのC末端ドメインはtRNASecのDアームの特徴的な構造と重点的に相互作用をしていた(図2b).まず,C末端ドメインおよびリンカーの複数のアミノ酸残基が,Dステムのリン酸および糖の骨格と水素結合ないし静電的な相互作用を形成していた.外側に突き出たtRNASecのDステムの構造と立体的および静電的によく適合しているといえる.さらに,tRNASecの塩基を特異的に認識する相互作用もみられた.tRNASec特有の短いDループはtRNASecの表面上に小さい穴を形成している.この内部にO-ホスホセリルtRNAキナーゼのC末端ドメインのアミノ酸残基Arg195の側鎖がぴったり収まっており,Dループの塩基および骨格のみならず,上記のU59とも水素結合していた(図2c).また,塩基の3つ組(U20:G19:C56)を構成するG19,U20は,Ile202およびIle212,Ser199,Lys203,Lys209と相互作用していた(図2d).このように,C末端ドメインはtRNASec特有の構造である長いDステム,小さいDループ,U20:G19:C56の3つ組,U16:U59ペアに完璧に適合し認識していることがわかった.
つづいて,O-ホスホセリルtRNAキナーゼのリン酸化活性におけるC末端ドメインの重要性を知るため,N末端ドメイン断片(C末端ドメインのないO-ホスホセリルtRNAキナーゼ断片)を調製し全長のO-ホスホセリルtRNAキナーゼと活性を比較した.N末端ドメイン断片では,全長O-ホスホセリルtRNAキナーゼよりリン酸化活性が劇的に低下したことから,C末端ドメインはO-ホスホセリルtRNAキナーゼの活性に決定的な寄与をしていることがわかった.また,O-ホスホセリルtRNAキナーゼとtRNASecとの結合に対するN末端ドメインおよびC末端ドメインの寄与を知るため,全長のO-ホスホセリルtRNAキナーゼ,N末端ドメイン断片,C末端ドメイン断片(N末端ドメインのないO-ホスホセリルtRNAキナーゼ断片)と,tRNASecとのゲルシフト実験を行ったところ,O-ホスホセリルtRNAキナーゼとtRNASecとの強い結合には,C末端ドメインによる結合が必須であることが示された.さらに,全長O-ホスホセリルtRNAキナーゼおよびC末端ドメイン断片とのtRNASecの解離定数を求めると,それぞれ,48 nM,39 nMであり,C末端ドメインがtRNASecへの結合を実質的に担っていることがわかった.O-ホスホセリルtRNAキナーゼのtRNASerに対する解離定数が1.3μMであるという報告をふまえると7),O-ホスホセリルtRNAキナーゼのC末端ドメインとtRNASecのDアームとの相補的で強い結合が,tRNASecのみを認識しtRNASerには作用しないというO-ホスホセリルtRNAキナーゼの高い特異性の基盤となっていると結論づけられた.
以上のように,立体構造および生化学的な解析の両面から,リン酸化活性部位のない小さなドメインであるC末端ドメインこそがtRNASecとtRNASerとの見分けに決定的であることが示された(図3).この識別はtRNAのDアームの形状にもとづいており選択性が高い.これにより,20種類の標準アミノ酸が用いられる通常の翻訳に影響を及ぼすことなく,セレン含有タンパク質の特定部位のみに正確にセレノシステインを導入することが可能になっているのだろう.
筆者らが明らかにしたtRNASec-O-ホスホセリルtRNAキナーゼ複合体の結晶構造から,柔軟性の高いリンカーでつながっているN末端ドメインとC末端ドメインとは互いに独立してtRNASecと結合・解離できることが示された.また,ゲルシフト実験により,O-ホスホセリルtRNAキナーゼのC末端ドメインとtRNASecのDアームは,N末端ドメインとアクセプターアームよりもはるかに高い親和性をもつことが示された.これらのことから,O-ホスホセリルtRNAキナーゼによるリン酸化反応ののち,C末端ドメインはDアームとの結合を保持したままN末端ドメインのみがアクセプターアームから離れうると考えられた.そこにセレノシステイン合成の次段階の反応を触媒する酵素であるO-ホスホセリルtRNA:セレノシステイニルtRNA合成酵素が近づいてN末端ドメインとすばやく入れ替われば,加水分解されやすいtRNASecとアミノ酸との結合を保護しつつtRNASecを受け渡すことができる.O-ホスホセリルtRNAキナーゼのリン酸化活性部位がセリン以外のアミノ酸も許容するという生化学的なデータは,この推論と矛盾しない4).実際にそうであれば,C末端ドメインとtRNASecとの相互作用は,tRNAの見分けのみならず,セレノシステインの生合成経路の一貫した品質保証にも役立っているといえるだろう.
今回,O-ホスホセリルtRNAキナーゼがtRNASec特有の構造を認識していること,さらに,構造の認識に特化したドメインであるC末端ドメインをもっていることがわかり,古細菌および真核生物におけるSer-tRNASecを選択的にセレノシステイン合成経路に導く分子機構が解明された.C末端ドメインは,あえてアンチコドンではなく,tRNASecのDアームに保存された複数の特徴的な構造と強力かつ特異的に相互作用する.この相互作用が,ここで論じたようなtRNAの見分けの機構以外においてもなんらかの意味をもっていたとしても不思議ではない.さらに,細胞内では稀少な存在であるはずのセレノシステインの生合成にかかわるタンパク質が基質の受け渡しの際にどのように高い効率を確保しているかなど,いまだ明らかでないことは多い.今後,構造解析を含むさまざまなアプローチによりセレノシステイン生合成への理解がさらに深まることを期待する.“21番目のアミノ酸”であるセレノシステインの生合成系は,生物が進化の過程で必要に応じて遺伝暗号を拡張した結果,生まれたものと考えられる.したがって,セレノシステイン生合成のしくみの理解は,多くの有用な機能をもった天然または非天然のアミノ酸をタンパク質の目的の位置に導入する系の開発への重要な基礎ともなるであろう.
東京大学大学院医学系研究科修士課程 在学中.
伊藤弓弦(Yuzuru Itoh)
東京大学分子細胞生物学研究所 助教.
関根俊一(Shun-ichi Sekine)
東京大学大学院理学系研究科 特任准教授.
横山茂之(Shigeyuki Yokoyama)
理化学研究所生命分子システム基盤研究領域 領域長,東京大学大学院理学系研究科 兼任教授.
© 2010 千葉志穂・伊藤弓弦・関根俊一・横山茂之 Licensed under CC 表示 2.1 日本
(1東京大学大学院理学系研究科 生物化学専攻,2理化学研究所生命分子システム基盤研究領域)
email:横山茂之
DOI: 10.7875/first.author.2010.021
Structural basis for the major role of O-phosphoseryl-tRNA kinase in the UGA-specific encoding of selenocysteine.
Shiho Chiba, Yuzuru Itoh, Shun-ichi Sekine, Shigeyuki Yokoyama
Molecular Cell, 39, 410-420 (2010)
要 約
セレンは,われわれヒトを含め多くの生物の生存に必須な微量元素である.生体内では“21番目のアミノ酸”として知られるセレノシステインを構成する元素としてタンパク質に取り込まれ,抗酸化作用をもつ酵素の活性中心などに用いられている.セレノシステインは,古細菌および真核生物の場合,セレノシステイン専用のtRNA(tRNASec)に対して3種類の酵素が順に作用することで合成される.第1段階の反応を触媒するセリルtRNA合成酵素は,tRNASecとセリンに対応するtRNA(tRNASer)の両方を基質とする.したがって,第2段階を担うO-ホスホセリルtRNAキナーゼがtRNASecとtRNASerとを厳密に区別して,tRNASecのみを選択的にセレノシステイン生合成経路に導いているものと考えられるが,その詳細な分子機構は不明であった.今回,筆者らは,古細菌tRNASecとO-ホスホセリルtRNAキナーゼとの複合体の結晶構造を決定した.これにより,O-ホスホセリルtRNAキナーゼがC末端ドメインを介してtRNASecに特有のDアームの立体構造を認識することでtRNASerと見分け,tRNASecを介したセレンの取り込みの厳密性を保証していることが明らかになった.
はじめに
セレンはヒトから細菌にいたるまで幅広い生物の生存に必須な微量元素である.ヒトにおけるセレン欠乏は,心筋症や高血圧,関節炎の症状と関連するほか,ある種のウイルス感染時には病原性増悪因子となりうる.一方,セレンの過剰摂取は神経系への障害やさまざまな内臓疾患をひき起こし有毒である1).つまり,生物には,本来は有毒なセレンを,毒性を発揮しないようなかたちで取り込み有効に利用するための制御されたしくみが備わっている.
セレノシステイン(Sec)は,その名のとおり,システインの硫黄原子(S)がセレン原子(Se)に置き換わった構造をしたアミノ酸である.セレノシステイン生合成にかかわる酵素は,真正細菌,古細菌,真核生物のいずれにも存在していることから,生体内でのセレノシステインの重要性がうかがえる.セレノシステイン残基はタンパク質のなかで主として酸化還元酵素の活性部位に存在する.セレノシステインの側鎖のセレノール基(-SeH)はシステインのチオール基(-SH)より求核反応性に富んでおり,化学反応を触媒するのに役立つ.たとえば,ヒトにおいてこれまでに同定されている25種類のセレン含有タンパク質には,細胞内外の酸化・抗酸化作用,甲状腺ホルモン代謝,グルコース代謝,精子の成熟にかかわるものが含まれる.また,10残基もセレノシステイン残基をもち,セレンの全身への輸送に役立っていると考えられるタンパク質もある.このように,生物は反応性が高く高濃度では有毒なセレンを,セレノシステインというアミノ酸としてタンパク質に取り込み,安全に活用している.
セレノシステインは,タンパク質合成に用いられる20種類の標準アミノ酸と同様に,リボソームにおける翻訳の過程をへてタンパク質に取り込まれる.そのため“21番目のアミノ酸”と称される.標準アミノ酸と同様に,セレノシステインにも専用のtRNA(tRNASec)が存在し,これがリボソームにセレノシステインを運び込む.しかし,おなじみのコドン表にセレノシステインに対応したコドンなどあっただろうか? じつは,tRNASecは“UGA”コドンに相補的なアンチコドンをもっている.UGAは通常は終止コドンとして機能する.ところが,mRNAにセレノシステイン挿入配列(Sec-insertion sequence:SECIS)とよばれる配列が存在すると,tRNASecとセレノシステインに特異的な翻訳伸長因子EF-Secとのはたらきにより,UGAコドンは終止コドンではなくSecを指定するコドンと解釈されタンパク質にSecが取り込まれるのである.
最近,筆者らは,ヒトのtRNASecの結晶構造を明らかにした2)(PDB ID:3A3A).これにより,tRNASecはアンチコドン以外に典型的なtRNAとは異なる特徴をもつことがわかった(図1a).まず,tRNASecは長いエクストラアームをもつ(これは,セリンやロイシンのtRNAと共通する).また,標準的なtRNAではアクセプターステムとTステムとをあわせた長さが12塩基対であるのに対し,tRNASecでは13塩基対である.そして,標準的なtRNAのDアームは3~4塩基対のステムと7~11塩基のループで構成されるのに対し,ヒトのtRNASecは6塩基対の長いステムと4塩基のループからなることもわかった.tRNASecのDアームは,この長いDステムが原因で通常のtRNAよりも立体構造上,大きく外側に張り出している.さらに,tRNASecに固有の3次元的な塩基対(U20:G19:C56)も見い出された.これらのtRNASecに特有の構造的な特徴の一部または全部が,セレノシステインの生合成を担う酵素がtRNASecを認識し,ほかの標準的なtRNAと識別するための目印になっているものと予想された.しかし,実際にどの特徴が決定的な要素であり,それがどのように認識されているかという構造的な基盤はこれまで明らかにされていなかった.
さて,ここで古細菌および真核生物におけるセレノシステイン生合成経路を紹介する(図1b).セレノシステインはtRNASecに結合した状態で複数の酵素の作用をへて合成される.まず,tRNASecはセリルtRNA合成酵素(Seryl-tRNA synthetase:SerRS)によってセリンを付加される(Ser-tRNASec).つぎに,この付加されたセリンの側鎖の水酸基(-OH)がO-ホスホセリルtRNAキナーゼ(O-phosphoseryl-tRNA kinase:PSTK)によってリン酸化される.このリン酸化セリンのリン酸基がO-ホスホセリルtRNA:セレノシステイニルtRNA合成酵素(Sep-tRNA:Sec-tRNA synthase:SepSecS)によりセレノール基(-SeH)に変換され,最終的にセレノシステインが結合したtRNASec(Sec-tRNASec)が完成する.
このセレノシステイン生合成経路は大きな危険ととなりあわせのシステムである.1段階目の反応を触媒するセリルtRNA合成酵素はセリンに対応するtRNA(tRNASer)にセリンを付加する機能を兼ねている.細胞内にはセリンのほうがセレノシステインよりも圧倒的に多く存在することから,tRNASerに対するセリンの付加こそがセリルtRNA合成酵素の主たる仕事である.ここで仮に,セリンを付加されたtRNASer(Ser-tRNASer)がtRNASecと誤認され,のちのセレノシステイン生合成経路に入ってしまったとする.そうすると,セリンに対応するコドンをもちながらセレノシステインと結合したtRNASer(Sec-tRNASer)ができてしまう.これでは,結果的にセリンが挿入されるはずの位置にセレノシステインが取り込まれたタンパク質が合成されてしまい,重篤な問題が生じる.すなわち,セリルtRNA合成酵素による反応産物のうちSer-tRNASecだけが選択的にセレノシステイン生合成経路に取り込まれるための,厳密な見分けの機構が不可欠なのである.
では,その厳密な見分けを保証するのはいったいどの段階の反応であろうか? じつは,O-ホスホセリルtRNAキナーゼはSer-tRNASecのみをリン酸化し,Ser-tRNASerを基質としないことが生化学的に示されている3-5)(図1b).つまり,なんらかの機構で2種類のtRNAを厳密に見分けていると考えられる.近年,報告されたO-ホスホセリルtRNAキナーゼとATP類似体との結晶構造(PDB ID:3A4L )からは,O-ホスホセリルtRNAキナーゼがtRNASecのアクセプターステムの長さを認識することが推測された6).しかし,アクセプターステムの長さを変えたtRNASec変異体でもO-ホスホセリルtRNAキナーゼによってリン酸化されることが知られており5),O-ホスホセリルtRNAキナーゼがtRNASecとtRNASerとを見分ける機構は依然として謎のままであった.
今回,筆者らが解析したtRNASecとO-ホスホセリルtRNAキナーゼの複合体の結晶構造により,tRNASecとO-ホスホセリルtRNAキナーゼとの特異的な相互作用のしくみが明らかになり,O-ホスホセリルtRNAキナーゼによるtRNAの厳密な選択性の謎が解けた.
1.tRNASecとO-ホスホセリルtRNAキナーゼとの複合体の結晶構造
筆者らは,古細菌のtRNASecとO-ホスホセリルtRNAキナーゼの複合体の立体構造をX線結晶構造解析により2.4~2.9Åの分解能で決定した(PDB ID:3ADB ,3ADC ,3ADD ).tRNASecはMethanopyrus kandleri由来のものを,O-ホスホセリルtRNAキナーゼはMethanocaldococcus jannaschii由来のものを用いた.M. jannaschiiのO-ホスホセリルtRNAキナーゼはセリンを結合したM. kandleriのtRNASecを効率よくリン酸化するが,tRNASerには作用しない.O-ホスホセリルtRNAキナーゼはホモ2量体を形成しており,単量体1つにつき1分子のtRNASecと結合していた(図2a).古細菌M. kandleriのtRNASecにはヒトtRNASecにおいてみられた前述の特徴がすべて共通していた2).これらの特徴は,古細菌および真核生物においてtRNASecに特有の特徴として広く保存されているものと考えられる.一方,248アミノ酸残基からなるO-ホスホセリルtRNAキナーゼ単量体は,リン酸化活性部位をもつN末端ドメイン(1~172残基)とC末端ドメイン(184~248残基),および,これらをつなぐリンカー(173~183残基)によって構成されていることがわかった.以前に報告されたM. jannaschiiのO-ホスホセリルtRNAキナーゼ単独での結晶構造(PDB ID:3A4L )では,N末端ドメインとC末端ドメインとが近接しひとつの大きなドメインを形成しているようにみえた6).しかし,今回の複合体の結晶構造では,O-ホスホセリルtRNAキナーゼのN末端ドメインとC末端ドメインとは互いに分離しており,それぞれ,tRNASecのアクセプターアーム,Dアームと結合することが明らかになった.また,N末端ドメイン,C末端ドメインのいずれも,tRNASecに特有のアンチコドンとは相互作用しておらず,O-ホスホセリルtRNAキナーゼにとってアンチコドンはtRNA識別の目印としてははたらいていないことがわかった.
2.tRNASecのDアームとO-ホスホセリルtRNAキナーゼのC末端ドメインの相互作用
tRNASecのDアームは6塩基対以上のDステムと4塩基のDループからなり,この特徴は古細菌と真核生物とで保存されている.今回明らかにした複合体の構造で,この長いDステムは通常のtRNAの3~4塩基対のステムの場合よりも大きく外側に張り出していた.また,Dループの残基G19とU20はTループ上のC56と塩基の3つ組(U20:G19:C56)を形成し,同じくDループのU16はtRNASecの内部でTループのU59と塩基対をつくっていた.これらは,いずれもヒトtRNASecにも共通してみられた特徴である2).一方,O-ホスホセリルtRNAキナーゼのC末端ドメインは3本のαヘリックスで構成されており,リンカー部分もあわせてtRNASecのDアームにそうように位置していた.
興味深いことに,O-ホスホセリルtRNAキナーゼのC末端ドメインはtRNASecのDアームの特徴的な構造と重点的に相互作用をしていた(図2b).まず,C末端ドメインおよびリンカーの複数のアミノ酸残基が,Dステムのリン酸および糖の骨格と水素結合ないし静電的な相互作用を形成していた.外側に突き出たtRNASecのDステムの構造と立体的および静電的によく適合しているといえる.さらに,tRNASecの塩基を特異的に認識する相互作用もみられた.tRNASec特有の短いDループはtRNASecの表面上に小さい穴を形成している.この内部にO-ホスホセリルtRNAキナーゼのC末端ドメインのアミノ酸残基Arg195の側鎖がぴったり収まっており,Dループの塩基および骨格のみならず,上記のU59とも水素結合していた(図2c).また,塩基の3つ組(U20:G19:C56)を構成するG19,U20は,Ile202およびIle212,Ser199,Lys203,Lys209と相互作用していた(図2d).このように,C末端ドメインはtRNASec特有の構造である長いDステム,小さいDループ,U20:G19:C56の3つ組,U16:U59ペアに完璧に適合し認識していることがわかった.
つづいて,O-ホスホセリルtRNAキナーゼのリン酸化活性におけるC末端ドメインの重要性を知るため,N末端ドメイン断片(C末端ドメインのないO-ホスホセリルtRNAキナーゼ断片)を調製し全長のO-ホスホセリルtRNAキナーゼと活性を比較した.N末端ドメイン断片では,全長O-ホスホセリルtRNAキナーゼよりリン酸化活性が劇的に低下したことから,C末端ドメインはO-ホスホセリルtRNAキナーゼの活性に決定的な寄与をしていることがわかった.また,O-ホスホセリルtRNAキナーゼとtRNASecとの結合に対するN末端ドメインおよびC末端ドメインの寄与を知るため,全長のO-ホスホセリルtRNAキナーゼ,N末端ドメイン断片,C末端ドメイン断片(N末端ドメインのないO-ホスホセリルtRNAキナーゼ断片)と,tRNASecとのゲルシフト実験を行ったところ,O-ホスホセリルtRNAキナーゼとtRNASecとの強い結合には,C末端ドメインによる結合が必須であることが示された.さらに,全長O-ホスホセリルtRNAキナーゼおよびC末端ドメイン断片とのtRNASecの解離定数を求めると,それぞれ,48 nM,39 nMであり,C末端ドメインがtRNASecへの結合を実質的に担っていることがわかった.O-ホスホセリルtRNAキナーゼのtRNASerに対する解離定数が1.3μMであるという報告をふまえると7),O-ホスホセリルtRNAキナーゼのC末端ドメインとtRNASecのDアームとの相補的で強い結合が,tRNASecのみを認識しtRNASerには作用しないというO-ホスホセリルtRNAキナーゼの高い特異性の基盤となっていると結論づけられた.
以上のように,立体構造および生化学的な解析の両面から,リン酸化活性部位のない小さなドメインであるC末端ドメインこそがtRNASecとtRNASerとの見分けに決定的であることが示された(図3).この識別はtRNAのDアームの形状にもとづいており選択性が高い.これにより,20種類の標準アミノ酸が用いられる通常の翻訳に影響を及ぼすことなく,セレン含有タンパク質の特定部位のみに正確にセレノシステインを導入することが可能になっているのだろう.
3.O-ホスホセリルtRNAキナーゼのC末端ドメインのもうひとつの役割の可能性
筆者らが明らかにしたtRNASec-O-ホスホセリルtRNAキナーゼ複合体の結晶構造から,柔軟性の高いリンカーでつながっているN末端ドメインとC末端ドメインとは互いに独立してtRNASecと結合・解離できることが示された.また,ゲルシフト実験により,O-ホスホセリルtRNAキナーゼのC末端ドメインとtRNASecのDアームは,N末端ドメインとアクセプターアームよりもはるかに高い親和性をもつことが示された.これらのことから,O-ホスホセリルtRNAキナーゼによるリン酸化反応ののち,C末端ドメインはDアームとの結合を保持したままN末端ドメインのみがアクセプターアームから離れうると考えられた.そこにセレノシステイン合成の次段階の反応を触媒する酵素であるO-ホスホセリルtRNA:セレノシステイニルtRNA合成酵素が近づいてN末端ドメインとすばやく入れ替われば,加水分解されやすいtRNASecとアミノ酸との結合を保護しつつtRNASecを受け渡すことができる.O-ホスホセリルtRNAキナーゼのリン酸化活性部位がセリン以外のアミノ酸も許容するという生化学的なデータは,この推論と矛盾しない4).実際にそうであれば,C末端ドメインとtRNASecとの相互作用は,tRNAの見分けのみならず,セレノシステインの生合成経路の一貫した品質保証にも役立っているといえるだろう.
おわりに
今回,O-ホスホセリルtRNAキナーゼがtRNASec特有の構造を認識していること,さらに,構造の認識に特化したドメインであるC末端ドメインをもっていることがわかり,古細菌および真核生物におけるSer-tRNASecを選択的にセレノシステイン合成経路に導く分子機構が解明された.C末端ドメインは,あえてアンチコドンではなく,tRNASecのDアームに保存された複数の特徴的な構造と強力かつ特異的に相互作用する.この相互作用が,ここで論じたようなtRNAの見分けの機構以外においてもなんらかの意味をもっていたとしても不思議ではない.さらに,細胞内では稀少な存在であるはずのセレノシステインの生合成にかかわるタンパク質が基質の受け渡しの際にどのように高い効率を確保しているかなど,いまだ明らかでないことは多い.今後,構造解析を含むさまざまなアプローチによりセレノシステイン生合成への理解がさらに深まることを期待する.“21番目のアミノ酸”であるセレノシステインの生合成系は,生物が進化の過程で必要に応じて遺伝暗号を拡張した結果,生まれたものと考えられる.したがって,セレノシステイン生合成のしくみの理解は,多くの有用な機能をもった天然または非天然のアミノ酸をタンパク質の目的の位置に導入する系の開発への重要な基礎ともなるであろう.
文 献
- Holben, D. H. & Smith, A. M.: The diverse role of selenium within selenoproteins: a review. J. Am. Diet. Assoc., 99, 836-843 (1999)[PubMed]
- Itoh, Y., Chiba, S., Sekine, S. et al.: Crystal structure of human selenocysteine tRNA. Nucleic Acids Res., 37, 6259-6268 (2009)[PubMed]
- Amberg, R., Mizutani, T., Wu, X. Q. et al.: Selenocysteine synthesis in mammalia: an identity switch from tRNASer to tRNASec. J. Mol. Biol., 263, 8-19 (1996)[PubMed]
- Carlson, B. A., Xu, X. M., Kryukov, G. V. et al.: Identification and characterization of phosphoseryl-tRNA[Ser]Sec kinase. Proc. Natl. Acad. Sci. USA, 101, 12848-12853 (2004)[PubMed]
- Sherrer, R. L., Ho, J. M., Soll, D.: Divergence of selenocysteine tRNA recognition by archaeal and eukaryotic O-phosphoseryl-tRNASec kinase. Nucleic Acids Res., 36, 1871-1880 (2008)[PubMed]
- Araiso, Y., Sherrer, R. L., Ishitani, R. et al.: Structure of a tRNA-dependent kinase essential for selenocysteine decoding. Proc. Natl. Acad. Sci. USA, 106, 16215-16220 (2009)[PubMed]
- Sherrer, R. L., O'Donoghue, P. & Soll, D.: Characterization and evolutionary history of an archaeal kinase involved in selenocysteinyl-tRNA formation. Nucleic Acids Res., 36, 1247-1259 (2008)[PubMed]
著者プロフィール
東京大学大学院医学系研究科修士課程 在学中.
伊藤弓弦(Yuzuru Itoh)
東京大学分子細胞生物学研究所 助教.
関根俊一(Shun-ichi Sekine)
東京大学大学院理学系研究科 特任准教授.
横山茂之(Shigeyuki Yokoyama)
理化学研究所生命分子システム基盤研究領域 領域長,東京大学大学院理学系研究科 兼任教授.
© 2010 千葉志穂・伊藤弓弦・関根俊一・横山茂之 Licensed under CC 表示 2.1 日本