複製フォークに呼び込まれたDbf4依存性キナーゼは減数分裂において複製と相同組換えとをリンクする
村上 創・Scott Keeney
(米国Memorial Sloan Kettering Cancer Center,Molecular Biology Program)
email:村上 創
DOI: 10.7875/first.author.2014.114
Temporospatial coordination of meiotic DNA replication and recombination via DDK recruitment to replisomes.
Hajime Murakami, Scott Keeney
Cell, 158, 861-873 (2014)
真核生物が減数分裂を開始すると,まず複製が起こり,これにつづいて相同染色体のあいだで相同組換えが起こる.減数分裂における相同組換えがDNA 2本鎖切断を契機として開始されることが発見されてから四半世紀がたち,減数分裂においてDNA 2本鎖切断をつかさどるタンパク質,および,DNA 2本鎖切断の頻度および分布を制御する染色体における種々の特性が明らかになった.しかし,DNA 2本鎖切断の開始の時期を制御する機構,とくに,先行する減数分裂における複製とのリンクについてはほとんど解明されていない.長いあいだ,DNA 2本鎖切断の形成される時期はその染色体の領域の複製の時期により局所的に決定されることが知られていた.今回,筆者らは,出芽酵母において,DNA複製装置の構成タンパク質であるTof1およびCsm3が減数分裂においてDbf4依存性キナーゼを複製フォークに呼び込み,DNA 2本鎖切断の形成に必須であるMer2のリン酸化が複製フォークの周辺において起こることを明らかにした.この発見により,Mer2のリン酸化というDNA 2本鎖切断の形成の初期の段階における必要条件が複製をおえた染色体の領域において限定的にみたされ,減数分裂における複製とDNA 2本鎖切断の形成とを時間的かつ空間的に同調する一連の分子機構が解明された.複製フォークへのキナーゼの呼び込みは,複製と染色体において起こるさまざまなイベントとを時間的かつ空間的に同調させるために一般に備わった分子機構なのかもしれない.
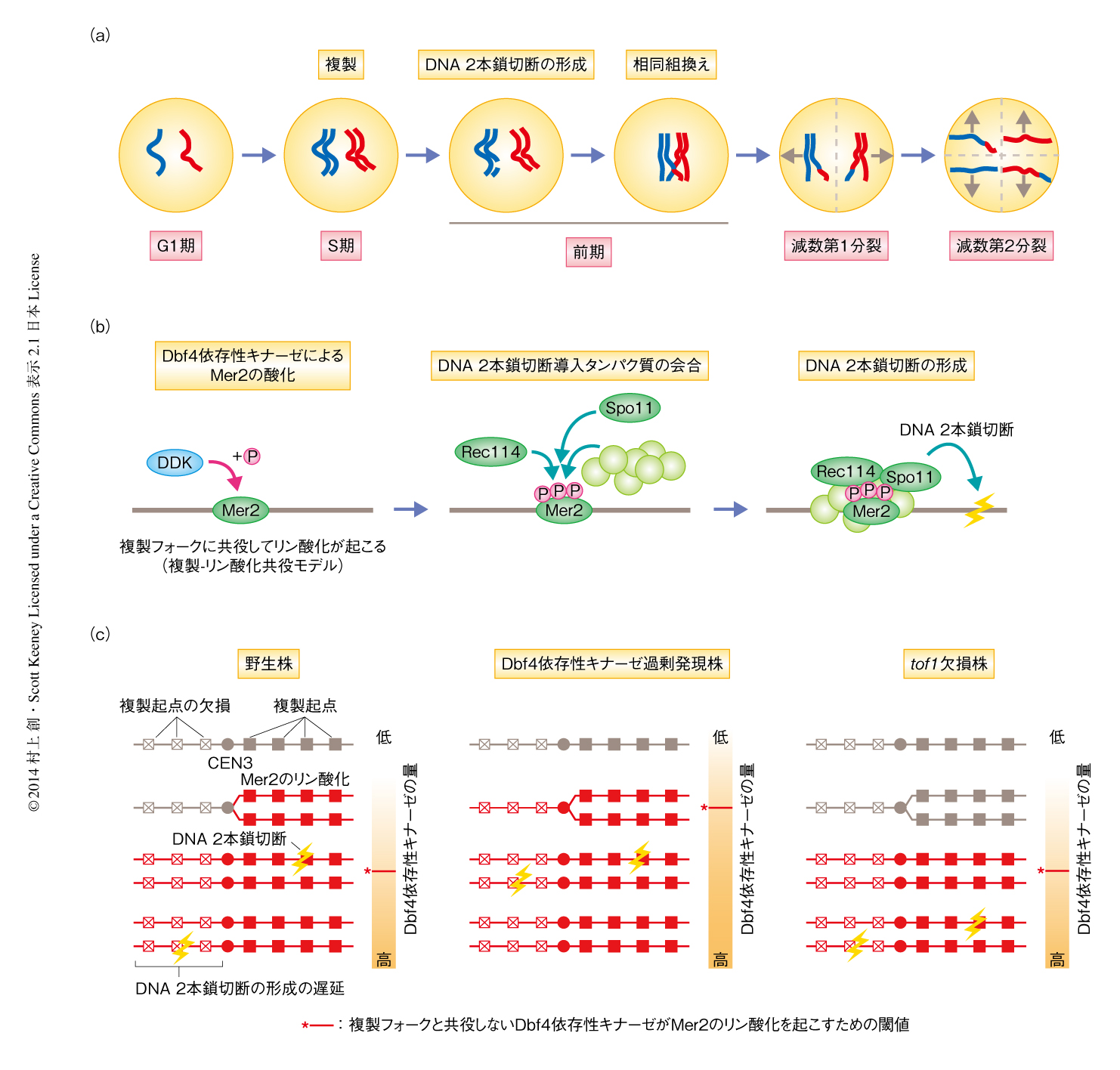
細胞における各種のイベントは正常な順序で起こるようにつねに制御されている.今回,筆者らは,多数のイベントが順序正しく起こる減数分裂において,減数分裂の初期に起こる複製と相同組換えとを直接的にリンクさせるしくみを解明した.減数分裂は1回の複製とこれにつづく連続した2回の細胞分裂からなり,2倍体の細胞から1倍体の配偶子が形成される(図1a).減数分裂の初期においては,複製ののち,相同染色体のあいだで相同組換えが起こり,それにつづく減数第1分裂を正確に行うために必須の相同染色体のあいだの物理的な結合(交差)が形成される.減数分裂における相同組換えはあらかじめ細胞にプログラムされたDNA 2本鎖切断の形成を契機として開始する.この自らのゲノムを切断する過程は時期,頻度,分布が厳密に制御されていると予想される.
2000年,出芽酵母の第3染色体の左腕におけるすべての複製起点を欠損させることにより,この領域において特異的に複製を遅延させ,このとき,DNA 2本鎖切断の形成の時期を第3染色体の左腕と右腕について比較することにより,複製の遅延を起こした左腕においてのみDNA 2本鎖切断の形成が遅延することが見い出された1).この発見により,DNA 2本鎖切断の形成はS期の完了をまってすべての染色体の領域において同時に開始されるのではなく,複製が完了した染色体の領域において局所的にDNA 2本鎖切断の形成が誘導され,それゆえ,おのおの染色体の領域における複製のタイミングがDNA 2本鎖切断の形成の時期を決定することが示唆された.この現象が発見された当初は,複製をおえた染色体がDNA 2本鎖切断の形成に必須であると考えられていたが,のちにこの考えは否定され2),複製とDNA 2本鎖切断の形成とが時間的かつ空間的に同調するという現象の真相は未解明のまま残されていた.そののち,DNA 2本鎖切断の形成の時期の制御機構を解明する鍵となる報告がいくつかなされた.減数分裂におけるDNA 2本鎖切断は,異なる生物種のあいだで広く保存されたSpo11により導入され,出芽酵母ではSpo11のほかにDNA 2本鎖切断導入タンパク質とよばれるさらに9つのタンパク質が必要とされることが知られている3).減数分裂の初期に染色体においてDNA 2本鎖切断導入タンパク質のひとつであるMer2がDbf4依存性キナーゼによりリン酸化されることにより,Spo11を含むDNA 2本鎖切断導入タンパク質が染色体において会合できるようになり,DNA 2本鎖切断が形成されることが示された4,5).また,Mer2それ自体の染色体へ結合はリン酸化を必要としないこと6),Mer2のリン酸化からDNA 2本鎖切断の形成には約90分かかることが示され5),Dbf4依存性キナーゼによるMer2のリン酸化が一連のDNA 2本鎖切断の形成の過程をひき起こすためのライセンス反応であることが示唆された(図1b).Dbf4依存性キナーゼは有糸分裂および減数分裂における複製起点の活性化に必要であり,また,Dbf4依存性キナーゼの量は減数分裂の初期に低少なく,減数分裂の進行にしたがい多くなることが知られている.減数分裂において複製を開始するために必要とされるDbf4依存性キナーゼの量は,DNA 2本鎖切断の形成を誘導するのに必要な量よりも少ないことが示されている5,7).このDbf4依存性キナーゼに対する感受性の違いから,DNA 2本鎖切断の形成は複製ののちに制御されていることは説明できるが,これだけでは複製とDNA 2本鎖切断の形成とが局所的に同調していることは説明できない.
以上,筆者らは,既知の現象を総合的に説明するモデルとして,DNA 2本鎖切断の形成の初期の過程を担うMer2のリン酸化が複製フォークの形成と共役して起こると仮定し8)(図1c),この研究において検証した.
このモデルは,DNA 2本鎖切断の形成に必要なMer2のリン酸化が,複製フォークが通過する染色体の領域にのみ限定的に起こることを想定している.これには,細胞においてDbf4依存性キナーゼの量が少なくほとんどが複製フォークの近傍に存在していることが条件になる.これを検証するため,減数分裂においてDbf4依存性キナーゼを過剰に発現させた.この過剰発現により複製していない染色体の領域にあるMer2をリン酸化することができれば,DNA 2本鎖切断の形成の初期の過程は複製の時期とは関係なく開始され,結果として,複製とDNA 2本鎖切断の形成との同調が破壊されると予想された.予想どおり,複製の遅延に起因した第3染色体の左腕におけるDNA 2本鎖切断の形成の遅延は,Dbf4依存性キナーゼの過剰発現により起こらなくなった(図1c).この結果から,Dbf4依存性キナーゼの過剰発現により複製とDNA 2本鎖切断の形成との同調は失われると結論された.
つぎの疑問点は,何が複製の完了した染色体の領域にDbf4依存性キナーゼを呼び込むのかであった.逆遺伝学的なアプローチによりいくつかの候補タンパク質を検証してみたが,すべて失敗におわった.なかばあきらめかけた際に,一報の論文が目にとまった.その論文では,分裂酵母においてSwi1とDfp1とが相互作用することが示されていた9).これらは,出芽酵母においてはDbf4とTof1に相当する.Dbf4はDbf4依存性キナーゼの構成タンパク質である.また,Tof1はCsm3およびMrc1と複製フォーク保護複合体を形成し,この複製フォーク保護複合体は複製フォークとともに染色体を移動することが知られている10).すなわち,もし出芽酵母の減数分裂においてTof1とDbf4とが相互作用するのであれば,Tof1はDbf4依存性キナーゼを複製フォークに呼び込むことが予想された.この可能性を検証するため,Tof1とDbf4との相互作用を免疫沈降法により調べ,Tof1とDbf4とが減数分裂において相互作用していることを確認した.ついで,tof1欠損株において複製とDNA 2本鎖切断の形成との同調について調べたところ,複製の遅延に起因したDNA 2本鎖切断の形成の遅延は起こらなくなっていた(図1c).この結果は,複製に共役したリン酸化の機構がTof1の欠損により破壊され,細胞におけるDbf4依存性キナーゼが一定の量,つまり,複製に共役しない遊離のDbf4依存性キナーゼがMer2をリン酸化するための閾値をこえた時点で,個々の染色体の領域における複製の時期とは関係なくMer2のリン酸化が起こることを示唆した(図1c).ここから予想されたように,tof1欠損株では野生株やDbf4依存性キナーゼを過剰に発現した株に比べ細胞の全体においてDNA 2本鎖切断の形成が遅れることが確認された.また,csm3欠損株ではtof1欠損株と同様に複製の遅延に起因したDNA 2本鎖切断の形成の遅延は起こらなかったが,mrc1欠損株では野生株と同様に複製の遅延に起因してDNA 2本鎖切断の形成は遅延した.以上から,Tof1およびCsm3が複製とDNA 2本鎖切断の形成の同調に必要であることが示された.
ここでかりに,Tof1が複製フォークとDbf4依存性キナーゼとをつなぐリンカーとして機能しているのであれば,Dbf4依存性キナーゼと複製フォークとを人工的に結びつけることによりTof1の必要性をバイパスできるであろうと考えた.この仮説を検証するため,複製フォークの構成タンパク質であるCdc45とDbf4との融合タンパク質をtof1欠損株において発現させたところ,予想どおり,Cdc45-Dbf4融合タンパク質を発現した株においては,Tof1が存在しないにもかかわらず複製とDNA 2本鎖切断の形成との同調が確認された.これらの結果から,Tof1およびCsm3がDbf4依存性キナーゼを複製フォークへ呼び込むことにより,複製とDNA 2本鎖切断の形成との時間的かつ空間的な同調が確立されると結論された.
このモデルは,複製フォークが通過する際にMer2のリン酸化が起こると仮定している.これが正しければ,Mer2のリン酸化も複製の時期により決定されるはずである.これを検証するため,クロマチン免疫沈降(chromatin immunoprecipitation:ChIP)法と次世代シークエンサーとを組み合わせたChIP-seq法により,染色体におけるMer2のリン酸化の状態について調べた.抗リン酸化Mer2抗体が得られなかったことから,DNA 2本鎖切断導入タンパク質のひとつでありMer2のリン酸化により染色体と結合するRec114について解析することにより,間接的にMer2のリン酸化の状態を調べた.その結果,Rec114は複製起点を欠損した第3染色体の左腕において特異的に染色体への結合が遅延した.さらに,Rec114の染色体への結合の時期を詳細に解析したところ,染色体の領域ごとにRec114の結合の時期はすべて異なることが観察された.また,このRec114の結合プロファイルはほかの染色体においても複製プロファイルと相関し,この相関はTof1の欠損によりいちじるしく低下した.以上により,染色体の領域のあいだでみられる複製の時期の差異が,おのおのの領域へのRec114の結合の時期に反映されていることが示された.また,Rec114の染色体への結合(あるいは,Mer2のリン酸化の状態)と複製は,複製起点の欠損により複製が遅延した領域のほかの領域でも,Tof1に依存して時間的かつ空間的に同調されていることが示唆された.
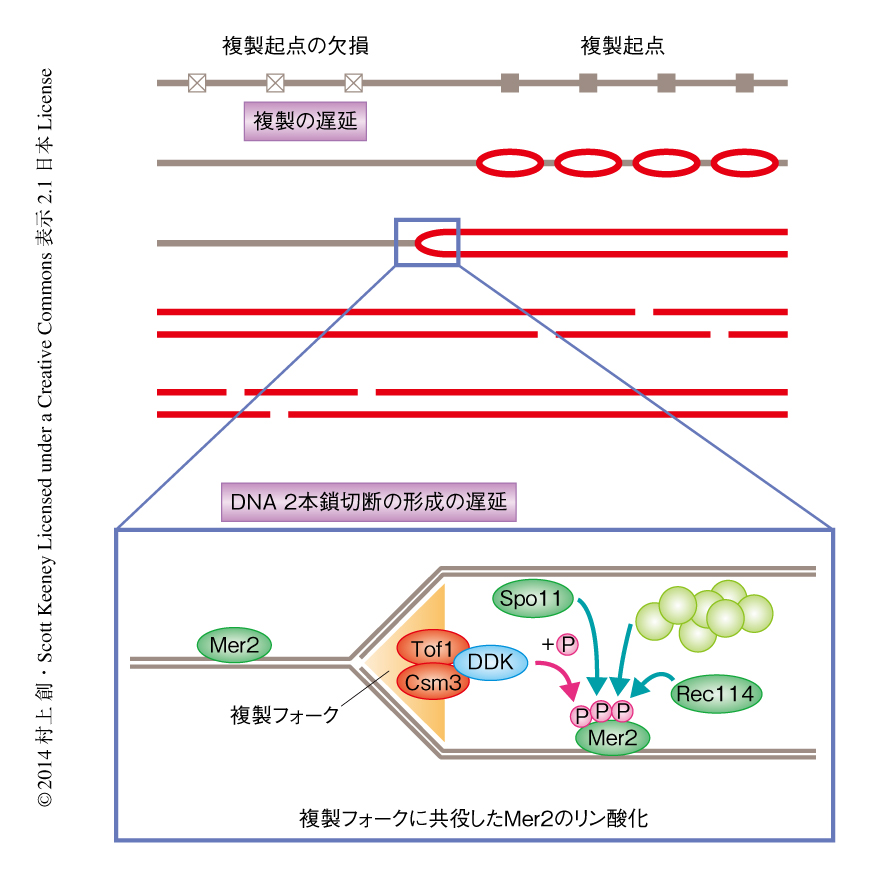
この研究により,長年にわたり未解明であった,減数分裂における複製とDNA 2本鎖切断の形成との同調の分子機構が明らかにされた(図2).この複製とDNA 2本鎖切断の形成との直接的なリンクにより,複製が完了した染色体の領域から即座にDNA 2本鎖切断の形成が誘導できるだけでなく,複製されていない染色体の領域におけるあやまったDNA 2本鎖切断の形成もふせぐことができる.また,S期においてはすべての染色体が例外なく複製フォークの通過をうける.この複製フォークにDNA 2本鎖切断の形成のオン/オフをつかさどるスイッチを組み込むことにより,ゲノムの全体をスキャンしてのちのDNA 2本鎖切断の形成および相同組換えに適するかどうかを査定するという機構がなりたっているのかもしれない.
また,この研究により,いくつかの新しい知見がもたらされた.第1に,減数分裂における相同組換えの初期の過程がS期の完了をまつことなく開始することがはじめて実験的に示された.第2に,染色体を移動する複製フォークに結合した複製フォーク保護複合体はDbf4依存性キナーゼと相互作用することが示された.これは,有糸分裂における複製や姉妹染色体の接着など,複製フォーク保護複合体の機能を理解するうえで有益なパラダイムを提供する.第3に,Dbf4依存性キナーゼが鍵となるリン酸化を起こすとき,細胞におけるその量は検出限界より少ないこと,しかも驚くべきことに,このように低い量に制御されていることがDbf4依存性キナーゼによる制御機構において必要条件になっていることが示された.これは,どのように細胞におけるイベントを複製の完了した染色体の領域のみに限定しているかを理解するうえで重要な知見である.今回,解明された新しい制御機構に関与するタンパク質は真核生物に広く保存されたユビキタスなものであり,この機構が生物種をこえて染色体におけるさまざまな過程に関与している可能性がある.
略歴:2004年 埼玉大学大学院理工学研究科 修了,同年 フランスCurie Instituteポスドク,2006年 米国Memorial Sloan Kettering Cancer Centerリサーチスカラー,2011年 同 リサーチアソシエートを経て,2014年より同 シニアリサーチサイエンティスト.
抱負:誰にでもわかる研究をとおして感動をとどけたい.
Scott Keeney
米国Memorial Sloan Kettering Cancer CenterにてProfessor.
研究室URL:http://www.mskcc.org/research/lab/scott-keeney
© 2014 村上 創・Scott Keeney Licensed under CC 表示 2.1 日本
(米国Memorial Sloan Kettering Cancer Center,Molecular Biology Program)
email:村上 創
DOI: 10.7875/first.author.2014.114
Temporospatial coordination of meiotic DNA replication and recombination via DDK recruitment to replisomes.
Hajime Murakami, Scott Keeney
Cell, 158, 861-873 (2014)
要 約
真核生物が減数分裂を開始すると,まず複製が起こり,これにつづいて相同染色体のあいだで相同組換えが起こる.減数分裂における相同組換えがDNA 2本鎖切断を契機として開始されることが発見されてから四半世紀がたち,減数分裂においてDNA 2本鎖切断をつかさどるタンパク質,および,DNA 2本鎖切断の頻度および分布を制御する染色体における種々の特性が明らかになった.しかし,DNA 2本鎖切断の開始の時期を制御する機構,とくに,先行する減数分裂における複製とのリンクについてはほとんど解明されていない.長いあいだ,DNA 2本鎖切断の形成される時期はその染色体の領域の複製の時期により局所的に決定されることが知られていた.今回,筆者らは,出芽酵母において,DNA複製装置の構成タンパク質であるTof1およびCsm3が減数分裂においてDbf4依存性キナーゼを複製フォークに呼び込み,DNA 2本鎖切断の形成に必須であるMer2のリン酸化が複製フォークの周辺において起こることを明らかにした.この発見により,Mer2のリン酸化というDNA 2本鎖切断の形成の初期の段階における必要条件が複製をおえた染色体の領域において限定的にみたされ,減数分裂における複製とDNA 2本鎖切断の形成とを時間的かつ空間的に同調する一連の分子機構が解明された.複製フォークへのキナーゼの呼び込みは,複製と染色体において起こるさまざまなイベントとを時間的かつ空間的に同調させるために一般に備わった分子機構なのかもしれない.
はじめに
細胞における各種のイベントは正常な順序で起こるようにつねに制御されている.今回,筆者らは,多数のイベントが順序正しく起こる減数分裂において,減数分裂の初期に起こる複製と相同組換えとを直接的にリンクさせるしくみを解明した.減数分裂は1回の複製とこれにつづく連続した2回の細胞分裂からなり,2倍体の細胞から1倍体の配偶子が形成される(図1a).減数分裂の初期においては,複製ののち,相同染色体のあいだで相同組換えが起こり,それにつづく減数第1分裂を正確に行うために必須の相同染色体のあいだの物理的な結合(交差)が形成される.減数分裂における相同組換えはあらかじめ細胞にプログラムされたDNA 2本鎖切断の形成を契機として開始する.この自らのゲノムを切断する過程は時期,頻度,分布が厳密に制御されていると予想される.
2000年,出芽酵母の第3染色体の左腕におけるすべての複製起点を欠損させることにより,この領域において特異的に複製を遅延させ,このとき,DNA 2本鎖切断の形成の時期を第3染色体の左腕と右腕について比較することにより,複製の遅延を起こした左腕においてのみDNA 2本鎖切断の形成が遅延することが見い出された1).この発見により,DNA 2本鎖切断の形成はS期の完了をまってすべての染色体の領域において同時に開始されるのではなく,複製が完了した染色体の領域において局所的にDNA 2本鎖切断の形成が誘導され,それゆえ,おのおの染色体の領域における複製のタイミングがDNA 2本鎖切断の形成の時期を決定することが示唆された.この現象が発見された当初は,複製をおえた染色体がDNA 2本鎖切断の形成に必須であると考えられていたが,のちにこの考えは否定され2),複製とDNA 2本鎖切断の形成とが時間的かつ空間的に同調するという現象の真相は未解明のまま残されていた.そののち,DNA 2本鎖切断の形成の時期の制御機構を解明する鍵となる報告がいくつかなされた.減数分裂におけるDNA 2本鎖切断は,異なる生物種のあいだで広く保存されたSpo11により導入され,出芽酵母ではSpo11のほかにDNA 2本鎖切断導入タンパク質とよばれるさらに9つのタンパク質が必要とされることが知られている3).減数分裂の初期に染色体においてDNA 2本鎖切断導入タンパク質のひとつであるMer2がDbf4依存性キナーゼによりリン酸化されることにより,Spo11を含むDNA 2本鎖切断導入タンパク質が染色体において会合できるようになり,DNA 2本鎖切断が形成されることが示された4,5).また,Mer2それ自体の染色体へ結合はリン酸化を必要としないこと6),Mer2のリン酸化からDNA 2本鎖切断の形成には約90分かかることが示され5),Dbf4依存性キナーゼによるMer2のリン酸化が一連のDNA 2本鎖切断の形成の過程をひき起こすためのライセンス反応であることが示唆された(図1b).Dbf4依存性キナーゼは有糸分裂および減数分裂における複製起点の活性化に必要であり,また,Dbf4依存性キナーゼの量は減数分裂の初期に低少なく,減数分裂の進行にしたがい多くなることが知られている.減数分裂において複製を開始するために必要とされるDbf4依存性キナーゼの量は,DNA 2本鎖切断の形成を誘導するのに必要な量よりも少ないことが示されている5,7).このDbf4依存性キナーゼに対する感受性の違いから,DNA 2本鎖切断の形成は複製ののちに制御されていることは説明できるが,これだけでは複製とDNA 2本鎖切断の形成とが局所的に同調していることは説明できない.
以上,筆者らは,既知の現象を総合的に説明するモデルとして,DNA 2本鎖切断の形成の初期の過程を担うMer2のリン酸化が複製フォークの形成と共役して起こると仮定し8)(図1c),この研究において検証した.
1.Dbf4依存性キナーゼは複製とDNA 2本鎖切断の形成とをリンクする
このモデルは,DNA 2本鎖切断の形成に必要なMer2のリン酸化が,複製フォークが通過する染色体の領域にのみ限定的に起こることを想定している.これには,細胞においてDbf4依存性キナーゼの量が少なくほとんどが複製フォークの近傍に存在していることが条件になる.これを検証するため,減数分裂においてDbf4依存性キナーゼを過剰に発現させた.この過剰発現により複製していない染色体の領域にあるMer2をリン酸化することができれば,DNA 2本鎖切断の形成の初期の過程は複製の時期とは関係なく開始され,結果として,複製とDNA 2本鎖切断の形成との同調が破壊されると予想された.予想どおり,複製の遅延に起因した第3染色体の左腕におけるDNA 2本鎖切断の形成の遅延は,Dbf4依存性キナーゼの過剰発現により起こらなくなった(図1c).この結果から,Dbf4依存性キナーゼの過剰発現により複製とDNA 2本鎖切断の形成との同調は失われると結論された.
2.Tof1はDbf4依存性キナーゼを複製フォークに呼び込む
つぎの疑問点は,何が複製の完了した染色体の領域にDbf4依存性キナーゼを呼び込むのかであった.逆遺伝学的なアプローチによりいくつかの候補タンパク質を検証してみたが,すべて失敗におわった.なかばあきらめかけた際に,一報の論文が目にとまった.その論文では,分裂酵母においてSwi1とDfp1とが相互作用することが示されていた9).これらは,出芽酵母においてはDbf4とTof1に相当する.Dbf4はDbf4依存性キナーゼの構成タンパク質である.また,Tof1はCsm3およびMrc1と複製フォーク保護複合体を形成し,この複製フォーク保護複合体は複製フォークとともに染色体を移動することが知られている10).すなわち,もし出芽酵母の減数分裂においてTof1とDbf4とが相互作用するのであれば,Tof1はDbf4依存性キナーゼを複製フォークに呼び込むことが予想された.この可能性を検証するため,Tof1とDbf4との相互作用を免疫沈降法により調べ,Tof1とDbf4とが減数分裂において相互作用していることを確認した.ついで,tof1欠損株において複製とDNA 2本鎖切断の形成との同調について調べたところ,複製の遅延に起因したDNA 2本鎖切断の形成の遅延は起こらなくなっていた(図1c).この結果は,複製に共役したリン酸化の機構がTof1の欠損により破壊され,細胞におけるDbf4依存性キナーゼが一定の量,つまり,複製に共役しない遊離のDbf4依存性キナーゼがMer2をリン酸化するための閾値をこえた時点で,個々の染色体の領域における複製の時期とは関係なくMer2のリン酸化が起こることを示唆した(図1c).ここから予想されたように,tof1欠損株では野生株やDbf4依存性キナーゼを過剰に発現した株に比べ細胞の全体においてDNA 2本鎖切断の形成が遅れることが確認された.また,csm3欠損株ではtof1欠損株と同様に複製の遅延に起因したDNA 2本鎖切断の形成の遅延は起こらなかったが,mrc1欠損株では野生株と同様に複製の遅延に起因してDNA 2本鎖切断の形成は遅延した.以上から,Tof1およびCsm3が複製とDNA 2本鎖切断の形成の同調に必要であることが示された.
ここでかりに,Tof1が複製フォークとDbf4依存性キナーゼとをつなぐリンカーとして機能しているのであれば,Dbf4依存性キナーゼと複製フォークとを人工的に結びつけることによりTof1の必要性をバイパスできるであろうと考えた.この仮説を検証するため,複製フォークの構成タンパク質であるCdc45とDbf4との融合タンパク質をtof1欠損株において発現させたところ,予想どおり,Cdc45-Dbf4融合タンパク質を発現した株においては,Tof1が存在しないにもかかわらず複製とDNA 2本鎖切断の形成との同調が確認された.これらの結果から,Tof1およびCsm3がDbf4依存性キナーゼを複製フォークへ呼び込むことにより,複製とDNA 2本鎖切断の形成との時間的かつ空間的な同調が確立されると結論された.
3.Mer2のリン酸化は複製と共役して起こる
このモデルは,複製フォークが通過する際にMer2のリン酸化が起こると仮定している.これが正しければ,Mer2のリン酸化も複製の時期により決定されるはずである.これを検証するため,クロマチン免疫沈降(chromatin immunoprecipitation:ChIP)法と次世代シークエンサーとを組み合わせたChIP-seq法により,染色体におけるMer2のリン酸化の状態について調べた.抗リン酸化Mer2抗体が得られなかったことから,DNA 2本鎖切断導入タンパク質のひとつでありMer2のリン酸化により染色体と結合するRec114について解析することにより,間接的にMer2のリン酸化の状態を調べた.その結果,Rec114は複製起点を欠損した第3染色体の左腕において特異的に染色体への結合が遅延した.さらに,Rec114の染色体への結合の時期を詳細に解析したところ,染色体の領域ごとにRec114の結合の時期はすべて異なることが観察された.また,このRec114の結合プロファイルはほかの染色体においても複製プロファイルと相関し,この相関はTof1の欠損によりいちじるしく低下した.以上により,染色体の領域のあいだでみられる複製の時期の差異が,おのおのの領域へのRec114の結合の時期に反映されていることが示された.また,Rec114の染色体への結合(あるいは,Mer2のリン酸化の状態)と複製は,複製起点の欠損により複製が遅延した領域のほかの領域でも,Tof1に依存して時間的かつ空間的に同調されていることが示唆された.
おわりに
この研究により,長年にわたり未解明であった,減数分裂における複製とDNA 2本鎖切断の形成との同調の分子機構が明らかにされた(図2).この複製とDNA 2本鎖切断の形成との直接的なリンクにより,複製が完了した染色体の領域から即座にDNA 2本鎖切断の形成が誘導できるだけでなく,複製されていない染色体の領域におけるあやまったDNA 2本鎖切断の形成もふせぐことができる.また,S期においてはすべての染色体が例外なく複製フォークの通過をうける.この複製フォークにDNA 2本鎖切断の形成のオン/オフをつかさどるスイッチを組み込むことにより,ゲノムの全体をスキャンしてのちのDNA 2本鎖切断の形成および相同組換えに適するかどうかを査定するという機構がなりたっているのかもしれない.
また,この研究により,いくつかの新しい知見がもたらされた.第1に,減数分裂における相同組換えの初期の過程がS期の完了をまつことなく開始することがはじめて実験的に示された.第2に,染色体を移動する複製フォークに結合した複製フォーク保護複合体はDbf4依存性キナーゼと相互作用することが示された.これは,有糸分裂における複製や姉妹染色体の接着など,複製フォーク保護複合体の機能を理解するうえで有益なパラダイムを提供する.第3に,Dbf4依存性キナーゼが鍵となるリン酸化を起こすとき,細胞におけるその量は検出限界より少ないこと,しかも驚くべきことに,このように低い量に制御されていることがDbf4依存性キナーゼによる制御機構において必要条件になっていることが示された.これは,どのように細胞におけるイベントを複製の完了した染色体の領域のみに限定しているかを理解するうえで重要な知見である.今回,解明された新しい制御機構に関与するタンパク質は真核生物に広く保存されたユビキタスなものであり,この機構が生物種をこえて染色体におけるさまざまな過程に関与している可能性がある.
文 献
- Borde, V., Goldman, A. S. & Lichten, M.: Direct coupling between meiotic DNA replication and recombination initiation. Science, 290, 806-809 (2000)[PubMed]
- Hochwagen, A., Tham, W. H., Brar, G. A. et al.: The FK506 binding protein Fpr3 counteracts protein phosphatase 1 to maintain meiotic recombination checkpoint activity. Cell, 122, 861-873 (2005)[PubMed]
- Keeney, S.: Spo11 and the formation of DNA double strand breaks in meiosis. Genome Dyn. Stab., 2, 81-123 (2008)[PubMed]
- Sasanuma, H., Hirota, K., Fukuda, T. et al.: Cdc7-dependent phosphorylation of Mer2 facilitates initiation of yeast meiotic recombination. Genes Dev., 22, 398-410 (2008)[PubMed]
- Wan, L., Niu, H., Futcher, B. et al.: Cdc28-Clb5 (CDK-S) and Cdc7-Dbf4 (DDK) collaborate to initiate meiotic recombination in yeast. Genes Dev., 22, 386-397 (2008)[PubMed]
- Panizza, S., Mendoza, M. A., Berlinger, M. et al.: Spo11-accessory proteins link double-strand break sites to the chromosome axis in early meiotic recombination. Cell, 146, 372-383 (2011)[PubMed]
- Matos, J., Lipp, J. J., Bogdanova, A. et al.: Dbf4-dependent CDC7 kinase links DNA replication to the segregation of homologous chromosomes in meiosis I. Cell, 135, 662-678 (2008)[PubMed]
- Murakami, H. & Keeney, S.: Regulating the formation of DNA double-strand breaks in meiosis. Genes Dev., 22, 286-292 (2008)[PubMed]
- Matsumoto, S., Ogino, K., Noguchi, E. et al.: Hsk1-Dfp1/Him1, the Cdc7-Dbf4 kinase in Schizosaccharomyces pombe, associates with Swi1, a component of the replication fork protection complex. J. Biol. Chem., 280, 42536-42542 (2005)[PubMed]
- Katou, Y., Kanoh, Y., Bando, M. et al.: S-phase checkpoint proteins Tof1 and Mrc1 form a stable replication-pausing complex. Nature, 424, 1078-1083 (2003)[PubMed]
著者プロフィール
略歴:2004年 埼玉大学大学院理工学研究科 修了,同年 フランスCurie Instituteポスドク,2006年 米国Memorial Sloan Kettering Cancer Centerリサーチスカラー,2011年 同 リサーチアソシエートを経て,2014年より同 シニアリサーチサイエンティスト.
抱負:誰にでもわかる研究をとおして感動をとどけたい.
Scott Keeney
米国Memorial Sloan Kettering Cancer CenterにてProfessor.
研究室URL:http://www.mskcc.org/research/lab/scott-keeney
© 2014 村上 創・Scott Keeney Licensed under CC 表示 2.1 日本