シロイヌナズナにおける能動的なDNA脱メチル化にはαクリスタリンドメインをもつタンパク質IDM2が関与する
三木 大介
(中国Shanghai Institutes for Biological Sciences,Shanghai Center for Plant Stress Biology)
email:三木大介
DOI: 10.7875/first.author.2014.108
Regulation of active DNA demethylation by an α-crystallin domain protein in Arabidopsis.
Weiqiang Qian, Daisuke Miki, Mingguang Lei, Xiaohong Zhu, Huiming Zhang, Yunhua Liu, Yan Li, Zhaobo Lang, Jing Wang, Kai Tang, Renyi Liu, Jian-Kang Zhu
Molecular Cell, 55, 361-371 (2014)
DNAのメチル化はヒストンの修飾とともに動物および植物において遺伝子の発現におけるエピジェネティックな制御機構として知られている.細胞においてDNAメチル化の状態を正確に制御するためには,DNAのメチル化と脱メチル化の両方のバランスがうまくとれていなければならない.DNAメチル化機構についてはさまざまな知見が蓄積されつつあるが,相反するDNA脱メチル化機構についてはあまり研究が進んでいない.この研究では,シロイヌナズナを用いた遺伝学的な手法により,DNA脱メチル化に関与する遺伝子としてIDM2遺伝子を同定し,その分子機構について解析した.IDM2遺伝子は低分子熱ショックタンパク質ファミリーに属するαクリスタリンドメインをもつタンパク質をコードしており,筆者らが以前に報告した,IDM1によるヒストンH3のLys18のアセチル化を制御することにより,DNA脱メチル化の領域特異性を決定していることが明らかになった.
エピジェネティックな制御機構はさまざまな生物種において報告されている.なかでも,DNAのメチル化は重要なもののひとつであり,真核生物の発生および分化において重要な役割を担っている1,2).また,DNAのメチル化とヒストンの修飾とが密接な関係にあることも明らかにされつつある.動物ではその大部分がCG配列におけるCのメチル化あるのに対し,植物ではCG配列,CHG配列,CHH配列(H:A,T,G)におけるCのメチル化という3つのタイプがあり,それぞれ異なる分子機構により制御されている.全ゲノムDNAメチル化シークエンスの結果,遺伝子をコードしている領域においてはおもにCG配列が,トランスポゾンやくり返し配列などのヘテロクロマチン領域ではCHG配列およびCHH配列が,高くDNAメチル化されている3).植物のMET1は動物のDNMT1の相同タンパク質でありCG配列の維持DNAメチル化にはたらく.CMT3は植物に特有のタンパク質でCHG配列の維持DNAメチル化においてヒストンH3のLys9のジメチル化と連携している2).動物のDNMT3の機能的な相同タンパク質であるDRM2および植物に特有のCMT2は3つすべての配列の新規DNAメチル化に関与している4).このように,DNAメチル化の分子機構については研究が進み多くが明らかになりつつあるが,DNA脱メチル化の分子機構についての報告は少ない.
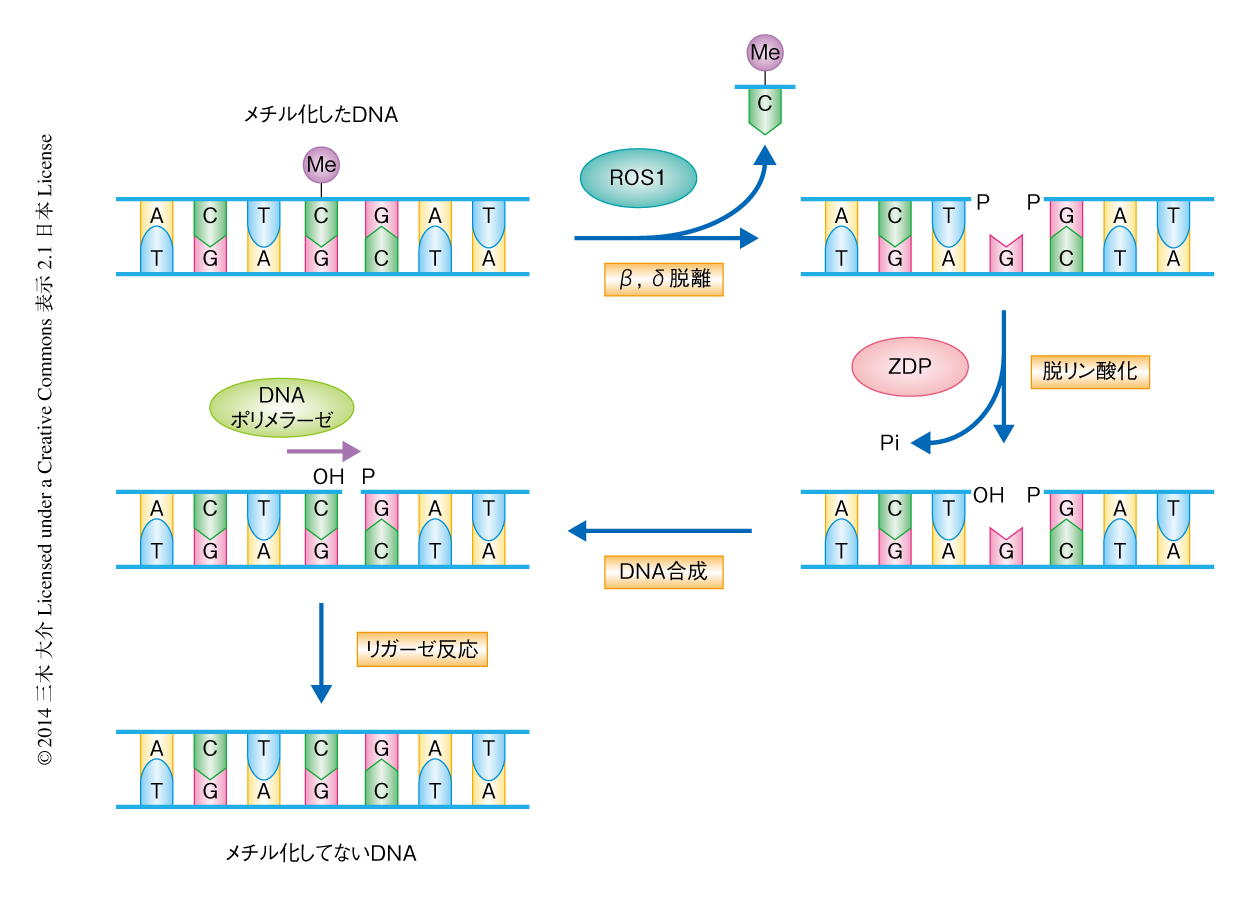
2002年に世界ではじめて,DNA脱メチル化において中心的な役割を担うDNAグリコシラーゼとして,ROS1がシロイヌナズナを用いた研究において発見された5).ROS1はメチル化したシトシン残基をDNA鎖から除去する.そののち,塩基除去修復経路により,メチル化されていないシトシン残基がそのギャップをうめDNA脱メチル化が完了する(図1).この一連の流れは動物におけるTET1を介したDNA脱メチル化経路と非常に類似しており,これらの機構が生物種をこえて高く保存されていることがわかる6).シロイヌナズナのros1変異体を用いた解析から,ゲノムにおいて約5000の領域がROS1によりDNA脱メチル化されていることが明らかになった7).しかし,これらの領域がどのように決定されているのか,その分子機構は謎のままであった.今回,新たに見い出されたIDM2と,以前に報告されたIDM1 7) とが協働し,とくに遺伝子をコードする領域においてDNA脱メチル化の配列特異性を決定していることが明らかになった.
シロイヌナズナのros1変異体において高度にDNAメチル化されている領域を用いて,内在性のChop-PCRマーカーを作製した7,8).これは,DNAメチル化に感受性の制限酵素により消化したゲノムDNA断片を鋳型としてPCRを行うと,ros1変異体では高度にDNAメチル化されているためバンドが検出されるが,野生型ではDNAメチル化が低いためバンドが検出されないというものである.このChop-PCRマーカーを用いてros1変異体のスクリーニングを行った結果,IDM1遺伝子およびIDM2遺伝子が同定された.ROS1によりDNA脱メチル化されている領域をマーカーにしたことより,これらの遺伝子は同じDNA脱メチル化経路においてはたらいていると推測された.
idm2変異体の全ゲノムDNAメチル化シークエンスを行ったところ,1218個の対立遺伝子特異的メチル化領域(differentially methylated region:DMR)が検出された.これらのうち約80%が遺伝子領域にあり,これはidm1変異体の場合と非常に類似していた.それに対して,ros1変異体においては約5000個の対立遺伝子特異的メチル化領域が検出されたが,その50%はトランスポゾン領域と重なっていた.さらに,idm2変異体とros1変異体の対立遺伝子特異的メチル化領域は約50%が重なっていたことからも,遺伝子領域におけるDNA脱メチル化にはIDM2遺伝子とROS1遺伝子とが共役して機能している可能性が高いことが考えられた.
idm2変異体の対立遺伝子特異的メチル化領域のなかからランダムに6つを選び,個別にバイサルファイトDNAメチル化解析を行った.選んだ6つのなかにはidm2変異体に特異的でidm1変異体およびros1変異体にはないものがあったにもかかわらず,idm1変異体およびros1変異体においては同じレベルのDNAメチル化の上昇が検出された.さらに,idm1 idm2二重変異体およびros1 idm2二重変異体においてDNAメチル化の相加的な上昇はみられなかったことから,IDM1遺伝子,IDM2遺伝子,ROS1遺伝子は同じDNA脱メチル化経路においてはたらいていることが強く示唆された.
遺伝学的な解析によりidm2変異体の原因遺伝子を特定したところ,植物において高く保存されているsHsp20と同じく,低分子熱ショックタンパク質ファミリーに属するαクリスタリンドメインをもつタンパク質をコードしていることが明らかになった.多くの低分子熱ショックタンパク質は熱ストレスにより発現が誘導されるが,IDM2遺伝子の発現は熱により誘導されなかった.さらに,通常,sHsp20は葉緑体やミトコンドリアに局在するが,IDM2は核への局在が観察された.
IDM2のもつαクリスタリンドメインに変異を導入したトランス遺伝子をidm2変異体に導入しその機能が回復するかどうか解析したところ,タンパク質の発現は検出されたにもかかわらずDNA脱メチル化は認められなかった.このことから,IDM2のαクリスタリンドメインは機能的なDNA脱メチル化に必須であることが示された.
酵母ツーハイブリッド法,スプリットルシフェラーゼ法,免疫共沈降法の結果,IDM2とIDM1とは直接に結合することが明らかになった.さらに,免疫染色を行ったところ,IDM2とIDM1は核においてドット状に共局在していた.筆者らは以前に,IDM1はROS1と共局在することを報告していた.よって,IDM2とROS1についても解析した結果,核において部分的な共局在が観察された.
筆者らは以前に,DNA脱メチル化に関与するタンパク質としてIDM1を報告した.IDM1は遺伝子領域において,ヒストンH3のLys4およびArg2がメチル化されていない領域のDNAメチル化を認識し,その領域のヒストンH3のLys18およびLys23をアセチル化する.これらのエピジェネティックな標識によりROS1をこの領域によび込みDNA脱メチル化を行う.つまり,IDM1はDNA脱メチル化の配列特異性を決定するタンパク質である.idm1変異体では対立遺伝子特異的メチル化領域においてヒストンH3のLys18およびLys23のアセチル化が低下していた.そこで,idm2変異体をクロマチン免疫沈降法により解析したところ,ヒストンH3のLys18のアセチル化は低下していたが,Lys23のアセチル化には変化が認められなかった.この結果は,IDM1の機能にIDM2が必須であることを示唆した(図2).
この研究では,IDM1がその機能を十分に発揮するためにはIDM2が必須であることが示された.IDM2はそのαクリスタリンドメインを介してIDM1と直接に結合していたことから,IDM2は植物においてIDM1の分子シャペロンとして機能する可能性が考えられた.しかし,IDM2の分子シャペロン活性を検出することはできなかったため,この結合がほかの意味をもつ可能性も捨てきれない.また,IDM1によりアセチル化されたヒストンH3のLys18およびLys23を,ROS1がどのように認識しDNA脱メチル化を行うのかも明らかになっていない7).今後,さらに精力的にDNA脱メチル化経路を解明していきたい.
略歴:2004年 奈良先端科学技術大学院大学バイオサイエンス研究科博士課程 修了,2012年より中国Shanghai Institutes for Biological Sciencesにてjunior group leader.
研究テーマ:植物のエピゲノム制御機構.
関心事:DNA脱メチル化.日中関係.
© 2014 三木 大介 Licensed under CC 表示 2.1 日本
(中国Shanghai Institutes for Biological Sciences,Shanghai Center for Plant Stress Biology)
email:三木大介
DOI: 10.7875/first.author.2014.108
Regulation of active DNA demethylation by an α-crystallin domain protein in Arabidopsis.
Weiqiang Qian, Daisuke Miki, Mingguang Lei, Xiaohong Zhu, Huiming Zhang, Yunhua Liu, Yan Li, Zhaobo Lang, Jing Wang, Kai Tang, Renyi Liu, Jian-Kang Zhu
Molecular Cell, 55, 361-371 (2014)
要 約
DNAのメチル化はヒストンの修飾とともに動物および植物において遺伝子の発現におけるエピジェネティックな制御機構として知られている.細胞においてDNAメチル化の状態を正確に制御するためには,DNAのメチル化と脱メチル化の両方のバランスがうまくとれていなければならない.DNAメチル化機構についてはさまざまな知見が蓄積されつつあるが,相反するDNA脱メチル化機構についてはあまり研究が進んでいない.この研究では,シロイヌナズナを用いた遺伝学的な手法により,DNA脱メチル化に関与する遺伝子としてIDM2遺伝子を同定し,その分子機構について解析した.IDM2遺伝子は低分子熱ショックタンパク質ファミリーに属するαクリスタリンドメインをもつタンパク質をコードしており,筆者らが以前に報告した,IDM1によるヒストンH3のLys18のアセチル化を制御することにより,DNA脱メチル化の領域特異性を決定していることが明らかになった.
はじめに
エピジェネティックな制御機構はさまざまな生物種において報告されている.なかでも,DNAのメチル化は重要なもののひとつであり,真核生物の発生および分化において重要な役割を担っている1,2).また,DNAのメチル化とヒストンの修飾とが密接な関係にあることも明らかにされつつある.動物ではその大部分がCG配列におけるCのメチル化あるのに対し,植物ではCG配列,CHG配列,CHH配列(H:A,T,G)におけるCのメチル化という3つのタイプがあり,それぞれ異なる分子機構により制御されている.全ゲノムDNAメチル化シークエンスの結果,遺伝子をコードしている領域においてはおもにCG配列が,トランスポゾンやくり返し配列などのヘテロクロマチン領域ではCHG配列およびCHH配列が,高くDNAメチル化されている3).植物のMET1は動物のDNMT1の相同タンパク質でありCG配列の維持DNAメチル化にはたらく.CMT3は植物に特有のタンパク質でCHG配列の維持DNAメチル化においてヒストンH3のLys9のジメチル化と連携している2).動物のDNMT3の機能的な相同タンパク質であるDRM2および植物に特有のCMT2は3つすべての配列の新規DNAメチル化に関与している4).このように,DNAメチル化の分子機構については研究が進み多くが明らかになりつつあるが,DNA脱メチル化の分子機構についての報告は少ない.
2002年に世界ではじめて,DNA脱メチル化において中心的な役割を担うDNAグリコシラーゼとして,ROS1がシロイヌナズナを用いた研究において発見された5).ROS1はメチル化したシトシン残基をDNA鎖から除去する.そののち,塩基除去修復経路により,メチル化されていないシトシン残基がそのギャップをうめDNA脱メチル化が完了する(図1).この一連の流れは動物におけるTET1を介したDNA脱メチル化経路と非常に類似しており,これらの機構が生物種をこえて高く保存されていることがわかる6).シロイヌナズナのros1変異体を用いた解析から,ゲノムにおいて約5000の領域がROS1によりDNA脱メチル化されていることが明らかになった7).しかし,これらの領域がどのように決定されているのか,その分子機構は謎のままであった.今回,新たに見い出されたIDM2と,以前に報告されたIDM1 7) とが協働し,とくに遺伝子をコードする領域においてDNA脱メチル化の配列特異性を決定していることが明らかになった.
1.IDM2遺伝子の同定
シロイヌナズナのros1変異体において高度にDNAメチル化されている領域を用いて,内在性のChop-PCRマーカーを作製した7,8).これは,DNAメチル化に感受性の制限酵素により消化したゲノムDNA断片を鋳型としてPCRを行うと,ros1変異体では高度にDNAメチル化されているためバンドが検出されるが,野生型ではDNAメチル化が低いためバンドが検出されないというものである.このChop-PCRマーカーを用いてros1変異体のスクリーニングを行った結果,IDM1遺伝子およびIDM2遺伝子が同定された.ROS1によりDNA脱メチル化されている領域をマーカーにしたことより,これらの遺伝子は同じDNA脱メチル化経路においてはたらいていると推測された.
2.IDM2遺伝子は多くの領域においてDNA脱メチル化にはたらく
idm2変異体の全ゲノムDNAメチル化シークエンスを行ったところ,1218個の対立遺伝子特異的メチル化領域(differentially methylated region:DMR)が検出された.これらのうち約80%が遺伝子領域にあり,これはidm1変異体の場合と非常に類似していた.それに対して,ros1変異体においては約5000個の対立遺伝子特異的メチル化領域が検出されたが,その50%はトランスポゾン領域と重なっていた.さらに,idm2変異体とros1変異体の対立遺伝子特異的メチル化領域は約50%が重なっていたことからも,遺伝子領域におけるDNA脱メチル化にはIDM2遺伝子とROS1遺伝子とが共役して機能している可能性が高いことが考えられた.
idm2変異体の対立遺伝子特異的メチル化領域のなかからランダムに6つを選び,個別にバイサルファイトDNAメチル化解析を行った.選んだ6つのなかにはidm2変異体に特異的でidm1変異体およびros1変異体にはないものがあったにもかかわらず,idm1変異体およびros1変異体においては同じレベルのDNAメチル化の上昇が検出された.さらに,idm1 idm2二重変異体およびros1 idm2二重変異体においてDNAメチル化の相加的な上昇はみられなかったことから,IDM1遺伝子,IDM2遺伝子,ROS1遺伝子は同じDNA脱メチル化経路においてはたらいていることが強く示唆された.
3.IDM2遺伝子はαクリスタリンドメインタンパク質をコードする
遺伝学的な解析によりidm2変異体の原因遺伝子を特定したところ,植物において高く保存されているsHsp20と同じく,低分子熱ショックタンパク質ファミリーに属するαクリスタリンドメインをもつタンパク質をコードしていることが明らかになった.多くの低分子熱ショックタンパク質は熱ストレスにより発現が誘導されるが,IDM2遺伝子の発現は熱により誘導されなかった.さらに,通常,sHsp20は葉緑体やミトコンドリアに局在するが,IDM2は核への局在が観察された.
IDM2のもつαクリスタリンドメインに変異を導入したトランス遺伝子をidm2変異体に導入しその機能が回復するかどうか解析したところ,タンパク質の発現は検出されたにもかかわらずDNA脱メチル化は認められなかった.このことから,IDM2のαクリスタリンドメインは機能的なDNA脱メチル化に必須であることが示された.
4.IDM2とIDM1とは結合し協同してはたらく
酵母ツーハイブリッド法,スプリットルシフェラーゼ法,免疫共沈降法の結果,IDM2とIDM1とは直接に結合することが明らかになった.さらに,免疫染色を行ったところ,IDM2とIDM1は核においてドット状に共局在していた.筆者らは以前に,IDM1はROS1と共局在することを報告していた.よって,IDM2とROS1についても解析した結果,核において部分的な共局在が観察された.
5.IDM2は細胞におけるIDM1の機能に対し重要な役割を担う
筆者らは以前に,DNA脱メチル化に関与するタンパク質としてIDM1を報告した.IDM1は遺伝子領域において,ヒストンH3のLys4およびArg2がメチル化されていない領域のDNAメチル化を認識し,その領域のヒストンH3のLys18およびLys23をアセチル化する.これらのエピジェネティックな標識によりROS1をこの領域によび込みDNA脱メチル化を行う.つまり,IDM1はDNA脱メチル化の配列特異性を決定するタンパク質である.idm1変異体では対立遺伝子特異的メチル化領域においてヒストンH3のLys18およびLys23のアセチル化が低下していた.そこで,idm2変異体をクロマチン免疫沈降法により解析したところ,ヒストンH3のLys18のアセチル化は低下していたが,Lys23のアセチル化には変化が認められなかった.この結果は,IDM1の機能にIDM2が必須であることを示唆した(図2).
おわりに
この研究では,IDM1がその機能を十分に発揮するためにはIDM2が必須であることが示された.IDM2はそのαクリスタリンドメインを介してIDM1と直接に結合していたことから,IDM2は植物においてIDM1の分子シャペロンとして機能する可能性が考えられた.しかし,IDM2の分子シャペロン活性を検出することはできなかったため,この結合がほかの意味をもつ可能性も捨てきれない.また,IDM1によりアセチル化されたヒストンH3のLys18およびLys23を,ROS1がどのように認識しDNA脱メチル化を行うのかも明らかになっていない7).今後,さらに精力的にDNA脱メチル化経路を解明していきたい.
文 献
- He, X. J., Chen, T. & Zhu, J. K.: Regulation and function of DNA methylation in plants and animals. Cell Res., 21, 442-465 (2011)[PubMed]
- Law, J. A. & Jacobsen, S. E.: Establishing, maintaining and modifying DNA methylation patterns in plants and animals. Nat. Rev. Genet., 11, 204-220 (2010)[PubMed]
- Zhang, X., Yazaki, J., Sundaresan, A. et al.: Genome-wide high-resolution mapping and functional analysis of DNA methylation in Arabidopsis. Cell, 126, 1189-1201 (2006)[PubMed]
- Zemach, A., Kim, M. Y., Hsieh, P. H. et al.: The Arabidopsis nucleosome remodeler DDM1 allows DNA methyltransferases to access H1-containing heterochromatin. Cell, 153, 193-205 (2013)[PubMed]
- Gong, Z., Morales-Ruiz, T., Ariza, R. R. et al.: ROS1, a repressor of transcriptional gene silencing in Arabidopsis, encodes a DNA glycosylase/lyase. Cell, 111, 803-814 (2002)[PubMed]
- Gong, Z. & Zhu, J. K.: Active DNA demethylation by oxidation and repair. Cell Res., 21, 1649-1651 (2011)[PubMed]
- Qian, W., Miki, D., Zhang, H. et al.: A histone acetyltransferase regulates active DNA demethylation in Arabidopsis. Science, 336, 1445-1448 (2012)[PubMed]
- Martinez-Macias, M. I., Qian, W., Miki, D. et al.: A DNA 3’ phosphatase functions in active DNA demethylation in Arabidopsis. Mol. Cell, 45, 357-370 (2012)[PubMed]
著者プロフィール
略歴:2004年 奈良先端科学技術大学院大学バイオサイエンス研究科博士課程 修了,2012年より中国Shanghai Institutes for Biological Sciencesにてjunior group leader.
研究テーマ:植物のエピゲノム制御機構.
関心事:DNA脱メチル化.日中関係.
© 2014 三木 大介 Licensed under CC 表示 2.1 日本