オートファジー始動複合体の形成の構造基盤
藤岡優子・野田展生
(微生物化学研究所 分子構造解析部)
email:藤岡優子,野田展生
DOI: 10.7875/first.author.2014.067
Structural basis of starvation-induced assembly of the autophagy initiation complex.
Yuko Fujioka, Sho W. Suzuki, Hayashi Yamamoto, Chika Kondo-Kakuta, Yayoi Kimura, Hisashi Hirano, Rinji Akada, Fuyuhiko Inagaki, Yoshinori Ohsumi, Nobuo N. Noda
Nature Structural & Molecular Biology, 21, 513-521 (2014)
出芽酵母においてはPASとよばれる構造体の形成がオートファジー始動の最初のステップである.PASの形成にはAtg13が飢餓に依存して脱リン酸化され,Atg1,Atg13,Atg17,Atg29,Atg31からなるAtg1複合体が形成されることが重要であるが,これまで,その詳細な分子機構は明らかにされていなかった.筆者らは,Atg13においてAtg1およびAtg17との結合領域を同定し,小型化したAtg1-Atg13複合体およびAtg13-Atg17-Atg29-Atg31複合体のX線結晶構造解析を行った.その結果,Atg13はMIMと名づけた領域を用いてAtg1のC末端側にあるMITドメインの2つ並んだ球状構造と結合すること,一方,Atg13は17BRと名づけた短い領域を用いてAtg17の疎水性ポケットと結合することが明らかになった.これらAtg1およびAtg17との相互作用は,Atg13のもつ特定のSerが飢餓に依存して脱リン酸化されることにより増強されていた.以上の知見から,飢餓によるAtg13の脱リン酸化がAtg1およびAtg17との結合をとおしてPASの形成をひき起こし,その結果,オートファジーが始動するというモデルが考えられた.
オートファジーは真核生物に保存された細胞における分解系である.その分解の対象は多岐にわたり,タンパク質やオルガネラのほか,細胞に侵入した病原体までも分解することができる1).オートファジーが誘導されると,突如として隔離膜とよばれるおわん状の膜構造が出現し,分解の対象を包み込みながら伸展する.隔離膜の端がつながると二重膜により包まれたオートファゴソームが形成される.つづいて,オートファゴソームの外膜が液胞あるいはリソソームと融合し,オートファゴソームの内膜と内容物が消化酵素により分解される.出芽酵母ではオートファゴソームの形成の過程において18ものオートファジー関連タンパク質(Atgタンパク質)が必須であることがわかっている2)(出芽酵母におけるオートファジーの研究については,荒木保弘・大隅良典, 領域融合レビュー, 1, e005, 2012 も参照されたい).これらAtgタンパク質は液胞の近傍にあるPAS(pre-autophagosomal structure,プレオートオートファゴソーム構造体)とよばれる動的な構造体に局在する3).隔離膜はPASから形成されるので,PASの構造および機能を明らかにすることはオートファゴソームの形成の分子機構を知るうえで必要不可欠である.
個々のタンパク質に焦点を移すと,オートファジーは飢餓により強く誘導されるが,実際に栄養状態を感知するのはTOR複合体1であり,そのシグナルはAtg13にリン酸化というかたちで伝達される4,5).Atg13は富栄養条件においてはTOR複合体1により高度にリン酸化されているが,飢餓条件においてTOR複合体1の活性が阻害されるとAtg13はすみやかに脱リン酸化される5).脱リン酸化したAtg13はAtgタンパク質のうち唯一のキナーゼであるAtg1と結合し,さらに,Atg17-Atg29-Atg31複合体が結合することによりPASの足場となる複合体であるAtg1複合体(Atg1-Atg13-Atg17-Atg29-Atg31複合体,哺乳類では,ULK1複合体)を構築する6).これらの相互作用はAtg1のもつキナーゼ活性を上昇させるが,その上昇はオートファジーの始動において必須であることがわかっている5).しかしながら,これまで,Atg13を介したAtg1複合体の形成の構造基盤は明らかにされておらず,これらのタンパク質がどのような相互作用により結合し,それらがリン酸化によりどのように制御されているのかは不明であった.
今回,筆者らは,Atg1-Atg13複合体およびAtg13-Atg17-Atg29-Atg31複合体のX線結晶構造解析を行った.また,飢餓に依存して脱リン酸化されるAtg13の残基を質量分析法により網羅的に同定した.得られた立体構造およびリン酸化部位の情報にもとづきin vivoおよびin vitroにおける変異解析を行い,Atg13の脱リン酸化がどのようにAtg1およびAtg17との結合を制御し,PASの構築をひき起こすのかを明らかにした.
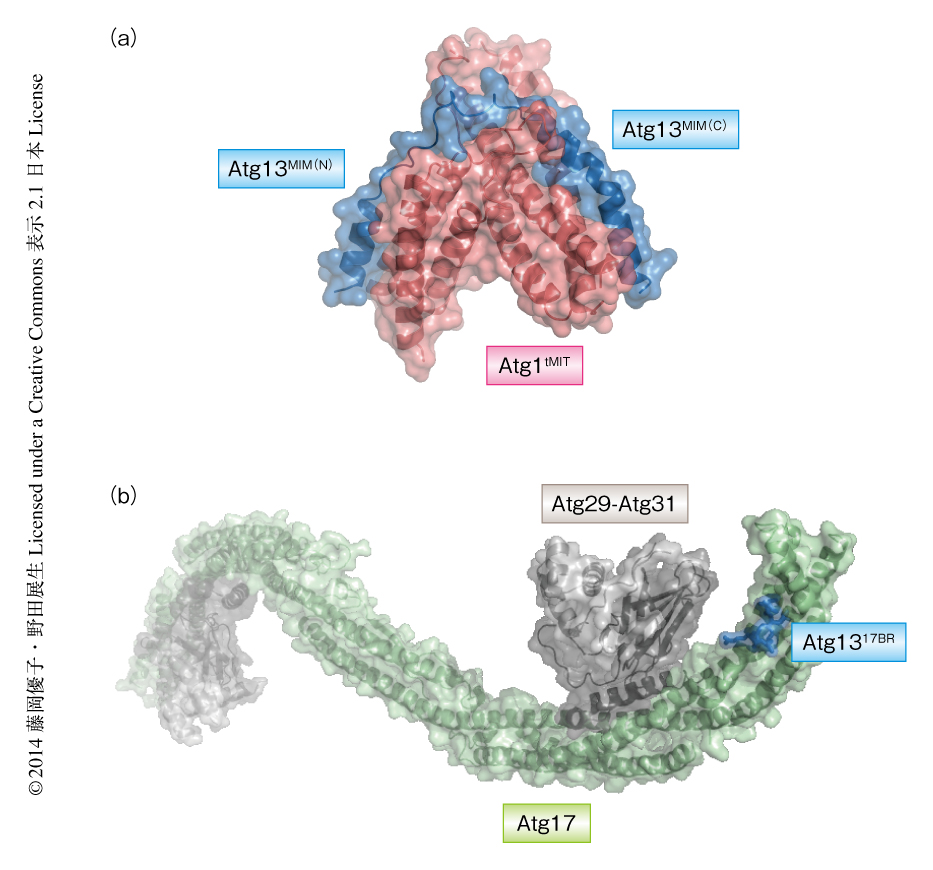
Atg13はN末端にHORMAドメインをもつが7),それ以外の約450残基は天然変性領域と予測される.Atg1との結合領域を同定した結果,天然変性領域にある62残基(残基番号460~521,このレビューにおける残基番号は,すべて出芽酵母の場合)がAtg1との結合において必要十分であった.Atg1はN末端側にキナーゼドメインをもち,C末端側には保存されたAtg13結合領域が存在する.耐熱性酵母Kluyveromyces marxianusに由来するAtg1のAtg13結合領域とAtg13のAtg1結合領域を共結晶化し,この小型化したAtg1-Atg13複合体の結晶構造を分解能2.2Åで決定した(PDB ID:4P1N,図1a).Atg1は3本のαヘリックスからなるMITドメインがタンデムに2つ並び(N末端側から,MIT1およびMIT2と命名した),それらは互いに密接に相互作用することによりひとつの球状構造をとっていた.一方,Atg13は2本のαヘリックスがループでつながれた伸びた構造をとっており,N末端側のαヘリックスはMIT2と,C末端側のαヘリックスはMIT1と結合していた.Atg1とAtg13の結合の様式は,ESCRTタンパク質などにおけるタンパク質間相互作用に用いられるMITドメインとMIM領域との結合の様式と類似していた.そこで,Atg13のAtg1結合領域をAtg13MIM,また,Atg13MIMのN末端側をAtg13MIM(N),C末端側をAtg13MIM(C) とよぶ.一方,Atg1のAtg13結合領域はAtg1tMITとよぶ.Atg1tMITとAtg13MIMの相互作用はおもに疎水性相互作用であり,MIT1あるいはMIT2それぞれの表面にできた疎水性の溝にそいAtg13MIMのもつ2つのαヘリックスが結合していた.Atg1tMITはAtg1のヒトのホモログであるULK1にも保存されているが,ULK1の2つのMITドメインはヒトのAtg13の天然変性領域にある約60残基の領域と安定な複合体を形成することが確認された.このことから,Atg1tMITとAtg13MIMとの相互作用の様式が進化において保存されていることが示唆された.
得られたAtg1tMIT-Atg13MIM複合体の結晶構造をもとに変異体の解析を行った.等温滴定型熱量測定により解離定数を求めたところ,Atg13MIM(N) はAtg1tMITに対し解離定数2.5μMの結合を示したのに対し,Atg13MIM(C) は同じ条件では結合を示さなかった.つまり,Atg1tMITとAtg13MIMとの相互作用はおもにAtg13MIM(N) により担われていることが明らかになった.しかし,Atg13MIMとAtg1tMITとでは解離定数0.36μMというより強い結合を示したことから,Atg13MIM(C) が補助的にはたらくことでAtg13MIM(N) とAtg1tMITとの結合を強固なものにしていると考えられた.また,結晶構造においてAtg1tMITの疎水性のポケットにつきささっていたAtg13MIM(N) のPhe468あるいはLeu472をAlaに置換した変異体では結合はみられなかった.この変異体を発現した株では,Atg13MIM欠損変異体を発現した株と同様に,飢餓条件においてもPASの形成が起こらず,オートファジー活性も示さなかった.また,Atg1の活性化の指標であるAtg1の自己リン酸化もこの変異体を発現した株では起こらなかった.これらのことから,得られたAtg1tMIT-Atg13MIM複合体の結晶構造は妥当なものであり,Atg1tMITとAtg13MIMとの結合がオートファジーにおいて必須であることが明らかになった.
Atg13におけるAtg17結合領域を探索した結果,Atg13の天然変性領域に存在するわずか13残基の領域(残基番号424~436)がAtg17との結合に十分であることがわかった.この領域を17BRと名づけ,Atg13の17BRをAtg1317BRとよぶ.最近,報告されたLachancea thermotoleransに由来するAtg17-Atg29-Atg31複合体の結晶構造8)(PDB ID:4HPQ)を参考にして,それにL. thermotoleransに由来するAtg1317BRを共結晶化し,Atg1317BR-Atg17-Atg29-Atg31複合体の結晶構造を分解能3.2Åで決定した(PDB ID:4P1W,図1b).Atg1317BRはAtg17のN末端の付近にある疎水性ポケットに2つの疎水性残基Phe430およびIle433をつきさして結合しており,さらに,Ser428およびSer429によりAtg17のAsp247と水素結合を形成していた.
得られたAtg1317BR-Atg17-Atg29-Atg31複合体の結晶構造をもとに変異体の解析を行った.等温滴定型熱量測定の結果,野生型Atg17とAtg1317BRとの解離定数は1.2μMという値を示したが,Asp247をAlaに置換したAtg17変異体ではAtg1317BRとの結合は観測されなかった.相互作用にかかわるほかの残基の重要性についても,免疫沈降実験により確認した.また,Asp247をAlaに置換したAtg17変異体を発現した株はPASの形成およびオートファジー活性を示さなかった.以上の結果から,得られたAtg1317BR-Atg17-Atg29-Atg31複合体の結晶構造は妥当なものであり,Atg1317BRとAtg17との結合がPASの形成およびオートファジーにおいて必須であることが明らかになった.
Atg1tMITとAtg13MIMとの結合がどのように制御されているのかを明らかにするため,質量分析法によりAtg13のリン酸化の状態を調べた.TOR複合体1の阻害剤であるラパマイシンを処理した出芽酵母から精製したAtg13と未処理の出芽酵母から精製したAtg13とを比べることにより,ラパマイシンの処理によりリン酸化のレベルが低下する51の残基を同定した.これらはAtg13MIM(N) には含まれずAtg13MIM(C) に含まれていたことから,Atg13MIM(C) に存在する5残基のSerをAspに置換した変異体を作製した.免疫沈降実験において,この変異体はラパマイシンを処理してもAtg1との結合はほとんどみられなくなった.等温滴定型熱量測定により解離定数を求めたところ,Atg1tMITと5残基のSerをAspに置換したAtg13MIM変異体との解離定数は1.2μMという値を示した.この値はAtg1tMITと野生型Atg13MIMとの解離定数の約3倍であったことから,Atg13MIM(C) はAtg13MIM(N) との結合をリン酸化により十分には増強できず,その結果,生体におけるAtg1-Atg13複合体の量は大きく減少すると考えられた.5残基のSerをAspに置換したAtg13変異体を発現した株では,野生型Atg13を発現した株に比べオートファジー活性が顕著に低下していたが,そのレベルはAtg13MIM(C) 欠損変異体を発現した株と同じ程度であった.
Atg13とAtg17との結合もAtg1とAtg13との結合と同様に,飢餓により増加するので,Atg13のリン酸化が制御の鍵をにぎっていると考えられた.そこで,さきの質量分析法による結果においてAtg1317BRに注目したところ,Ser428およびSer429が含まれていた.さきに述べたように,Atg13のSer428およびSer429はAtg17のAsp247と水素結合を形成しており,Atg1317BRとAtg17との結合に必須であった.このSerがリン酸化されることはAsp247との水素結合を破壊するだけでなく,電荷的な反発もひき起こす.実際に,Ser429をAspに置換したAtg13変異体はAtg17との結合を示さなかった.また,この変異体を発現した株ではPASの形成がまったくみられず,オートファジー活性もほとんど示さなかった.
リン酸化Ser428およびリン酸化Ser429を特異的に認識する抗体を作製し,生体におけるこれらSerのリン酸化の状態を確認したところ,富栄養条件においてはリン酸化が検出されたが,ラパマイシンを処理したとき,あるいは,飢餓条件においてリン酸化はほとんどみられなくなった.すなわち,Atg13のSer428およびSer429は富栄養条件においてリン酸化されており,飢餓により脱リン酸化することが示された.以上のことから,Atg13とAtg17との結合はAtg13のSer428およびSer429のリン酸化により負に制御されていることが明らかになった.
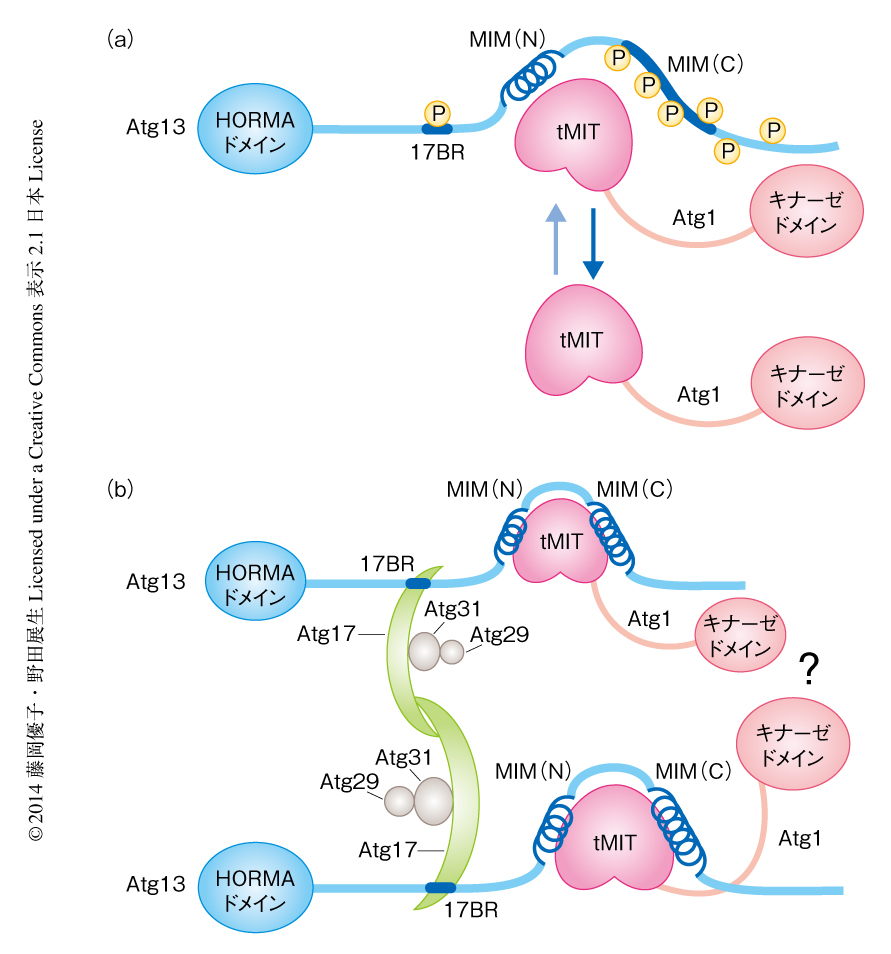
今回,明らかにされた結晶構造や,それにもとづくin vitroおよびin vivoにおける解析の結果を要約したモデルはつぎのようなものである(図2).富栄養条件においてAtg13はTOR複合体1により高度にリン酸化されている.Atg1との結合領域は2つの領域に分かれており,Atg13MIM(N) がAtg1との結合の主要な部分を担っており,Atg13MIM(C) はAtg1との結合の制御にかかわっている.Atg13MIM(N) だけでもAtg1と結合できるが,Atg13MIM(C) が脱リン酸化されてAtg1との結合にくわわると,Atg1とAtg13との親和性は増強され,結果として,細胞におけるAtg1-Atg13複合体の割合が増加する.Atg13とAtg17との結合もリン酸化により制御されている.ただし,この場合は結合に直接的にかかわるAtg13のもつSerそれ自体がリン酸化により制御されており,このSerがリン酸化されると電荷的な反発によりAtg17と結合できなくなる.飢餓条件においてAtg13が脱リン酸化されると,Atg13がAtg1およびAtg17と強く結合することによりAtg1複合体(Atg1-Atg13-Atg17-Atg29-Atg31複合体)が完成し,それがPASの形成の足場となりオートファジーが始動する.
今回の論文では,出芽酵母におけるAtg1複合体の形成機構を解明したが,哺乳類ではULK1,Atg13,FIP200,Atg101のつくるULK1複合体がオートファジーの始動について同様の役割をはたすことが知られている9).ULK1にはAtg1のMITドメインが保存されていることから,Atg13との相互作用はMITドメインとMIM領域との相互作用と考えられる.しかし,出芽酵母においてAtg1との結合の制御にはたらいているAtg13MIM(C) のもつSerは哺乳類のAtg13において保存されておらず,実際に,ULK1複合体は栄養状態にかかわらずつねにAtg13と結合していると報告されている.一方,Atg17とFIP200との配列相同性は低いが,Atg17と同様にFIP200もコイルドコイルタンパク質と予測されること,また,機能的にも類似していることから,FIP200はAtg17の機能ホモログと考えられている10).哺乳類の場合,FIP200もまた恒常的にULK1と複合体を形成していると報告されている10).しかしながら,出芽酵母におけるAtg13とAtg17との結合のリン酸化による厳密な制御を目のあたりにすると,哺乳類におけるAtg13とFIP200との結合もまたリン酸化により制御されていることが期待される.たとえば,ULK1複合体の状態を保ったまま,構成タンパク質のあいだの相互作用の組替えなどが脱リン酸化によりひき起こされる可能性も考えられる.
今回,明らかになった結晶構造にはAtg1のもつキナーゼドメインは含まれておらず,Atg1複合体の形成がAtg1のキナーゼ活性をどのように制御しているのかは未解明のままである.Atg1の活性化にはそれ自体の二量体化による自己リン酸化が重要であるとの報告があるので,Atg17が二量体として存在することが関係するというアイディアもうかぶが,全容の解明については,今後,行われるであろうキナーゼドメインを含んだ結晶構造の解析に期待したい.
略歴:2009年 北海道大学大学院薬学研究科にて博士号取得,同 研究員を経て,2011年より微生物化学研究所 博士研究員.
研究テーマ:オートファジーの構造生物学.
関心事:子供の成長,結晶の成長.
野田 展生(Nobuo N. Noda)
微生物化学研究所 主席研究員.
© 2014 藤岡優子・野田展生 Licensed under CC 表示 2.1 日本
(微生物化学研究所 分子構造解析部)
email:藤岡優子,野田展生
DOI: 10.7875/first.author.2014.067
Structural basis of starvation-induced assembly of the autophagy initiation complex.
Yuko Fujioka, Sho W. Suzuki, Hayashi Yamamoto, Chika Kondo-Kakuta, Yayoi Kimura, Hisashi Hirano, Rinji Akada, Fuyuhiko Inagaki, Yoshinori Ohsumi, Nobuo N. Noda
Nature Structural & Molecular Biology, 21, 513-521 (2014)
要 約
出芽酵母においてはPASとよばれる構造体の形成がオートファジー始動の最初のステップである.PASの形成にはAtg13が飢餓に依存して脱リン酸化され,Atg1,Atg13,Atg17,Atg29,Atg31からなるAtg1複合体が形成されることが重要であるが,これまで,その詳細な分子機構は明らかにされていなかった.筆者らは,Atg13においてAtg1およびAtg17との結合領域を同定し,小型化したAtg1-Atg13複合体およびAtg13-Atg17-Atg29-Atg31複合体のX線結晶構造解析を行った.その結果,Atg13はMIMと名づけた領域を用いてAtg1のC末端側にあるMITドメインの2つ並んだ球状構造と結合すること,一方,Atg13は17BRと名づけた短い領域を用いてAtg17の疎水性ポケットと結合することが明らかになった.これらAtg1およびAtg17との相互作用は,Atg13のもつ特定のSerが飢餓に依存して脱リン酸化されることにより増強されていた.以上の知見から,飢餓によるAtg13の脱リン酸化がAtg1およびAtg17との結合をとおしてPASの形成をひき起こし,その結果,オートファジーが始動するというモデルが考えられた.
はじめに
オートファジーは真核生物に保存された細胞における分解系である.その分解の対象は多岐にわたり,タンパク質やオルガネラのほか,細胞に侵入した病原体までも分解することができる1).オートファジーが誘導されると,突如として隔離膜とよばれるおわん状の膜構造が出現し,分解の対象を包み込みながら伸展する.隔離膜の端がつながると二重膜により包まれたオートファゴソームが形成される.つづいて,オートファゴソームの外膜が液胞あるいはリソソームと融合し,オートファゴソームの内膜と内容物が消化酵素により分解される.出芽酵母ではオートファゴソームの形成の過程において18ものオートファジー関連タンパク質(Atgタンパク質)が必須であることがわかっている2)(出芽酵母におけるオートファジーの研究については,荒木保弘・大隅良典, 領域融合レビュー, 1, e005, 2012 も参照されたい).これらAtgタンパク質は液胞の近傍にあるPAS(pre-autophagosomal structure,プレオートオートファゴソーム構造体)とよばれる動的な構造体に局在する3).隔離膜はPASから形成されるので,PASの構造および機能を明らかにすることはオートファゴソームの形成の分子機構を知るうえで必要不可欠である.
個々のタンパク質に焦点を移すと,オートファジーは飢餓により強く誘導されるが,実際に栄養状態を感知するのはTOR複合体1であり,そのシグナルはAtg13にリン酸化というかたちで伝達される4,5).Atg13は富栄養条件においてはTOR複合体1により高度にリン酸化されているが,飢餓条件においてTOR複合体1の活性が阻害されるとAtg13はすみやかに脱リン酸化される5).脱リン酸化したAtg13はAtgタンパク質のうち唯一のキナーゼであるAtg1と結合し,さらに,Atg17-Atg29-Atg31複合体が結合することによりPASの足場となる複合体であるAtg1複合体(Atg1-Atg13-Atg17-Atg29-Atg31複合体,哺乳類では,ULK1複合体)を構築する6).これらの相互作用はAtg1のもつキナーゼ活性を上昇させるが,その上昇はオートファジーの始動において必須であることがわかっている5).しかしながら,これまで,Atg13を介したAtg1複合体の形成の構造基盤は明らかにされておらず,これらのタンパク質がどのような相互作用により結合し,それらがリン酸化によりどのように制御されているのかは不明であった.
今回,筆者らは,Atg1-Atg13複合体およびAtg13-Atg17-Atg29-Atg31複合体のX線結晶構造解析を行った.また,飢餓に依存して脱リン酸化されるAtg13の残基を質量分析法により網羅的に同定した.得られた立体構造およびリン酸化部位の情報にもとづきin vivoおよびin vitroにおける変異解析を行い,Atg13の脱リン酸化がどのようにAtg1およびAtg17との結合を制御し,PASの構築をひき起こすのかを明らかにした.
1.Atg1とAtg13との結合の様式とオートファジーにおける役割
Atg13はN末端にHORMAドメインをもつが7),それ以外の約450残基は天然変性領域と予測される.Atg1との結合領域を同定した結果,天然変性領域にある62残基(残基番号460~521,このレビューにおける残基番号は,すべて出芽酵母の場合)がAtg1との結合において必要十分であった.Atg1はN末端側にキナーゼドメインをもち,C末端側には保存されたAtg13結合領域が存在する.耐熱性酵母Kluyveromyces marxianusに由来するAtg1のAtg13結合領域とAtg13のAtg1結合領域を共結晶化し,この小型化したAtg1-Atg13複合体の結晶構造を分解能2.2Åで決定した(PDB ID:4P1N,図1a).Atg1は3本のαヘリックスからなるMITドメインがタンデムに2つ並び(N末端側から,MIT1およびMIT2と命名した),それらは互いに密接に相互作用することによりひとつの球状構造をとっていた.一方,Atg13は2本のαヘリックスがループでつながれた伸びた構造をとっており,N末端側のαヘリックスはMIT2と,C末端側のαヘリックスはMIT1と結合していた.Atg1とAtg13の結合の様式は,ESCRTタンパク質などにおけるタンパク質間相互作用に用いられるMITドメインとMIM領域との結合の様式と類似していた.そこで,Atg13のAtg1結合領域をAtg13MIM,また,Atg13MIMのN末端側をAtg13MIM(N),C末端側をAtg13MIM(C) とよぶ.一方,Atg1のAtg13結合領域はAtg1tMITとよぶ.Atg1tMITとAtg13MIMの相互作用はおもに疎水性相互作用であり,MIT1あるいはMIT2それぞれの表面にできた疎水性の溝にそいAtg13MIMのもつ2つのαヘリックスが結合していた.Atg1tMITはAtg1のヒトのホモログであるULK1にも保存されているが,ULK1の2つのMITドメインはヒトのAtg13の天然変性領域にある約60残基の領域と安定な複合体を形成することが確認された.このことから,Atg1tMITとAtg13MIMとの相互作用の様式が進化において保存されていることが示唆された.
得られたAtg1tMIT-Atg13MIM複合体の結晶構造をもとに変異体の解析を行った.等温滴定型熱量測定により解離定数を求めたところ,Atg13MIM(N) はAtg1tMITに対し解離定数2.5μMの結合を示したのに対し,Atg13MIM(C) は同じ条件では結合を示さなかった.つまり,Atg1tMITとAtg13MIMとの相互作用はおもにAtg13MIM(N) により担われていることが明らかになった.しかし,Atg13MIMとAtg1tMITとでは解離定数0.36μMというより強い結合を示したことから,Atg13MIM(C) が補助的にはたらくことでAtg13MIM(N) とAtg1tMITとの結合を強固なものにしていると考えられた.また,結晶構造においてAtg1tMITの疎水性のポケットにつきささっていたAtg13MIM(N) のPhe468あるいはLeu472をAlaに置換した変異体では結合はみられなかった.この変異体を発現した株では,Atg13MIM欠損変異体を発現した株と同様に,飢餓条件においてもPASの形成が起こらず,オートファジー活性も示さなかった.また,Atg1の活性化の指標であるAtg1の自己リン酸化もこの変異体を発現した株では起こらなかった.これらのことから,得られたAtg1tMIT-Atg13MIM複合体の結晶構造は妥当なものであり,Atg1tMITとAtg13MIMとの結合がオートファジーにおいて必須であることが明らかになった.
2.Atg13とAtg17との結合の様式とオートファジーにおける役割
Atg13におけるAtg17結合領域を探索した結果,Atg13の天然変性領域に存在するわずか13残基の領域(残基番号424~436)がAtg17との結合に十分であることがわかった.この領域を17BRと名づけ,Atg13の17BRをAtg1317BRとよぶ.最近,報告されたLachancea thermotoleransに由来するAtg17-Atg29-Atg31複合体の結晶構造8)(PDB ID:4HPQ)を参考にして,それにL. thermotoleransに由来するAtg1317BRを共結晶化し,Atg1317BR-Atg17-Atg29-Atg31複合体の結晶構造を分解能3.2Åで決定した(PDB ID:4P1W,図1b).Atg1317BRはAtg17のN末端の付近にある疎水性ポケットに2つの疎水性残基Phe430およびIle433をつきさして結合しており,さらに,Ser428およびSer429によりAtg17のAsp247と水素結合を形成していた.
得られたAtg1317BR-Atg17-Atg29-Atg31複合体の結晶構造をもとに変異体の解析を行った.等温滴定型熱量測定の結果,野生型Atg17とAtg1317BRとの解離定数は1.2μMという値を示したが,Asp247をAlaに置換したAtg17変異体ではAtg1317BRとの結合は観測されなかった.相互作用にかかわるほかの残基の重要性についても,免疫沈降実験により確認した.また,Asp247をAlaに置換したAtg17変異体を発現した株はPASの形成およびオートファジー活性を示さなかった.以上の結果から,得られたAtg1317BR-Atg17-Atg29-Atg31複合体の結晶構造は妥当なものであり,Atg1317BRとAtg17との結合がPASの形成およびオートファジーにおいて必須であることが明らかになった.
3.Atg13MIM(C) のリン酸化がAtg1との結合を制御する
Atg1tMITとAtg13MIMとの結合がどのように制御されているのかを明らかにするため,質量分析法によりAtg13のリン酸化の状態を調べた.TOR複合体1の阻害剤であるラパマイシンを処理した出芽酵母から精製したAtg13と未処理の出芽酵母から精製したAtg13とを比べることにより,ラパマイシンの処理によりリン酸化のレベルが低下する51の残基を同定した.これらはAtg13MIM(N) には含まれずAtg13MIM(C) に含まれていたことから,Atg13MIM(C) に存在する5残基のSerをAspに置換した変異体を作製した.免疫沈降実験において,この変異体はラパマイシンを処理してもAtg1との結合はほとんどみられなくなった.等温滴定型熱量測定により解離定数を求めたところ,Atg1tMITと5残基のSerをAspに置換したAtg13MIM変異体との解離定数は1.2μMという値を示した.この値はAtg1tMITと野生型Atg13MIMとの解離定数の約3倍であったことから,Atg13MIM(C) はAtg13MIM(N) との結合をリン酸化により十分には増強できず,その結果,生体におけるAtg1-Atg13複合体の量は大きく減少すると考えられた.5残基のSerをAspに置換したAtg13変異体を発現した株では,野生型Atg13を発現した株に比べオートファジー活性が顕著に低下していたが,そのレベルはAtg13MIM(C) 欠損変異体を発現した株と同じ程度であった.
4.Atg1317BRの脱リン酸化がAtg17との結合を誘導する
Atg13とAtg17との結合もAtg1とAtg13との結合と同様に,飢餓により増加するので,Atg13のリン酸化が制御の鍵をにぎっていると考えられた.そこで,さきの質量分析法による結果においてAtg1317BRに注目したところ,Ser428およびSer429が含まれていた.さきに述べたように,Atg13のSer428およびSer429はAtg17のAsp247と水素結合を形成しており,Atg1317BRとAtg17との結合に必須であった.このSerがリン酸化されることはAsp247との水素結合を破壊するだけでなく,電荷的な反発もひき起こす.実際に,Ser429をAspに置換したAtg13変異体はAtg17との結合を示さなかった.また,この変異体を発現した株ではPASの形成がまったくみられず,オートファジー活性もほとんど示さなかった.
リン酸化Ser428およびリン酸化Ser429を特異的に認識する抗体を作製し,生体におけるこれらSerのリン酸化の状態を確認したところ,富栄養条件においてはリン酸化が検出されたが,ラパマイシンを処理したとき,あるいは,飢餓条件においてリン酸化はほとんどみられなくなった.すなわち,Atg13のSer428およびSer429は富栄養条件においてリン酸化されており,飢餓により脱リン酸化することが示された.以上のことから,Atg13とAtg17との結合はAtg13のSer428およびSer429のリン酸化により負に制御されていることが明らかになった.
5.飢餓によるAtg1複合体の形成のモデル
今回,明らかにされた結晶構造や,それにもとづくin vitroおよびin vivoにおける解析の結果を要約したモデルはつぎのようなものである(図2).富栄養条件においてAtg13はTOR複合体1により高度にリン酸化されている.Atg1との結合領域は2つの領域に分かれており,Atg13MIM(N) がAtg1との結合の主要な部分を担っており,Atg13MIM(C) はAtg1との結合の制御にかかわっている.Atg13MIM(N) だけでもAtg1と結合できるが,Atg13MIM(C) が脱リン酸化されてAtg1との結合にくわわると,Atg1とAtg13との親和性は増強され,結果として,細胞におけるAtg1-Atg13複合体の割合が増加する.Atg13とAtg17との結合もリン酸化により制御されている.ただし,この場合は結合に直接的にかかわるAtg13のもつSerそれ自体がリン酸化により制御されており,このSerがリン酸化されると電荷的な反発によりAtg17と結合できなくなる.飢餓条件においてAtg13が脱リン酸化されると,Atg13がAtg1およびAtg17と強く結合することによりAtg1複合体(Atg1-Atg13-Atg17-Atg29-Atg31複合体)が完成し,それがPASの形成の足場となりオートファジーが始動する.
おわりに
今回の論文では,出芽酵母におけるAtg1複合体の形成機構を解明したが,哺乳類ではULK1,Atg13,FIP200,Atg101のつくるULK1複合体がオートファジーの始動について同様の役割をはたすことが知られている9).ULK1にはAtg1のMITドメインが保存されていることから,Atg13との相互作用はMITドメインとMIM領域との相互作用と考えられる.しかし,出芽酵母においてAtg1との結合の制御にはたらいているAtg13MIM(C) のもつSerは哺乳類のAtg13において保存されておらず,実際に,ULK1複合体は栄養状態にかかわらずつねにAtg13と結合していると報告されている.一方,Atg17とFIP200との配列相同性は低いが,Atg17と同様にFIP200もコイルドコイルタンパク質と予測されること,また,機能的にも類似していることから,FIP200はAtg17の機能ホモログと考えられている10).哺乳類の場合,FIP200もまた恒常的にULK1と複合体を形成していると報告されている10).しかしながら,出芽酵母におけるAtg13とAtg17との結合のリン酸化による厳密な制御を目のあたりにすると,哺乳類におけるAtg13とFIP200との結合もまたリン酸化により制御されていることが期待される.たとえば,ULK1複合体の状態を保ったまま,構成タンパク質のあいだの相互作用の組替えなどが脱リン酸化によりひき起こされる可能性も考えられる.
今回,明らかになった結晶構造にはAtg1のもつキナーゼドメインは含まれておらず,Atg1複合体の形成がAtg1のキナーゼ活性をどのように制御しているのかは未解明のままである.Atg1の活性化にはそれ自体の二量体化による自己リン酸化が重要であるとの報告があるので,Atg17が二量体として存在することが関係するというアイディアもうかぶが,全容の解明については,今後,行われるであろうキナーゼドメインを含んだ結晶構造の解析に期待したい.
文 献
- Mizushima, N. & Komatsu, M.: Autophagy: renovation of cells and tissues. Cell, 147, 728-741 (2011)[PubMed]
- Mizushima, N., Yoshimori, T. & Ohsumi, Y.: The role of Atg proteins in autophagosome formation. Annu. Rev. Cell Dev. Biol., 27, 107-132 (2011)[PubMed]
- Suzuki, K. & Ohsumi, Y.: Current knowledge of the pre-autophagosomal structure (PAS). FEBS Lett., 584, 1280-1286 (2010)[PubMed]
- Noda, T. & Ohsumi, Y.: Tor, a phosphatidylinositol kinase homologue, controls autophagy in yeast. J. Biol. Chem., 273, 3963-3966 (1998)[PubMed]
- Kamada, Y., Funakoshi, T., Shintani, T. et al.: Tor-mediated induction of autophagy via an Apg1 protein kinase complex. J. Cell Biol., 150, 1507-1513 (2000)[PubMed]
- Kawamata, T., Kamada, Y., Kabeya, Y. et al.: Organization of the pre-autophagosomal structure responsible for autophagosome formation. Mol. Biol. Cell, 19, 2039-2050 (2008)[PubMed]
- Jao, C. C., Ragusa, M. J., Stanley, R. E. et al.: A HORMA domain in Atg13 mediates PI 3-kinase recruitment in autophagy. Proc. Natl. Acad. Sci. USA, 110, 5486-5491 (2013)[PubMed]
- Ragusa, M. J., Stanley, R. E., & Hurley, J. H.: Architecture of the Atg17 complex as a scaffold for autophagosome biogenesis. Cell, 151, 1501-1512 (2012)[PubMed]
- Mizushima, N.: The role of the Atg1/ULK1 complex in autophagy regulation. Curr. Opin. Cell Biol., 22, 132-139 (2010)[PubMed]
- Hara, T., Takamura, A., Kishi, C. et al.: FIP200, a ULK-interacting protein, is required for autophagosome formation in mammalian cells. J. Cell Biol., 181, 497-510 (2008)[PubMed]
著者プロフィール
略歴:2009年 北海道大学大学院薬学研究科にて博士号取得,同 研究員を経て,2011年より微生物化学研究所 博士研究員.
研究テーマ:オートファジーの構造生物学.
関心事:子供の成長,結晶の成長.
野田 展生(Nobuo N. Noda)
微生物化学研究所 主席研究員.
© 2014 藤岡優子・野田展生 Licensed under CC 表示 2.1 日本