脱ユビキチン化酵素OTUB2によるDNA 2本鎖切断に応答性のユビキチン化およびDNA修復経路の選択の制御
加藤希世子・中嶋一裕・中田慎一郎
(大阪大学大学院医学系研究科 細胞応答制御学)
email:加藤希世子,中嶋一裕,中田慎一郎
DOI: 10.7875/first.author.2014.038
Fine-tuning of DNA damage-dependent ubiquitination by OTUB2 supports the DNA repair pathway choice.
Kiyoko Kato, Kazuhiro Nakajima, Ayako Ui, Yuri Muto-Terao, Hideaki Ogiwara, Shinichiro Nakada
Molecular Cell, 53, 617-630 (2014)
DNA 2本鎖切断修復においては,リン酸化についで起こるユビキチン化による翻訳後修飾が重要な役割を担う.ユビキチンリガーゼRNF8に依存性のユビキチン化は,DNA 2本鎖切断応答に関連するタンパク質のDNA 2本鎖切断部位への局在およびクロマチン構造の変化を誘導し,遺伝子変異や細胞死を抑制する重要な役割を担っている.しかし,DNA 2本鎖切断修復におけるユビキチン化の機能はあまりよくわかっていない.この研究では,RNF8によるユビキチン化および脱ユビキチン化酵素OTUB2による脱ユビキチン化による,DNA 2本鎖切断に応答性の相反的なユビキチン化の制御が,適切なDNA修復経路を選択する機構を支援していることを示した.DNA 2本鎖切断応答の初期に起こるRNF8に依存性の急激なユビキチン化はDNA修復経路の選択を非相同末端結合にかたよらせてしまうが,脱ユビキチン化酵素OTUB2による抑制的な制御により適切なレベルのユビキチン化が起こり,これにより相同組換え修復を選択することの可能な環境がつくられていると考えられた.
DNA 2本鎖切断(DNA double-strand break:DSB)は,適切に修復されなければ遺伝子変異や細胞死の誘導される非常に危険なDNA損傷である.DNA 2本鎖切断修復には,大きく分けると相同組換え修復(homologous recombinaiton:HR)と非相同末端結合(non-homologous end joining:NHEJ)の2つの経路が存在する.相同組換え修復は姉妹染色体を鋳型としてDNA配列をもとのとおりに修復する経路であり,細胞周期のS期とG2期において限定的に利用され,時間もかかる.一方,非相同末端結合は切断により生じたDNA末端をすばやく直接に結合させる経路で,細胞周期のすべてにおいて利用されるが,修復部位においてDNAが変異する場合がある.S期およびG2期にはどちらの経路も選択することが可能であるが,その選択にかかわる分子機構が解明されはじめたのは最近のことである.
相同組換え修復と非相同末端結合との違いは,切断により生じたDNA末端の処理の違いにおいて顕著に現われる.相同組換え修復では,切断により生じたDNA末端を姉妹染色分体に侵入させるため,一方のDNA鎖を削り込み3’側の飛び出した1本鎖DNAをつくりだす必要がある.削り込みを受けたDNA末端は非相同末端結合では修復されにくい.このことから,DNA末端の削り込みをうけるか,DNA末端の保護が行われるかが,DNA修復経路の選択における第1の分岐点となる.
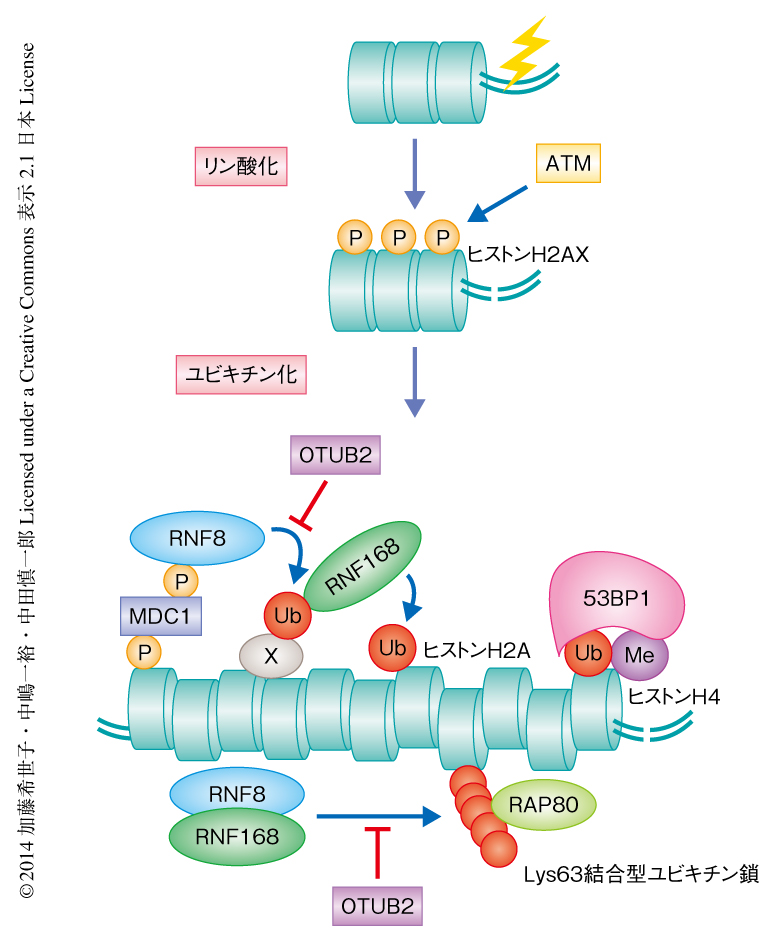
近年の研究により,DNA 2本鎖切断部位に集積するタンパク質のうち,53BP1およびRAP80はDNA末端の削り込みを抑制しDNA末端の保護にはたらくことが明らかにされている1-6).53BP1およびRAP80のDNA 2本鎖切断部位への局在は,ユビキチンリガーゼであるRNF8およびRNF168による,DNA 2本鎖切断部位におけるさまざまなタンパク質のユビキチン化に依存している7,8)(図1).そこで,筆者らは,RNF8およびRNF168に依存性のユビキチン化が増強された場合にはDNA末端の削り込みが強く抑制され,DNA修復経路は非相同末端結合にかたよるのではないかという仮説をたてた.この仮説を検証するため,まず,非相同末端結合が開始されるまでの短い時間にDNA 2本鎖切断部位におけるユビキチン化を抑制するような脱ユビキチン化酵素を発見し,その脱ユビキチン化酵素の発現を抑制した細胞,すなわち,DNA 2本鎖切断に依存性のユビキチン化が増強していると予想される細胞におけるDNA修復経路の選択の状況を検証することにした.
DNA 2本鎖切断を起こす薬剤のひとつであるネオカルチノスタチンによる処理から5分のちの,DNA 2本鎖切断部位へのRAP80の集積の増強をリードアウトとして,すべての脱ユビキチン化酵素に対するsiRNAライブラリーを用いてスクリーニングを行った.その結果,5つのヒットが得られ,そのなかからOTUB2の機能について解析を進めた.siRNAによりOTUB2をノックダウンした細胞をネオカルチノスタチンにより処理したのち,時間をおって固定し,結合型ユビキチンに特異的な抗体やDNA 2本鎖切断応答に関連するタンパク質に対する抗体を用いて免疫染色を行った.OTUB2ノックダウン細胞におけるDNA 2本鎖切断部位のユビキチン化は,ネオカルチノスタチンによる処理の5~20分のちには増強していたものの,1時間のちには対照となる細胞と同じ程度になっていた.ユビキチン化の上流において起こるヒストンH2AXのリン酸化およびMDC1のDNA 2本鎖切断部位への局在に変化はなかった.このことから,OTUB2ノックダウン細胞ではユビキチン化が通常より加速していると考えられた.RNF8によるユビキチン化に依存してDNA 2本鎖切断部位に局在するユビキチンリガーゼRNF168,RAP80,53BP1のDNA 2本鎖切断部位への集積も,ネオカルチノスタチンによる処理の5~30分のちには増強していた.一方,OTUB2と同時にRNF8あるいはRNF168をノックダウンした場合には,53BP1のDNA 2本鎖切断部位への局在は完全に抑制された.このことから,OTUB2はRNF8に依存性のDNA 2本鎖切断応答に抑制的にかかわっていると考えられた.
FLAGタグを付加したOTUB2を過剰発現させた細胞をネオカルチノスタチンにより処理したのち1時間において固定し,抗FLAG抗体およびDNA 2本鎖切断応答に関連するタンパク質に対する抗体を用いて免疫染色を行った.野生型のOTUB2を過剰発現させた場合には,DNA 2本鎖切断部位におけるユビキチン化,および,DNA 2本鎖切断部位へのRNF168,53BP1,RAP80の局在は強く抑制されていた.一方,脱ユビキチン活性中心に変異をもつOTUB2の過剰発現ではこのような抑制は認められなかった.DNA 2本鎖切断応答シグナルの上流からみていくと,OTUB2の過剰発現はヒストンH2AXのリン酸化,および,DNA 2本鎖切断部位へのMDC1およびRNF8の局在に対し影響を及ぼさなかった.DNA 2本鎖切断部位へのRNF168のリクルートには,RNF8のDNA 2本鎖切断部位への局在およびユビキチン化活性が必要であることから8),OTUB2はRNF8に依存性のユビキチン化を脱ユビキチン化酵素活性に依存して抑制していると考えられた(図1).
OTUB2ときわめてよく似た構造をもつ脱ユビキチン化酵素としてOTUB1がある.以前に筆者らは,OTUB1がユビキチン結合酵素と結合してその活性を抑制することを報告した9)(新着論文レビュー でも掲載).OTUB1の場合,脱ユビキチン化酵素の活性に非依存的にDNA 2本鎖切断部位におけるユビキチン化を抑制することから,OTUB1とOTUB2がDNA 2本鎖切断応答を抑制する分子機構は完全に異なることが示唆された.
OTUB2をノックダウンしてDNA 2本鎖切断応答の初期に起こるユビキチン化を増強すると,DNA修復経路のバランスはどうなるだろうか.細胞をネオカルチノスタチンにより処理したのち,DNA 2本鎖切断のマーカーとして汎用されているリン酸化H2AXおよび結合型ユビキチンのフォーカス形成を経時的に測定した.対照となる細胞と比べOTUB2ノックダウン細胞ではすみやかにフォーカスが消失したことから,DNA 2本鎖切断が早期に減少していることが考えられた.そこで,ネオカルチノスタチン処理ののちのDNA 2本鎖切断の修復の効率を,細胞1個あたりのDNA切断の量を測定できるコメットアッセイ法を用いて求めたところ,OTUB2ノックダウン細胞は処理ののち2時間においてより高い修復効率を示した.さらに,DNA依存性プロテインキナーゼの阻害剤を用いて非相同末端結合を抑制したところ,OTUB2ノックダウン細胞においてDNA 2本鎖切断修復の促進が抑制された.
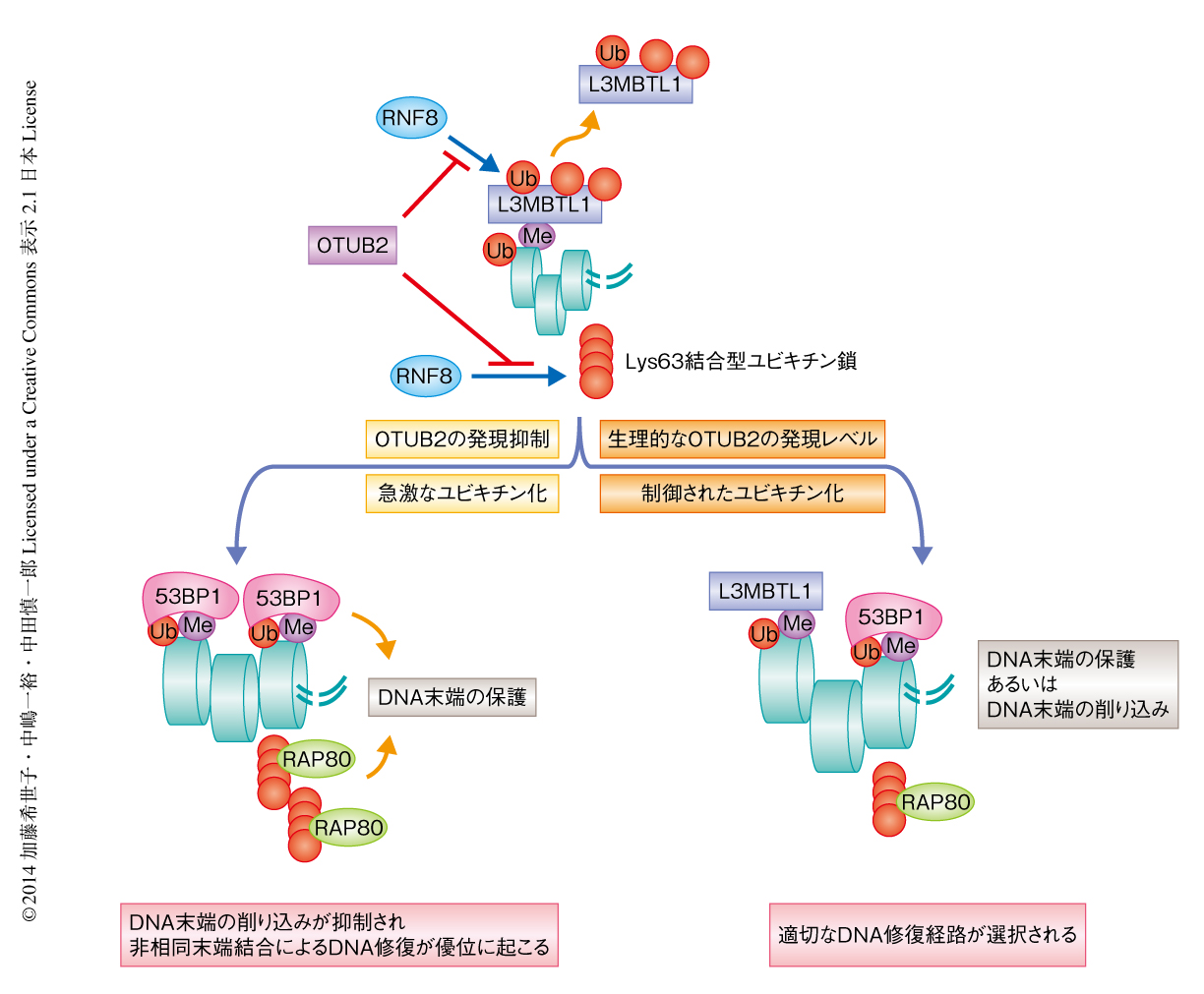
相同組換え修復によりDNA 2本鎖切断が修復されるとGFPが発現するレポーターアッセイにより相同組換え修復の効率を測定したところ,OTUB2ノックダウン細胞では相同組換え修復の効率は低下していた.さらに,OTUB2ノックダウン細胞ではDNA末端の削り込みによりつくりだされた1本鎖DNAに結合するRPA,および,RPAと交換されるかたちで1本鎖DNAに結合して姉妹染色分体との組換えを担うタンパク質であるRAD51のDNA 2本鎖切断部位への局在も低下していた.OTUB2と同時にRAP80あるいは53BP1をノックダウンすることにより,RPAおよびRAD51のフォーカス形成は回復した.これらのことから,OTUB2ノックダウン細胞では53BP1およびRAP80に依存したDNA末端の保護が強まることにより非相同末端結合が優先的に選択され,相同組換え修復が選択されにくい状況になっていることが示された(図2).
OTUB2はDNA 2本鎖切断部位においてどのようなユビキチン化を抑制しているのだろうか.RAP80および53BP1のDNA 2本鎖切断部位への局在には,ユビキチンのLys63とGly76とが共有結合をくり返したLys63結合型ユビキチン鎖の伸長,および,ヒストンH2Aのユビキチン化が必要である.RAP80はK63結合型ユビキチン鎖に特異的に結合し,53BP1は20番目のLysがジメチル化したヒストンH4,および,15番目のLysがモノユビキチン化したヒストンH2A結合することにより,DNA 2本鎖切断部位に局在することが知られている.しかし,ヒストンH4の20番目のLysのジメチル基にはPolycombタンパク質であるL3MBTL1が結合しており,L3MBTL1がRNF8によるユビキチン化をうけてDNA 2本鎖切断部位から取り除かれなければ,53BP1はここにアクセスできない.そこで,OTUB2がL3MBTL1のユビキチン化,Lys63結合型ユビキチン鎖の伸長,ヒストンH2Aのユビキチン化を抑制するかどうか検証した.細胞にHisタグを付加したL3MBTL1,RNF8,ユビキチンを強制発現させ,変性条件においてL3MBTL1をプルダウンしたところ,ユビキチン化したL3MBTL1からなるスメアをウェスタンブロット法により検出することができた.この実験系において,野生型のOTUB2を同時に強制発現させたところL3MBTL1のユビキチン化は顕著に抑制されたが,脱ユビキチン活性中心に変異をもつOTUB2を強制発現させても抑制は起こらなかった.RNF8の強制発現の代わりに薬剤によりDNA 2本鎖切断を発生させても同様の結果が得られた.また,in vitroにおいて,OTUB2はユビキチン化したL3MBTL1を脱ユビキチン化したが,OTUB1は脱ユビキチン化しなかった.さらに,OTUB2はLys63結合型ユビキチン鎖の伸長を抑制し,かつ,Lys63結合型テトラユビキチンをモノユビキチンに分解した.これに対し,RNF168の過剰発現により起こる細胞内におけるヒストンのユビキチン化は,OTUB2を同時に過剰発現したときには抑制されなかったが,OTUB1を同時に過剰発現したときには抑制された.これらのことから,OTUB2はL3MBTL1のユビキチン化およびLys63結合型ユビキチン鎖の伸長を抑制していると結論づけた.
トポイソメラーゼIの阻害剤であるカンプトテシンによるDNA 1本鎖切断は,DNA複製をへてDNA 2本鎖切断に変換され,相同組換え修復により修復されることが知られている.OTUB2ノックダウン細胞は,DNA修復経路の選択が非相同末端結合にかたよることから予想されたとおり,カンプトテシンに対し高い感受性を示した.
筆者らが今回の研究により得たデータを総合すると,DNA 2本鎖切断の際に起こるRNF8に依存性のユビキチン化が急速に進む場合,DNA末端の保護にはたらくタンパク質もまた急速にDNA 2本鎖切断部位に集積するため,非相同末端結合が選択されやすく,また,相同組換え修復は選択されにくくなると考えられた.OTUB2は急激なユビキチン化を抑制するためのブレーキとしてはたらき,ユビキチン化のレベルを適切に微調整しているのではないかと考えられる.しかし,OTUB2はDNA修復経路の選択に積極的にかかわるというよりむしろ,DNA修復経路の選択が適切に行われるための環境をつくりだしているのではないだろうか.ユビキチン化の下流におけるDNA修復経路の選択の研究もさかんに行われており,近い将来,DNA修復経路の選択機構の概要が明らかにされることが期待される.
略歴:2011年 大阪大学大学院医学系研究科 修了,2012年より同 特任研究員.
研究テーマ:DNA 2本鎖切断に対する応答.
抱負:細胞の応答機構を解析し,社会に役立つものをつくる.
中嶋 一裕(Kazuhiro Nakajima)
略歴:大阪大学大学院医学系研究科 在学中.
研究テーマ:DNA 2本鎖切断修復の機構.
中田 慎一郎(Shinichiro Nakada)
大阪大学大学院医学系研究科 独立准教授.
© 2014 加藤希世子・中嶋一裕・中田慎一郎 Licensed under CC 表示 2.1 日本
(大阪大学大学院医学系研究科 細胞応答制御学)
email:加藤希世子,中嶋一裕,中田慎一郎
DOI: 10.7875/first.author.2014.038
Fine-tuning of DNA damage-dependent ubiquitination by OTUB2 supports the DNA repair pathway choice.
Kiyoko Kato, Kazuhiro Nakajima, Ayako Ui, Yuri Muto-Terao, Hideaki Ogiwara, Shinichiro Nakada
Molecular Cell, 53, 617-630 (2014)
要 約
DNA 2本鎖切断修復においては,リン酸化についで起こるユビキチン化による翻訳後修飾が重要な役割を担う.ユビキチンリガーゼRNF8に依存性のユビキチン化は,DNA 2本鎖切断応答に関連するタンパク質のDNA 2本鎖切断部位への局在およびクロマチン構造の変化を誘導し,遺伝子変異や細胞死を抑制する重要な役割を担っている.しかし,DNA 2本鎖切断修復におけるユビキチン化の機能はあまりよくわかっていない.この研究では,RNF8によるユビキチン化および脱ユビキチン化酵素OTUB2による脱ユビキチン化による,DNA 2本鎖切断に応答性の相反的なユビキチン化の制御が,適切なDNA修復経路を選択する機構を支援していることを示した.DNA 2本鎖切断応答の初期に起こるRNF8に依存性の急激なユビキチン化はDNA修復経路の選択を非相同末端結合にかたよらせてしまうが,脱ユビキチン化酵素OTUB2による抑制的な制御により適切なレベルのユビキチン化が起こり,これにより相同組換え修復を選択することの可能な環境がつくられていると考えられた.
はじめに
DNA 2本鎖切断(DNA double-strand break:DSB)は,適切に修復されなければ遺伝子変異や細胞死の誘導される非常に危険なDNA損傷である.DNA 2本鎖切断修復には,大きく分けると相同組換え修復(homologous recombinaiton:HR)と非相同末端結合(non-homologous end joining:NHEJ)の2つの経路が存在する.相同組換え修復は姉妹染色体を鋳型としてDNA配列をもとのとおりに修復する経路であり,細胞周期のS期とG2期において限定的に利用され,時間もかかる.一方,非相同末端結合は切断により生じたDNA末端をすばやく直接に結合させる経路で,細胞周期のすべてにおいて利用されるが,修復部位においてDNAが変異する場合がある.S期およびG2期にはどちらの経路も選択することが可能であるが,その選択にかかわる分子機構が解明されはじめたのは最近のことである.
相同組換え修復と非相同末端結合との違いは,切断により生じたDNA末端の処理の違いにおいて顕著に現われる.相同組換え修復では,切断により生じたDNA末端を姉妹染色分体に侵入させるため,一方のDNA鎖を削り込み3’側の飛び出した1本鎖DNAをつくりだす必要がある.削り込みを受けたDNA末端は非相同末端結合では修復されにくい.このことから,DNA末端の削り込みをうけるか,DNA末端の保護が行われるかが,DNA修復経路の選択における第1の分岐点となる.
近年の研究により,DNA 2本鎖切断部位に集積するタンパク質のうち,53BP1およびRAP80はDNA末端の削り込みを抑制しDNA末端の保護にはたらくことが明らかにされている1-6).53BP1およびRAP80のDNA 2本鎖切断部位への局在は,ユビキチンリガーゼであるRNF8およびRNF168による,DNA 2本鎖切断部位におけるさまざまなタンパク質のユビキチン化に依存している7,8)(図1).そこで,筆者らは,RNF8およびRNF168に依存性のユビキチン化が増強された場合にはDNA末端の削り込みが強く抑制され,DNA修復経路は非相同末端結合にかたよるのではないかという仮説をたてた.この仮説を検証するため,まず,非相同末端結合が開始されるまでの短い時間にDNA 2本鎖切断部位におけるユビキチン化を抑制するような脱ユビキチン化酵素を発見し,その脱ユビキチン化酵素の発現を抑制した細胞,すなわち,DNA 2本鎖切断に依存性のユビキチン化が増強していると予想される細胞におけるDNA修復経路の選択の状況を検証することにした.
1.OTUB2のノックダウンによりDNA 2本鎖切断部位におけるユビキチン化は増強する
DNA 2本鎖切断を起こす薬剤のひとつであるネオカルチノスタチンによる処理から5分のちの,DNA 2本鎖切断部位へのRAP80の集積の増強をリードアウトとして,すべての脱ユビキチン化酵素に対するsiRNAライブラリーを用いてスクリーニングを行った.その結果,5つのヒットが得られ,そのなかからOTUB2の機能について解析を進めた.siRNAによりOTUB2をノックダウンした細胞をネオカルチノスタチンにより処理したのち,時間をおって固定し,結合型ユビキチンに特異的な抗体やDNA 2本鎖切断応答に関連するタンパク質に対する抗体を用いて免疫染色を行った.OTUB2ノックダウン細胞におけるDNA 2本鎖切断部位のユビキチン化は,ネオカルチノスタチンによる処理の5~20分のちには増強していたものの,1時間のちには対照となる細胞と同じ程度になっていた.ユビキチン化の上流において起こるヒストンH2AXのリン酸化およびMDC1のDNA 2本鎖切断部位への局在に変化はなかった.このことから,OTUB2ノックダウン細胞ではユビキチン化が通常より加速していると考えられた.RNF8によるユビキチン化に依存してDNA 2本鎖切断部位に局在するユビキチンリガーゼRNF168,RAP80,53BP1のDNA 2本鎖切断部位への集積も,ネオカルチノスタチンによる処理の5~30分のちには増強していた.一方,OTUB2と同時にRNF8あるいはRNF168をノックダウンした場合には,53BP1のDNA 2本鎖切断部位への局在は完全に抑制された.このことから,OTUB2はRNF8に依存性のDNA 2本鎖切断応答に抑制的にかかわっていると考えられた.
2.OTUB2はRNF8によるユビキチン化反応を阻害する
FLAGタグを付加したOTUB2を過剰発現させた細胞をネオカルチノスタチンにより処理したのち1時間において固定し,抗FLAG抗体およびDNA 2本鎖切断応答に関連するタンパク質に対する抗体を用いて免疫染色を行った.野生型のOTUB2を過剰発現させた場合には,DNA 2本鎖切断部位におけるユビキチン化,および,DNA 2本鎖切断部位へのRNF168,53BP1,RAP80の局在は強く抑制されていた.一方,脱ユビキチン活性中心に変異をもつOTUB2の過剰発現ではこのような抑制は認められなかった.DNA 2本鎖切断応答シグナルの上流からみていくと,OTUB2の過剰発現はヒストンH2AXのリン酸化,および,DNA 2本鎖切断部位へのMDC1およびRNF8の局在に対し影響を及ぼさなかった.DNA 2本鎖切断部位へのRNF168のリクルートには,RNF8のDNA 2本鎖切断部位への局在およびユビキチン化活性が必要であることから8),OTUB2はRNF8に依存性のユビキチン化を脱ユビキチン化酵素活性に依存して抑制していると考えられた(図1).
OTUB2ときわめてよく似た構造をもつ脱ユビキチン化酵素としてOTUB1がある.以前に筆者らは,OTUB1がユビキチン結合酵素と結合してその活性を抑制することを報告した9)(新着論文レビュー でも掲載).OTUB1の場合,脱ユビキチン化酵素の活性に非依存的にDNA 2本鎖切断部位におけるユビキチン化を抑制することから,OTUB1とOTUB2がDNA 2本鎖切断応答を抑制する分子機構は完全に異なることが示唆された.
3.OTUB2の発現抑制により初期のDNA 2本鎖切断修復は促進されるが相同組換え修復は抑制される
OTUB2をノックダウンしてDNA 2本鎖切断応答の初期に起こるユビキチン化を増強すると,DNA修復経路のバランスはどうなるだろうか.細胞をネオカルチノスタチンにより処理したのち,DNA 2本鎖切断のマーカーとして汎用されているリン酸化H2AXおよび結合型ユビキチンのフォーカス形成を経時的に測定した.対照となる細胞と比べOTUB2ノックダウン細胞ではすみやかにフォーカスが消失したことから,DNA 2本鎖切断が早期に減少していることが考えられた.そこで,ネオカルチノスタチン処理ののちのDNA 2本鎖切断の修復の効率を,細胞1個あたりのDNA切断の量を測定できるコメットアッセイ法を用いて求めたところ,OTUB2ノックダウン細胞は処理ののち2時間においてより高い修復効率を示した.さらに,DNA依存性プロテインキナーゼの阻害剤を用いて非相同末端結合を抑制したところ,OTUB2ノックダウン細胞においてDNA 2本鎖切断修復の促進が抑制された.
相同組換え修復によりDNA 2本鎖切断が修復されるとGFPが発現するレポーターアッセイにより相同組換え修復の効率を測定したところ,OTUB2ノックダウン細胞では相同組換え修復の効率は低下していた.さらに,OTUB2ノックダウン細胞ではDNA末端の削り込みによりつくりだされた1本鎖DNAに結合するRPA,および,RPAと交換されるかたちで1本鎖DNAに結合して姉妹染色分体との組換えを担うタンパク質であるRAD51のDNA 2本鎖切断部位への局在も低下していた.OTUB2と同時にRAP80あるいは53BP1をノックダウンすることにより,RPAおよびRAD51のフォーカス形成は回復した.これらのことから,OTUB2ノックダウン細胞では53BP1およびRAP80に依存したDNA末端の保護が強まることにより非相同末端結合が優先的に選択され,相同組換え修復が選択されにくい状況になっていることが示された(図2).
4.OTUB2はL3MBTL1のユビキチン化およびLys63結合型ユビキチン鎖の伸長を抑制する
OTUB2はDNA 2本鎖切断部位においてどのようなユビキチン化を抑制しているのだろうか.RAP80および53BP1のDNA 2本鎖切断部位への局在には,ユビキチンのLys63とGly76とが共有結合をくり返したLys63結合型ユビキチン鎖の伸長,および,ヒストンH2Aのユビキチン化が必要である.RAP80はK63結合型ユビキチン鎖に特異的に結合し,53BP1は20番目のLysがジメチル化したヒストンH4,および,15番目のLysがモノユビキチン化したヒストンH2A結合することにより,DNA 2本鎖切断部位に局在することが知られている.しかし,ヒストンH4の20番目のLysのジメチル基にはPolycombタンパク質であるL3MBTL1が結合しており,L3MBTL1がRNF8によるユビキチン化をうけてDNA 2本鎖切断部位から取り除かれなければ,53BP1はここにアクセスできない.そこで,OTUB2がL3MBTL1のユビキチン化,Lys63結合型ユビキチン鎖の伸長,ヒストンH2Aのユビキチン化を抑制するかどうか検証した.細胞にHisタグを付加したL3MBTL1,RNF8,ユビキチンを強制発現させ,変性条件においてL3MBTL1をプルダウンしたところ,ユビキチン化したL3MBTL1からなるスメアをウェスタンブロット法により検出することができた.この実験系において,野生型のOTUB2を同時に強制発現させたところL3MBTL1のユビキチン化は顕著に抑制されたが,脱ユビキチン活性中心に変異をもつOTUB2を強制発現させても抑制は起こらなかった.RNF8の強制発現の代わりに薬剤によりDNA 2本鎖切断を発生させても同様の結果が得られた.また,in vitroにおいて,OTUB2はユビキチン化したL3MBTL1を脱ユビキチン化したが,OTUB1は脱ユビキチン化しなかった.さらに,OTUB2はLys63結合型ユビキチン鎖の伸長を抑制し,かつ,Lys63結合型テトラユビキチンをモノユビキチンに分解した.これに対し,RNF168の過剰発現により起こる細胞内におけるヒストンのユビキチン化は,OTUB2を同時に過剰発現したときには抑制されなかったが,OTUB1を同時に過剰発現したときには抑制された.これらのことから,OTUB2はL3MBTL1のユビキチン化およびLys63結合型ユビキチン鎖の伸長を抑制していると結論づけた.
5.OTUB2ノックダウン細胞はトポイソメラーゼI阻害剤に高い感受性を示す
トポイソメラーゼIの阻害剤であるカンプトテシンによるDNA 1本鎖切断は,DNA複製をへてDNA 2本鎖切断に変換され,相同組換え修復により修復されることが知られている.OTUB2ノックダウン細胞は,DNA修復経路の選択が非相同末端結合にかたよることから予想されたとおり,カンプトテシンに対し高い感受性を示した.
おわりに
筆者らが今回の研究により得たデータを総合すると,DNA 2本鎖切断の際に起こるRNF8に依存性のユビキチン化が急速に進む場合,DNA末端の保護にはたらくタンパク質もまた急速にDNA 2本鎖切断部位に集積するため,非相同末端結合が選択されやすく,また,相同組換え修復は選択されにくくなると考えられた.OTUB2は急激なユビキチン化を抑制するためのブレーキとしてはたらき,ユビキチン化のレベルを適切に微調整しているのではないかと考えられる.しかし,OTUB2はDNA修復経路の選択に積極的にかかわるというよりむしろ,DNA修復経路の選択が適切に行われるための環境をつくりだしているのではないだろうか.ユビキチン化の下流におけるDNA修復経路の選択の研究もさかんに行われており,近い将来,DNA修復経路の選択機構の概要が明らかにされることが期待される.
文 献
- Escribano-Diaz, C., Orthwein, A., Fradet-Turcotte, A. et al.: A cell cycle-dependent regulatory circuit composed of 53BP1-RIF1 and BRCA1-CtIP controls DNA repair pathway choice. Mol. Cell, 49, 872-883 (2013)[PubMed]
- Hu, Y., Scully, R., Sobhian, B. et al.: RAP80-directed tuning of BRCA1 homologous recombination function at ionizing radiation-induced nuclear foci. Genes Dev., 25, 685-700 (2011)[PubMed]
- Coleman, K. A. & Greenberg, R. A.: The BRCA1-RAP80 complex regulates DNA repair mechanism utilization by restricting end resection. J. Biol. Chem., 286, 13669-13680 (2011)[PubMed]
- Zimmermann, M., Lottersberger, F., Buonomo, S. B. et al.: 53BP1 regulates DSB repair using Rif1 to control 5' end resection. Science, 339, 700-704 (2013)[PubMed]
- Chapman, J. R., Barral, P., Vannier, J. B. et al.: RIF1 is essential for 53BP1-dependent nonhomologous end joining and suppression of DNA double-strand break resection. Mol. Cell, 49, 858-871 (2013)[PubMed]
- Di Virgilio, M., Callen, E., Yamane, A. et al.: Rif1 prevents resection of DNA breaks and promotes immunoglobulin class switching. Science, 339, 711-715 (2013)[PubMed]
- Kolas, N. K., Chapman, J. R., Nakada, S. et al.: Orchestration of the DNA-damage response by the RNF8 ubiquitin ligase. Science, 318, 1637-1640 (2007)[PubMed]
- Stewart, G. S., Panier, S., Townsend, K. et al.: The RIDDLE syndrome protein mediates a ubiquitin-dependent signaling cascade at sites of DNA damage. Cell, 136, 420-434 (2009)[PubMed]
- Nakada, S., Tai, I., Panier, S. et al.: Non-canonical inhibition of DNA damage-dependent ubiquitination by OTUB1. Nature, 466, 941-946 (2010)[PubMed] [新着論文レビュー]
著者プロフィール
略歴:2011年 大阪大学大学院医学系研究科 修了,2012年より同 特任研究員.
研究テーマ:DNA 2本鎖切断に対する応答.
抱負:細胞の応答機構を解析し,社会に役立つものをつくる.
中嶋 一裕(Kazuhiro Nakajima)
略歴:大阪大学大学院医学系研究科 在学中.
研究テーマ:DNA 2本鎖切断修復の機構.
中田 慎一郎(Shinichiro Nakada)
大阪大学大学院医学系研究科 独立准教授.
© 2014 加藤希世子・中嶋一裕・中田慎一郎 Licensed under CC 表示 2.1 日本