Tet1による始原生殖細胞におけるゲノムインプリンティングの消去
山口 新平
(米国Harvard Medical School,Boston Children’s Hospital)
email:山口新平
DOI: 10.7875/first.author.2013.164
Role of Tet1 in erasure of genomic imprinting.
Shinpei Yamaguchi, Li Shen, Yuting Liu, Damian Sendler, Yi Zhang
Nature, 504, 460-464 (2013)
ゲノムインプリンティングは対立遺伝子に特異的な遺伝子発現の様式であり,親の世代の精子および卵子それぞれに異なるパターンで樹立されたDNAのメチル化により制御される.精子あるいは卵子に特異的なDNAメチル化パターンの形成にさきだち,始原生殖細胞において体細胞型のDNAメチル化パターンの消去が起こるが,その分子機構はほとんど解明されていなかった.この研究において,筆者らは,5-メチル化シトシンに特異的なジオキシゲナーゼTet1が始原生殖細胞におけるインプリンティング遺伝子のDNAメチル化を消去するタンパク質であることを示した.Tet1を欠損した雄のマウスと野生型の雌のマウスとの交配により得られたマウスは,胎生致死などそれぞれの個体で異なる多様な表現型を示した.Tet1ノックアウトマウスにおいては,本来は始原生殖細胞において消去されるべきインプリンティング遺伝子のDNAメチル化が消去されず,精子でも高くDNAメチル化した状態で維持されていた.親の世代から残存したDNAメチル化はつぎの世代に伝わり,インプリンティング遺伝子の発現の異常およびさまざまな表現型をひき起こしていた.以上の結果から,Tet1がインプリンティング遺伝子のDNAメチル化の脱メチル化に重要な役割をはたしていることが証明された.
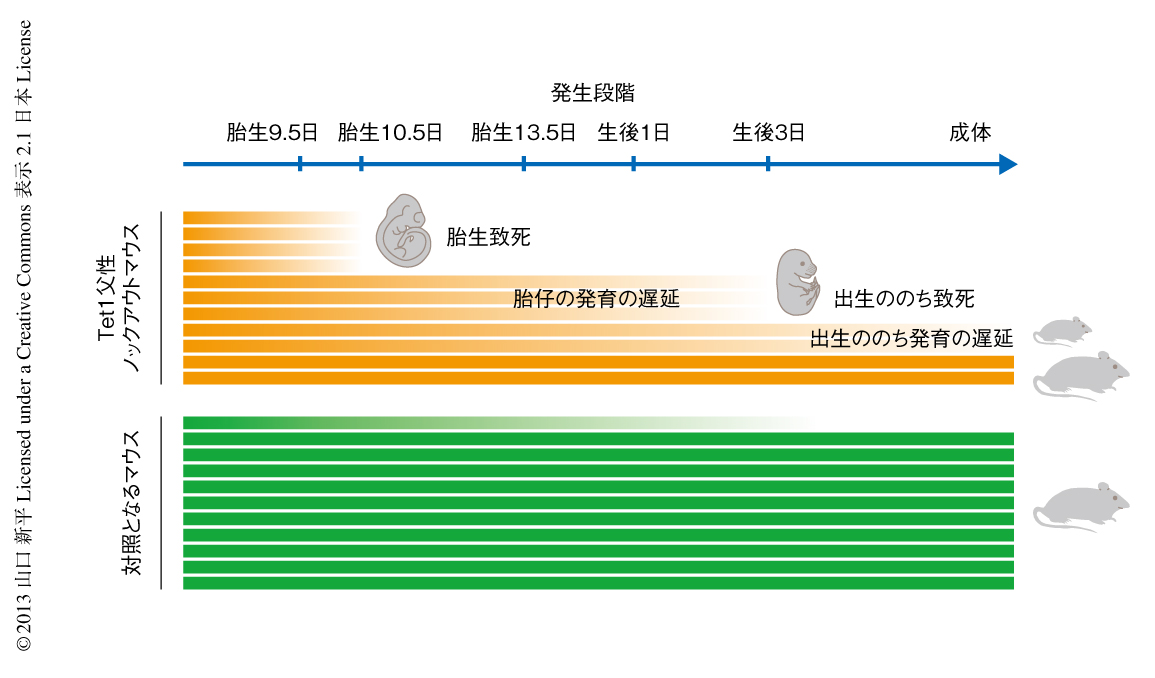
われわれ哺乳類では,ほとんどの遺伝子は父母それぞれに由来する2つの対立遺伝子から同じレベルで発現している.しかし,インプリンティング遺伝子は,父親(精子)由来あるいは母親(卵子)由来のどちらかの対立遺伝子からのみ発現するという特徴的な発現パターンを示す.この対立遺伝子に特異的な発現は,おもに対立遺伝子特異的メチル化領域(differentially methylated region:DMR)におけるDNAメチル化のパターンにより制御されている1).雄では胎仔期から新生仔期の精原細胞において,雌では出生ののちの卵母細胞において,精子あるいは卵子に特異的なDNAメチル化パターンが形成される.しかし,マウスの精子および卵子はもともと体細胞型のDNAメチル化パターンをもつ始原生殖細胞から分化する.そのため,精子あるいは卵子に特異的なDNAメチル化パターンが形成されるまえに,この体細胞型のDNAメチル化パターンを消去することが必要である.胎生8.0~13.5日の始原生殖細胞において起こるゲノムリプログラミングをつうじほとんどすべてのゲノムはDNA脱メチル化することがわかっていたが1),インプリンティング遺伝子のDNAメチル化の消去における責任タンパク質および分子機構は長いあいだ不明であった(図1).
ゲノムのリセットともいえる重要なイベントの機構が長らく不明だったのは,哺乳類におけるDNA脱メチル化の機構がほとんどわかっていなかったことが原因であった.しかし,2009年,Tetファミリーが5-メチル化シトシンを特異的に酸化する活性をもつことが発見され,この分野の研究が急速に進んだ2,3).筆者らは,Tetファミリーのうちゲノムリプログラミングの起こる時期の始原生殖細胞において特異的に発現するTet1に注目し,Tet1が減数分裂に関連する遺伝子におけるDNA脱メチル化に重要であることを発見した4)(新着論文レビュー でも掲載).はたして,Tet1はインプリンティング遺伝子のDNAメチル化の消去にもはたらいているのだろうか,それが,この研究の出発点であった.
インプリンティング遺伝子のDNAメチル化の消去の異常は精子に残存するDNAメチル化として受け継がれ,残存したDNAメチル化は受精ののちのつぎの世代に影響を及ぼすであろうと仮説をたてた.そこで,雄のTet1ノックアウトマウスと雌の野生型マウスとを交配し,次世代としてTet1父性ノックアウトマウスを得た.対照となるマウスとしては,雄のTet1ヘテロノックアウトマウスと雌の野生型マウスとの交配により得たマウスを用いた.Tet1父性ノックアウトマウスはTet1遺伝子についてヘテロであり,対照となるマウスの半分とは同一である.にもかかわらず,Tet1父性ノックアウトマウスのみに非常に多様な表現型が認められた.Tet1父性ノックアウトマウスは生まれたときの体の大きさに非常に大きなばらつきがあり,新生仔の体重は,対照となるマウスでは全個体の約70%が1.2~1.3 gにおさまるのに対し,Tet1父性ノックアウトマウスは1.1 g以下あるいは1.4 g以上の個体が70%以上をしめた.Tet1父性ノックアウトマウスの胎盤は対照となるマウスと比較して顕著に小さく,その大きさは体の大きさと正の相関を示したことから,少なくとも,体の大きさの減少は胎盤の異常が原因であることが示唆された.さらに,Tet1父性ノックアウトマウスの新生仔はその半分が出生から3日以内に死亡した.生き残ったマウスも大きなばらつきをもちながら発育に遅延を示し,3週齢の時点においてTet1父性ノックアウトマウスのうち約半数の個体は対照となるマウスに比べ30~70%の体重しかなかった.
このような発育の異常にくわえ,Tet1父性ノックアウトマウスは生まれてくる個体の数がすでに少ないことがわかった.野生型マウスとTet1ヘテロノックアウトマウスとの交配では平均7.5匹の仔が得られるのに対し,野生型マウスとTet1ノックアウトマウスとの交配では平均4匹しか生まれてこなかった.そこで,胎生13.5日において解剖を行ったところ,Tet1父性ノックアウトでは約35%の胚がすでに死亡し吸収されていることがわかった.胎生10.5日では約35%の胚が発生に明らかな異常を示していた.胎生9.5日では胚に形態的な異常はみつからなかったが,約35%の胚で胎盤に異常があった.組織学的な解析の結果,これらの胎盤では絨毛膜板の伸展が認められず,ラビリンスの形成も認められなかった.
以上の結果から,Tet1父性ノックアウトマウスの一部は,胚性致死,胎仔および胎盤の発生の異常,新生仔致死,出生ののちの発育の異常という多様な表現型を示すことがわかった(図2).
インプリンティング遺伝子の異常は胎盤あるいは胚の発生において表現型を示すことが知られている1).胎生致死の表現型が父方の対立遺伝子に特異的に発現するインプリンティング遺伝子Peg10遺伝子のノックアウトマウスと類似していたことから,胎生9.5日の胚におけるPeg10遺伝子の発現を解析した.その結果,異常な胎盤をもつ胚ではPeg10遺伝子の発現が完全に抑制されていることがわかった.胎生9.5日の胚における遺伝子発現プロファイルをRNAシークエンシング法により解析すると,対照となるマウスの胚において発現の認められた81個のインプリンティング遺伝子のうち,11~46個のインプリンティング遺伝子がTet1父性ノックアウトマウスの胚において異常な発現を示していた.表現型と同様,インプリンティング遺伝子の発現異常のパターンにも胚ごとにばらつきがみられたが,共通の傾向として,父方の対立遺伝子より発現するインプリンティング遺伝子が抑制され,母方の対立遺伝子より発現するインプリンティング遺伝子が活性化していた.このことから,父方の対立遺伝子が母方の対立遺伝子の発現様式を獲得していることが示唆された.
Tet1父性ノックアウトマウスにおけるインプリンティング遺伝子の発現の異常がDNAメチル化の異常に起因するのかどうか明らかにするため,Peg10遺伝子の対立遺伝子特異的メチル化領域におけるDNAメチル化について解析した.対照となる胚では予想どおり約50%のDNAメチル化が認められたのに対し,Peg10遺伝子の発現が抑制されていたTet1父性ノックアウトマウスの胚ではその対立遺伝子特異的メチル化領域はほぼ100%がDNAメチル化していた.このことは,通常はDNAメチル化をうけない父方の対立遺伝子もDNAメチル化されていることを示した.Peg10遺伝子のほかにも,Igf2r遺伝子の発現が異常な胚,および,Impact遺伝子の発現が異常な胚では,それぞれの対立遺伝子特異的メチル化領域においてほぼ100%のDNAメチル化が認められた.
胎盤の発達の異常も胚と同様にインプリンティング遺伝子の発現の異常が原因なのかどうか明らかにするため,胎生19.5日の胎盤を解析した.その結果,異常に小さい胎盤においてはIgf2r遺伝子およびPeg3遺伝子の発現に異常が認められた.胚における結果と同様,Peg3遺伝子の発現が抑制されていた胎盤ではその対立遺伝子特異的メチル化領域におけるDNAメチル化が上昇していた.Peg3ノックアウトマウスは同様の胎盤の異常を示すことから,Tet1父性ノックアウトマウスの胚における胎盤の発達の異常もインプリンティング遺伝子の発現の異常が原因であることが示唆された.
これらの結果から,通常はDNAメチル化をうけない父方の対立遺伝子がDNAメチル化されることがTet1父性ノックアウトマウスにおけるインプリンティング遺伝子の発現異常の原因であり,発生の異常につながることが示唆された.
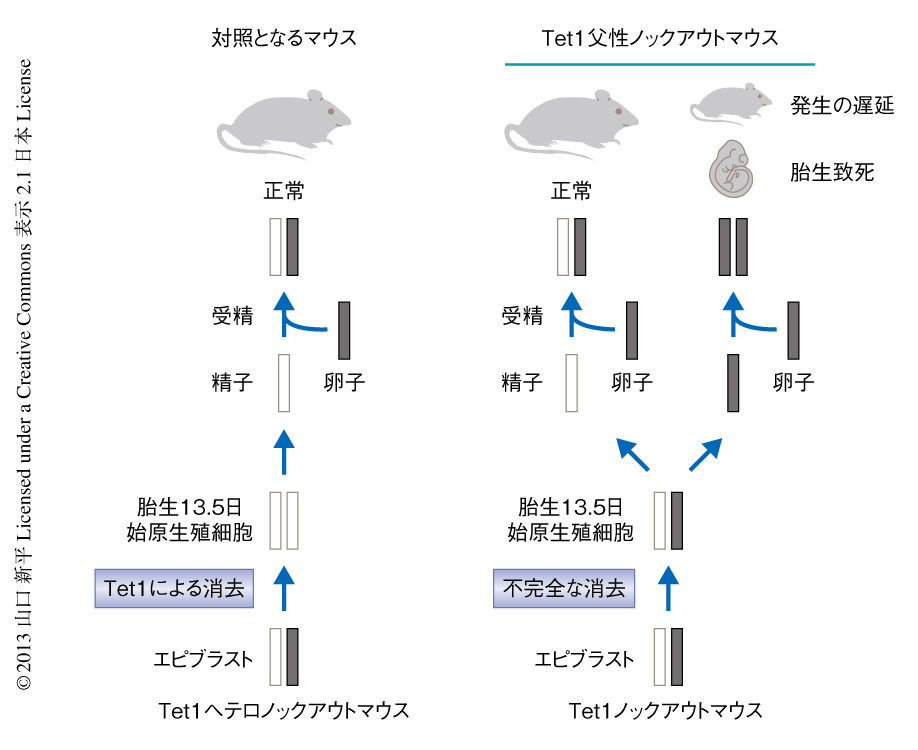
Tet1父性ノックアウトマウスの母親は野生型であるため,その胚および胎盤において認められたインプリンティング遺伝子の発現の異常は,父親であるTet1ノックアウトマウスの精子における異常なDNAメチル化が直接の原因だと予想された.もしTet1がインプリンティング遺伝子のDNAメチル化の消去に機能しているなら,Tet1ノックアウトマウスの精子においてみられる異常はゲノムリプログラミングの起こった直後の始原生殖細胞においてすでに現われているはずである.この仮説を検証するため,Tet1ノックアウトマウスの精子およびゲノムリプログラミングの直後である胎生13.5日の雄の始原生殖細胞の両方において,RRBS(reduced representation bisulfite sequencing)法5) を用いてゲノムワイドなDNAメチル化パターンを解析した.筆者らの過去の報告とは矛盾せず4),ゲノム全体的なDNAメチル化のパターンは精子でも始原生殖細胞でも大きな異常は認められなかった.これは,Tet1ノックアウトマウスにおいてもゲノムワイドなDNA脱メチル化やDNA再メチル化が異常なく起こっていることを示唆した.しかし,詳細な解析の結果,Tet1ノックアウトマウスの精子では493,始原生殖細胞では9665の高くDNAメチル化した領域が同定された.そして予想どおり,精子および始原生殖細胞の両方で共通して高くDNAメチル化している領域には,Peg10遺伝子,Peg3遺伝子,Igf2r遺伝子を含む,多数のインプリンティング遺伝子が存在していた.このことから,Tet1はインプリンティング遺伝子を含む特定の領域のDNA脱メチル化に機能しており,Tet1が欠損することにより消去されずに残ったDNAメチル化は精子,そして,受精ののちの胚にまで伝わり発生に異常をきたすことがわかった.
Tet1父性ノックアウトマウスが非常に多様な表現型を現わすのは,Tet1ノックアウトマウスの精子のひとつひとつがそれぞれ異なるDNAメチル化の異常パターンをもつためである.たとえば,体細胞ではPeg10遺伝子の対立遺伝子特異的メチル化領域は母方の対立遺伝子のみがDNAメチル化されていて,父方の対立遺伝子はDNAメチル化をうけていない.野生型マウスあるいはTet1ヘテロノックアウトマウスでは始原生殖細胞においてこの領域がDNA脱メチル化され,精子まで低いDNAメチル化の状態が維持される.しかし,Tet1ノックアウトマウスの始原生殖細胞ではDNA脱メチル化の異常のため母方の対立遺伝子にDNAメチル化が残存したまま減数分裂にまで進行してしまい,その結果,2つのパターンの精子が出現することになる.すなわち,父方の対立遺伝子に由来するPeg10遺伝子の対立遺伝子特異的メチル化領域を受け継いだ精子はこの領域にはもともとDNAメチル化が存在しないため,次世代にも表現型は現われない.それに対し,母方の対立遺伝子に由来するPeg10遺伝子の対立遺伝子特異的メチル化領域を受け継いだ精子はこの領域にDNAメチル化が残存しているため,受精により両方の対立遺伝子がDNAメチル化されたマウスが生まれ,このマウスではPeg10遺伝子の発現は抑制される(図3).マウスのゲノムには100以上のインプリンティング遺伝子が報告されており,それぞれのもつ対立遺伝子に特異的なDNAメチル化のパターンが減数分裂において起こる相同組換えをつうじシャッフルされる.そのため,異なるDNAメチル化パターンをもつ精子が生まれ,次世代に多様な表現型をひき起こすものと予想された.
雌の生殖系列においてもTet1はインプリンティング遺伝子のDNAメチル化の消去に寄与しているかどうか検証するため,雌のTet1ノックアウトマウスと雄の野生型マウスとを交配してTet1母性ノックアウトマウスを作製した.Tet1父性ノックアウトとは対照的に,Tet1母性ノックアウトマウスの胚の胎盤は対照となるマウスと比較して有意に大きかった.興味深いことに,約25%のTet1母性ノックアウトマウスは胚の発生の途中で吸収されてしまっていたが,この異常な胚の胎盤だけは比較的正常に発生していた.このような異常な胚の胎盤では,Meg3遺伝子,Mirg遺伝子,Rasgrf1遺伝子など,父方の対立遺伝子がDNAメチル化をうける領域に存在する遺伝子の発現レベルが顕著に変化し,これらの領域のDNAメチル化が有意に上昇していた.これは,始原生殖細胞において消去されるべき父方の対立遺伝子におけるDNAメチル化が卵子に残存してしまっていることを示唆した.これらの結果から,Tet1は雌の生殖系列においてもインプリンティング遺伝子のDNAメチル化の消去に機能していることが示唆された.
最後に,この研究の結果の信頼性をさらに裏づけるため,過去にほかの研究室において樹立された別のTet1ノックアウトマウス系統6) を用いて追加実験を行った.これまでの結果と同様に,この系統を用いて作製したTet1父性ノックアウトマウスも新生仔の体の大きさに大きなばらつきを示し,胎盤の発生の異常,胎生致死,発育の異常など,さまざまな表現型が認められた.この系統から作製したTet1父性ノックアウトマウスの胚においても,Peg10遺伝子,Peg3遺伝子,Igf2r遺伝子などインプリンティング遺伝子の発現に異常がみられたことから,インプリンティング遺伝子のDNAメチル化の消去へのTet1の関与がさらに裏づけられた.
なぜTet1はインプリンティング遺伝子や減数分裂に関連する遺伝子のみを標的としてDNAを脱メチル化しているのだろうか? ゲノムワイドなDNA脱メチル化は2段階で行われている点に着目した3,7,8).始原生殖細胞ではゲノムの約70%が胎生9.5日よりまえにDNA脱メチル化される7)(初期DNA脱メチル化).このことはすなわち,Tet1の発現が上昇する胎生10.5日の始原生殖細胞において,すでにゲノムの大部分はDNA脱メチル化が完了していることを意味する.実際に,今回,RRBS法により解析した領域は胎生9.5日の始原生殖細胞においてほぼ完全にDNA脱メチル化していた.しかし,Tet1ノックアウトマウスの始原生殖細胞において高くDNAメチル化していた領域に限定して解析すると,胎生9.5~11.5日の始原生殖細胞においてもDNAメチル化は高く維持されていた.このことから,Tet1の標的となる領域のDNAメチル化は初期DNA脱メチル化においても選択的に維持されており,この選択的な維持の機構こそがTet1の標的となる領域を決定することが示唆された.
近年のゲノム研究の発展から始原生殖細胞におけるDNA脱メチル化の機構は急速に解明されてきた3,7).しかしじつは,酵素に依存する能動的なDNA脱メチル化の存在およびその生理的な意義を決定づける報告はほとんどなかった.この研究の以前にも,AidやTdgなどがインプリンティング遺伝子のDNAメチル化の消去にかかわるという報告はあったが9,10),いずれも生殖細胞のゲノムワイドなDNAメチル化あるいは次世代への影響などは検証されておらず,決定的な証拠は示されていなかった.始原生殖細胞におけるDNAメチル化,精子におけるDNAメチル化,そして,次世代のマウスにおけるインプリンティング遺伝子のDNAメチル化,発現の異常,発生の異常を含む表現型,という一連の証拠を示してインプリンティング遺伝子のDNAメチル化の消去における責任タンパク質を同定したのは,この研究が世界ではじめてである.非常に競争の激しいテーマではあったが,ひとつひとつ証拠を積みあげていくのは未踏峰を一歩一歩登りつめていく過程に似て,その過程自体を非常に楽しむことができた.
この研究の結果から,ゲノムインプリンティングの消去はTet1に依存しており,その異常はつぎの世代まで伝わってしまうことが明らかになったことで,始原生殖細胞における能動的なDNA脱メチル化の重要性が証明された.しかし,このようなリスクがあるにもかかわらず,なぜインプリンティング遺伝子は初期のゲノムワイドなDNA脱メチル化においても消去されず維持されているのだろうか? この疑問こそが始原生殖細胞におけるゲノムリプログラミングの意義を明かす鍵であると考え,研究を継続している.
略歴:2010年 京都大学大学院医学研究科 修了,同年 京都大学再生医科学研究所 研究員,2011年 米国North Carolina大学Chapel Hill校 研究員を経て,2012年より米国Harvard Medical School研究員.
研究テーマ:生殖細胞および体細胞におけるリプログラミングの分子機構.
関心事:ああ後期脱メチル化遺伝子よ,どうしてあなたは初期脱メチル化から守られているの?
© 2013 山口 新平 Licensed under CC 表示 2.1 日本
(米国Harvard Medical School,Boston Children’s Hospital)
email:山口新平
DOI: 10.7875/first.author.2013.164
Role of Tet1 in erasure of genomic imprinting.
Shinpei Yamaguchi, Li Shen, Yuting Liu, Damian Sendler, Yi Zhang
Nature, 504, 460-464 (2013)
要 約
ゲノムインプリンティングは対立遺伝子に特異的な遺伝子発現の様式であり,親の世代の精子および卵子それぞれに異なるパターンで樹立されたDNAのメチル化により制御される.精子あるいは卵子に特異的なDNAメチル化パターンの形成にさきだち,始原生殖細胞において体細胞型のDNAメチル化パターンの消去が起こるが,その分子機構はほとんど解明されていなかった.この研究において,筆者らは,5-メチル化シトシンに特異的なジオキシゲナーゼTet1が始原生殖細胞におけるインプリンティング遺伝子のDNAメチル化を消去するタンパク質であることを示した.Tet1を欠損した雄のマウスと野生型の雌のマウスとの交配により得られたマウスは,胎生致死などそれぞれの個体で異なる多様な表現型を示した.Tet1ノックアウトマウスにおいては,本来は始原生殖細胞において消去されるべきインプリンティング遺伝子のDNAメチル化が消去されず,精子でも高くDNAメチル化した状態で維持されていた.親の世代から残存したDNAメチル化はつぎの世代に伝わり,インプリンティング遺伝子の発現の異常およびさまざまな表現型をひき起こしていた.以上の結果から,Tet1がインプリンティング遺伝子のDNAメチル化の脱メチル化に重要な役割をはたしていることが証明された.
はじめに
われわれ哺乳類では,ほとんどの遺伝子は父母それぞれに由来する2つの対立遺伝子から同じレベルで発現している.しかし,インプリンティング遺伝子は,父親(精子)由来あるいは母親(卵子)由来のどちらかの対立遺伝子からのみ発現するという特徴的な発現パターンを示す.この対立遺伝子に特異的な発現は,おもに対立遺伝子特異的メチル化領域(differentially methylated region:DMR)におけるDNAメチル化のパターンにより制御されている1).雄では胎仔期から新生仔期の精原細胞において,雌では出生ののちの卵母細胞において,精子あるいは卵子に特異的なDNAメチル化パターンが形成される.しかし,マウスの精子および卵子はもともと体細胞型のDNAメチル化パターンをもつ始原生殖細胞から分化する.そのため,精子あるいは卵子に特異的なDNAメチル化パターンが形成されるまえに,この体細胞型のDNAメチル化パターンを消去することが必要である.胎生8.0~13.5日の始原生殖細胞において起こるゲノムリプログラミングをつうじほとんどすべてのゲノムはDNA脱メチル化することがわかっていたが1),インプリンティング遺伝子のDNAメチル化の消去における責任タンパク質および分子機構は長いあいだ不明であった(図1).
ゲノムのリセットともいえる重要なイベントの機構が長らく不明だったのは,哺乳類におけるDNA脱メチル化の機構がほとんどわかっていなかったことが原因であった.しかし,2009年,Tetファミリーが5-メチル化シトシンを特異的に酸化する活性をもつことが発見され,この分野の研究が急速に進んだ2,3).筆者らは,Tetファミリーのうちゲノムリプログラミングの起こる時期の始原生殖細胞において特異的に発現するTet1に注目し,Tet1が減数分裂に関連する遺伝子におけるDNA脱メチル化に重要であることを発見した4)(新着論文レビュー でも掲載).はたして,Tet1はインプリンティング遺伝子のDNAメチル化の消去にもはたらいているのだろうか,それが,この研究の出発点であった.
1.父親におけるTet1の欠損により次世代は多様な表現型を示す
インプリンティング遺伝子のDNAメチル化の消去の異常は精子に残存するDNAメチル化として受け継がれ,残存したDNAメチル化は受精ののちのつぎの世代に影響を及ぼすであろうと仮説をたてた.そこで,雄のTet1ノックアウトマウスと雌の野生型マウスとを交配し,次世代としてTet1父性ノックアウトマウスを得た.対照となるマウスとしては,雄のTet1ヘテロノックアウトマウスと雌の野生型マウスとの交配により得たマウスを用いた.Tet1父性ノックアウトマウスはTet1遺伝子についてヘテロであり,対照となるマウスの半分とは同一である.にもかかわらず,Tet1父性ノックアウトマウスのみに非常に多様な表現型が認められた.Tet1父性ノックアウトマウスは生まれたときの体の大きさに非常に大きなばらつきがあり,新生仔の体重は,対照となるマウスでは全個体の約70%が1.2~1.3 gにおさまるのに対し,Tet1父性ノックアウトマウスは1.1 g以下あるいは1.4 g以上の個体が70%以上をしめた.Tet1父性ノックアウトマウスの胎盤は対照となるマウスと比較して顕著に小さく,その大きさは体の大きさと正の相関を示したことから,少なくとも,体の大きさの減少は胎盤の異常が原因であることが示唆された.さらに,Tet1父性ノックアウトマウスの新生仔はその半分が出生から3日以内に死亡した.生き残ったマウスも大きなばらつきをもちながら発育に遅延を示し,3週齢の時点においてTet1父性ノックアウトマウスのうち約半数の個体は対照となるマウスに比べ30~70%の体重しかなかった.
このような発育の異常にくわえ,Tet1父性ノックアウトマウスは生まれてくる個体の数がすでに少ないことがわかった.野生型マウスとTet1ヘテロノックアウトマウスとの交配では平均7.5匹の仔が得られるのに対し,野生型マウスとTet1ノックアウトマウスとの交配では平均4匹しか生まれてこなかった.そこで,胎生13.5日において解剖を行ったところ,Tet1父性ノックアウトでは約35%の胚がすでに死亡し吸収されていることがわかった.胎生10.5日では約35%の胚が発生に明らかな異常を示していた.胎生9.5日では胚に形態的な異常はみつからなかったが,約35%の胚で胎盤に異常があった.組織学的な解析の結果,これらの胎盤では絨毛膜板の伸展が認められず,ラビリンスの形成も認められなかった.
以上の結果から,Tet1父性ノックアウトマウスの一部は,胚性致死,胎仔および胎盤の発生の異常,新生仔致死,出生ののちの発育の異常という多様な表現型を示すことがわかった(図2).
2.Tet1父性ノックアウトマウスはインプリンティング遺伝子のDNAメチル化および遺伝子の発現に異常を示す
インプリンティング遺伝子の異常は胎盤あるいは胚の発生において表現型を示すことが知られている1).胎生致死の表現型が父方の対立遺伝子に特異的に発現するインプリンティング遺伝子Peg10遺伝子のノックアウトマウスと類似していたことから,胎生9.5日の胚におけるPeg10遺伝子の発現を解析した.その結果,異常な胎盤をもつ胚ではPeg10遺伝子の発現が完全に抑制されていることがわかった.胎生9.5日の胚における遺伝子発現プロファイルをRNAシークエンシング法により解析すると,対照となるマウスの胚において発現の認められた81個のインプリンティング遺伝子のうち,11~46個のインプリンティング遺伝子がTet1父性ノックアウトマウスの胚において異常な発現を示していた.表現型と同様,インプリンティング遺伝子の発現異常のパターンにも胚ごとにばらつきがみられたが,共通の傾向として,父方の対立遺伝子より発現するインプリンティング遺伝子が抑制され,母方の対立遺伝子より発現するインプリンティング遺伝子が活性化していた.このことから,父方の対立遺伝子が母方の対立遺伝子の発現様式を獲得していることが示唆された.
Tet1父性ノックアウトマウスにおけるインプリンティング遺伝子の発現の異常がDNAメチル化の異常に起因するのかどうか明らかにするため,Peg10遺伝子の対立遺伝子特異的メチル化領域におけるDNAメチル化について解析した.対照となる胚では予想どおり約50%のDNAメチル化が認められたのに対し,Peg10遺伝子の発現が抑制されていたTet1父性ノックアウトマウスの胚ではその対立遺伝子特異的メチル化領域はほぼ100%がDNAメチル化していた.このことは,通常はDNAメチル化をうけない父方の対立遺伝子もDNAメチル化されていることを示した.Peg10遺伝子のほかにも,Igf2r遺伝子の発現が異常な胚,および,Impact遺伝子の発現が異常な胚では,それぞれの対立遺伝子特異的メチル化領域においてほぼ100%のDNAメチル化が認められた.
胎盤の発達の異常も胚と同様にインプリンティング遺伝子の発現の異常が原因なのかどうか明らかにするため,胎生19.5日の胎盤を解析した.その結果,異常に小さい胎盤においてはIgf2r遺伝子およびPeg3遺伝子の発現に異常が認められた.胚における結果と同様,Peg3遺伝子の発現が抑制されていた胎盤ではその対立遺伝子特異的メチル化領域におけるDNAメチル化が上昇していた.Peg3ノックアウトマウスは同様の胎盤の異常を示すことから,Tet1父性ノックアウトマウスの胚における胎盤の発達の異常もインプリンティング遺伝子の発現の異常が原因であることが示唆された.
これらの結果から,通常はDNAメチル化をうけない父方の対立遺伝子がDNAメチル化されることがTet1父性ノックアウトマウスにおけるインプリンティング遺伝子の発現異常の原因であり,発生の異常につながることが示唆された.
3.Tet1ノックアウトマウスの精子および始原生殖細胞においてはインプリンティング遺伝子におけるDNAメチル化が消去されない
Tet1父性ノックアウトマウスの母親は野生型であるため,その胚および胎盤において認められたインプリンティング遺伝子の発現の異常は,父親であるTet1ノックアウトマウスの精子における異常なDNAメチル化が直接の原因だと予想された.もしTet1がインプリンティング遺伝子のDNAメチル化の消去に機能しているなら,Tet1ノックアウトマウスの精子においてみられる異常はゲノムリプログラミングの起こった直後の始原生殖細胞においてすでに現われているはずである.この仮説を検証するため,Tet1ノックアウトマウスの精子およびゲノムリプログラミングの直後である胎生13.5日の雄の始原生殖細胞の両方において,RRBS(reduced representation bisulfite sequencing)法5) を用いてゲノムワイドなDNAメチル化パターンを解析した.筆者らの過去の報告とは矛盾せず4),ゲノム全体的なDNAメチル化のパターンは精子でも始原生殖細胞でも大きな異常は認められなかった.これは,Tet1ノックアウトマウスにおいてもゲノムワイドなDNA脱メチル化やDNA再メチル化が異常なく起こっていることを示唆した.しかし,詳細な解析の結果,Tet1ノックアウトマウスの精子では493,始原生殖細胞では9665の高くDNAメチル化した領域が同定された.そして予想どおり,精子および始原生殖細胞の両方で共通して高くDNAメチル化している領域には,Peg10遺伝子,Peg3遺伝子,Igf2r遺伝子を含む,多数のインプリンティング遺伝子が存在していた.このことから,Tet1はインプリンティング遺伝子を含む特定の領域のDNA脱メチル化に機能しており,Tet1が欠損することにより消去されずに残ったDNAメチル化は精子,そして,受精ののちの胚にまで伝わり発生に異常をきたすことがわかった.
Tet1父性ノックアウトマウスが非常に多様な表現型を現わすのは,Tet1ノックアウトマウスの精子のひとつひとつがそれぞれ異なるDNAメチル化の異常パターンをもつためである.たとえば,体細胞ではPeg10遺伝子の対立遺伝子特異的メチル化領域は母方の対立遺伝子のみがDNAメチル化されていて,父方の対立遺伝子はDNAメチル化をうけていない.野生型マウスあるいはTet1ヘテロノックアウトマウスでは始原生殖細胞においてこの領域がDNA脱メチル化され,精子まで低いDNAメチル化の状態が維持される.しかし,Tet1ノックアウトマウスの始原生殖細胞ではDNA脱メチル化の異常のため母方の対立遺伝子にDNAメチル化が残存したまま減数分裂にまで進行してしまい,その結果,2つのパターンの精子が出現することになる.すなわち,父方の対立遺伝子に由来するPeg10遺伝子の対立遺伝子特異的メチル化領域を受け継いだ精子はこの領域にはもともとDNAメチル化が存在しないため,次世代にも表現型は現われない.それに対し,母方の対立遺伝子に由来するPeg10遺伝子の対立遺伝子特異的メチル化領域を受け継いだ精子はこの領域にDNAメチル化が残存しているため,受精により両方の対立遺伝子がDNAメチル化されたマウスが生まれ,このマウスではPeg10遺伝子の発現は抑制される(図3).マウスのゲノムには100以上のインプリンティング遺伝子が報告されており,それぞれのもつ対立遺伝子に特異的なDNAメチル化のパターンが減数分裂において起こる相同組換えをつうじシャッフルされる.そのため,異なるDNAメチル化パターンをもつ精子が生まれ,次世代に多様な表現型をひき起こすものと予想された.
4.Tet1母性ノックアウトマウスもインプリンティング遺伝子のDNAメチル化および遺伝子の発現に異常を示す
雌の生殖系列においてもTet1はインプリンティング遺伝子のDNAメチル化の消去に寄与しているかどうか検証するため,雌のTet1ノックアウトマウスと雄の野生型マウスとを交配してTet1母性ノックアウトマウスを作製した.Tet1父性ノックアウトとは対照的に,Tet1母性ノックアウトマウスの胚の胎盤は対照となるマウスと比較して有意に大きかった.興味深いことに,約25%のTet1母性ノックアウトマウスは胚の発生の途中で吸収されてしまっていたが,この異常な胚の胎盤だけは比較的正常に発生していた.このような異常な胚の胎盤では,Meg3遺伝子,Mirg遺伝子,Rasgrf1遺伝子など,父方の対立遺伝子がDNAメチル化をうける領域に存在する遺伝子の発現レベルが顕著に変化し,これらの領域のDNAメチル化が有意に上昇していた.これは,始原生殖細胞において消去されるべき父方の対立遺伝子におけるDNAメチル化が卵子に残存してしまっていることを示唆した.これらの結果から,Tet1は雌の生殖系列においてもインプリンティング遺伝子のDNAメチル化の消去に機能していることが示唆された.
最後に,この研究の結果の信頼性をさらに裏づけるため,過去にほかの研究室において樹立された別のTet1ノックアウトマウス系統6) を用いて追加実験を行った.これまでの結果と同様に,この系統を用いて作製したTet1父性ノックアウトマウスも新生仔の体の大きさに大きなばらつきを示し,胎盤の発生の異常,胎生致死,発育の異常など,さまざまな表現型が認められた.この系統から作製したTet1父性ノックアウトマウスの胚においても,Peg10遺伝子,Peg3遺伝子,Igf2r遺伝子などインプリンティング遺伝子の発現に異常がみられたことから,インプリンティング遺伝子のDNAメチル化の消去へのTet1の関与がさらに裏づけられた.
5.Tet1の標的となる領域は初期DNA脱メチル化において選択的に維持されている
なぜTet1はインプリンティング遺伝子や減数分裂に関連する遺伝子のみを標的としてDNAを脱メチル化しているのだろうか? ゲノムワイドなDNA脱メチル化は2段階で行われている点に着目した3,7,8).始原生殖細胞ではゲノムの約70%が胎生9.5日よりまえにDNA脱メチル化される7)(初期DNA脱メチル化).このことはすなわち,Tet1の発現が上昇する胎生10.5日の始原生殖細胞において,すでにゲノムの大部分はDNA脱メチル化が完了していることを意味する.実際に,今回,RRBS法により解析した領域は胎生9.5日の始原生殖細胞においてほぼ完全にDNA脱メチル化していた.しかし,Tet1ノックアウトマウスの始原生殖細胞において高くDNAメチル化していた領域に限定して解析すると,胎生9.5~11.5日の始原生殖細胞においてもDNAメチル化は高く維持されていた.このことから,Tet1の標的となる領域のDNAメチル化は初期DNA脱メチル化においても選択的に維持されており,この選択的な維持の機構こそがTet1の標的となる領域を決定することが示唆された.
おわりに
近年のゲノム研究の発展から始原生殖細胞におけるDNA脱メチル化の機構は急速に解明されてきた3,7).しかしじつは,酵素に依存する能動的なDNA脱メチル化の存在およびその生理的な意義を決定づける報告はほとんどなかった.この研究の以前にも,AidやTdgなどがインプリンティング遺伝子のDNAメチル化の消去にかかわるという報告はあったが9,10),いずれも生殖細胞のゲノムワイドなDNAメチル化あるいは次世代への影響などは検証されておらず,決定的な証拠は示されていなかった.始原生殖細胞におけるDNAメチル化,精子におけるDNAメチル化,そして,次世代のマウスにおけるインプリンティング遺伝子のDNAメチル化,発現の異常,発生の異常を含む表現型,という一連の証拠を示してインプリンティング遺伝子のDNAメチル化の消去における責任タンパク質を同定したのは,この研究が世界ではじめてである.非常に競争の激しいテーマではあったが,ひとつひとつ証拠を積みあげていくのは未踏峰を一歩一歩登りつめていく過程に似て,その過程自体を非常に楽しむことができた.
この研究の結果から,ゲノムインプリンティングの消去はTet1に依存しており,その異常はつぎの世代まで伝わってしまうことが明らかになったことで,始原生殖細胞における能動的なDNA脱メチル化の重要性が証明された.しかし,このようなリスクがあるにもかかわらず,なぜインプリンティング遺伝子は初期のゲノムワイドなDNA脱メチル化においても消去されず維持されているのだろうか? この疑問こそが始原生殖細胞におけるゲノムリプログラミングの意義を明かす鍵であると考え,研究を継続している.
文 献
- Bartolomei, M. S. & Ferguson-Smith, A. C.: Mammalian genomic imprinting. Cold Spring Harb. Perspect. Biol., 3, a002592 (2011)[PubMed]
- Tahiliani, M., Koh, K. P., Shen, Y. et al.: Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science, 324, 930-935 (2009)[PubMed]
- Kohli, R. M. & Zhang, Y.: TET enzymes, TDG and the dynamics of DNA demethylation. Nature, 502, 472-479 (2013)[PubMed]
- Yamaguchi, S., Hong, K., Liu, R. et al.: Tet1 controls meiosis by regulating meiotic gene expression. Nature 492, 443-447 (2012)[PubMed] [新着論文レビュー]
- Meissner, A., Gnirke, A., Bell, G. W. et al.: Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis. Nucleic Acids Res., 33, 5868-5877 (2005)[PubMed]
- Dawlaty, M. M., Ganz, K., Powell, B. E. et al.: Tet1 is dispensable for maintaining pluripotency and its loss is compatible with embryonic and postnatal development. Cell Stem Cell, 9, 166-175 (2011)[PubMed]
- Seisenberger, S., Andrews, S., Krueger, F. et al.: The dynamics of genome-wide DNA methylation reprogramming in mouse primordial germ cells. Mol. Cell, 48, 849-862 (2012)[PubMed]
- Yamaguchi, S., Hong, K., Liu, R. et al.: Dynamics of 5-methylcytosine and 5-hydroxymethylcytosine during germ cell reprogramming. Cell Res., 23, 329-339 (2013)[PubMed]
- Popp, C., Dean, W., Feng, S. et al.: Genome-wide erasure of DNA methylation in mouse primordial germ cells is affected by AID deficiency. Nature, 463, 1101-1105 (2010)[PubMed]
- Cortellino, S., Xu, J., Sannai, M. et al.: Thymine DNA glycosylase is essential for active DNA demethylation by linked deamination-base excision repair. Cell, 146, 67-79 (2011)[PubMed]
著者プロフィール
略歴:2010年 京都大学大学院医学研究科 修了,同年 京都大学再生医科学研究所 研究員,2011年 米国North Carolina大学Chapel Hill校 研究員を経て,2012年より米国Harvard Medical School研究員.
研究テーマ:生殖細胞および体細胞におけるリプログラミングの分子機構.
関心事:ああ後期脱メチル化遺伝子よ,どうしてあなたは初期脱メチル化から守られているの?
© 2013 山口 新平 Licensed under CC 表示 2.1 日本