単一の生細胞におけるプロテオームとトランスクリプトームとを単一分子検出感度で定量化する
谷口 雄一
(米国Harvard大学Department of Chemistry and Chemical Biology)
email:谷口雄一
DOI: 10.7875/first.author.2010.013
Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells.
Yuichi Taniguchi, Paul J. Choi, Gene-Wei Li, Huiyi Chen, Mohan Babu, Jeremy Hearn, Andrew Emili, X. Sunney Xie
Science, 329, 533-538(2010)
単一細胞のレベルでは内在するmRNA数とタンパク質数とがたえず乱雑に変動している.このため,ひとつひとつの細胞は,たとえ同じゲノムをもっていても,それぞれが個性的な振る舞いを示す.筆者らは,単一細胞内におけるmRNAとタンパク質の発現プロファイリングを単一分子検出レベルの感度で行うことにより,単一細胞のもつ特性の乱雑さをシステムワイドで定量化し,そこにあるゲノム共通の法則性を明らかにした.そのために,蛍光タンパク質遺伝子をそれぞれの遺伝子のC末端に結合させた大腸菌ライブラリーを1000株以上にわたって作製し,マイクロチップ上で単一分子感度での計測をシステマティックに行うことにより,それぞれの遺伝子におけるmRNAとタンパク質の絶対個数,ばらつき,細胞内局在などの情報を網羅的に取得した.その結果,全体の98%の遺伝子は発現するタンパク質数の分布において特定の共通構造をもっており,それらの分布構造の大きさは量子ノイズやグローバル因子による極限をもつことが判明した.
生物は内在するゲノムから数千から数万にわたる種類のタンパク質を生み出すことによって生命活動を行っている.近年,これらの膨大な生物情報を網羅的に取得し,生物を包括的に理解しようとする研究が急速に進展している.2003年にヒトゲノムが完全解読され,現在ではゲノム解読の高速化・低価格化が注目を集める一方で,より直接的に機能レベルの情報を取得する手法として,ゲノム(DNA)の発現産物であるmRNAやタンパク質の発現量を網羅的に調べるトランスクリプトミクスやプロテオミクスに関する研究開発に関心が集まっている.cDNAマイクロアレイ法やRNA-seq法,質量分析法などの技術開発によって発現産物の量をより高感度に探ることが可能となってきているが,いまだ単一分子検出レベルの高感度の実現にはいたっていない.
その一方で,近年のレーザー蛍光顕微鏡技術の発展により,単一細胞内で起こる遺伝子発現を単一分子レベルで検出することが可能になってきた1,2).筆者らは今回,こうした単一分子計測技術を応用することにより,モデル生物である大腸菌(Escherichia coli)について,単一分子・単一細胞レベルでのmRNAとタンパク質の発現プロファイリングをはじめて実現した.
単一分子・単一細胞プロファイリングにおいては,ひとつひとつの細胞に存在するmRNAとタンパク質の絶対個数がそれぞれ決定される.細胞では1つあるいは2つの遺伝子座から確率論的にmRNA,そして,タンパク質の発現が行われているので,ひとつひとつの細胞は同じゲノムをもっていても,内在するmRNAとタンパク質の個数のうちわけには大きな多様性があり,さらにこれは,時々刻々と変化している.つまり,細胞は確率的な遺伝子発現を利用して,表現型の異なる細胞をたえず自発的に生み出しているといえる.こうした乱雑さは生物の大きな特徴であり,これを利用することで細胞の分化や異質化を誘導したり,環境変化に対する生物種の適応度を高めたりしていると考えられている3,4).この研究では,大腸菌について個体レベルでの乱雑さをプロテオームレベルおよびトランスクリプトームレベルで定量化し,そのゲノムに共通する原理を探ることをめざした.
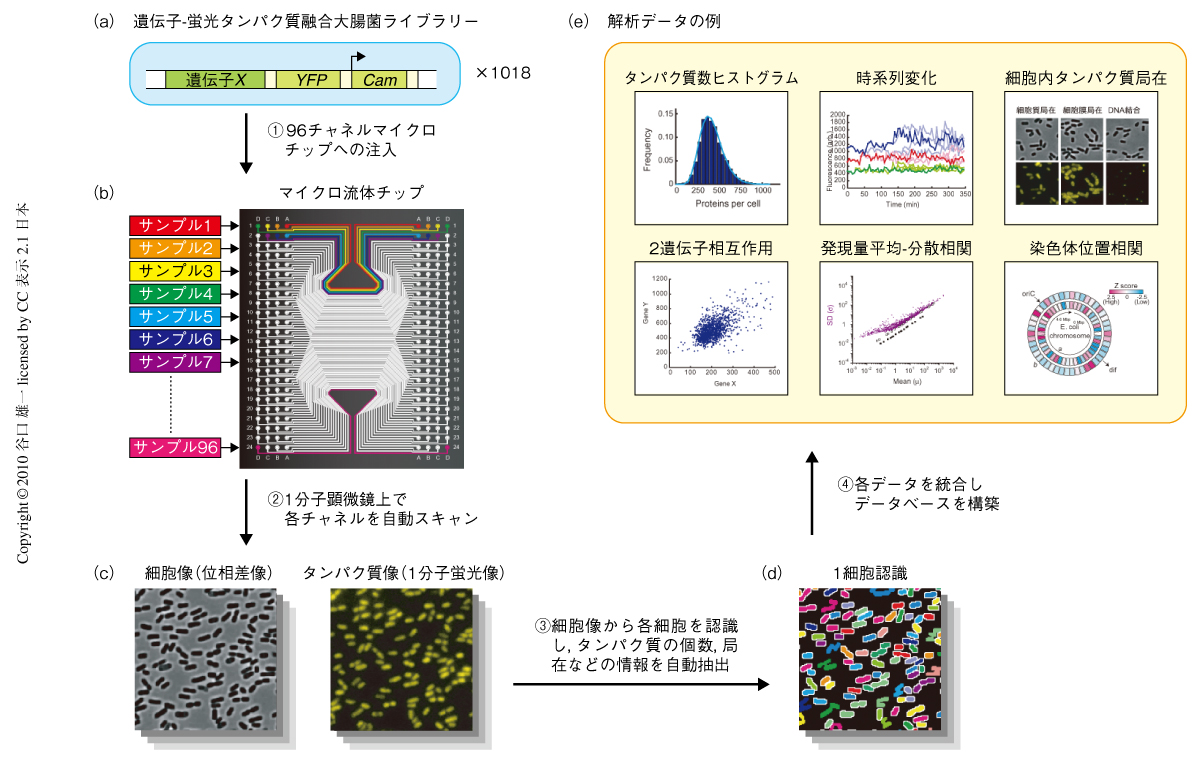
1分子・1細胞レベルで大腸菌がタンパク質を発現するようすを調べるため,大腸菌染色体内のそれぞれの遺伝子に黄色蛍光タンパク質Venusの遺伝子を導入した大腸菌株ライブラリーを構築した(図1a).このライブラリーは,大腸菌のそれぞれの遺伝子に対応した計1018種類の大腸菌株により構成されており,おのおのの株においては対応する遺伝子のC末端に蛍光タンパク質の遺伝子が挿入されている.遺伝子発現と連動して生じる蛍光タンパク質の蛍光をレーザー顕微鏡により単一分子感度でとらえることによって,遺伝子発現の単一分子観測が可能となる1).
ライブラリーの作製にあたっては,共同研究者であるカナダToronto大学のEmili教授のグループが2006年に作製した,SPA(sequential peptide affinity)ライブラリーを利用した5).このライブラリーでは大腸菌のそれぞれの遺伝子のC末端にタンパク質精製用のSPAタグが挿入されていたが,このタグをλ-Red相同組換え法を用いてVenusの遺伝子に置き換える方法をとることによって,ユニバーサルなプライマーを用いて廉価かつ効率的にライブラリーの作製を行うことができた.
膨大な数のライブラリー株をレーザー顕微鏡によりハイスループットで解析するため,ソフトリソグラフィー技術を用いてシリコン成型したマイクロ流体チップを開発した6)(図1b).このチップは平行に並んだ96のサンプル流路により構成されており,マルチチャネルピペッターを用いてそれぞれに異なるライブラリー株を注入することによって,96のライブラリー株を並列的に2次元配列することができる.チップの底面は薄型カバーガラスになっているためレーザー顕微鏡による高開口数での観察が可能であり,3次元電動ステージを用いてスキャンすることにより多サンプル連続解析が可能となった.チップの3次元スキャン,自動フォーカス,光路の切替え,画像撮影,画像分析など,解析の一連の流れをコンピューターで完全自動化することにより,それぞれのライブラリー株あたり,25秒間に平均4000個の細胞の解析を行うことができた.
解析により得られるライブラリー株の位相差像と蛍光像の代表例を表す(図1c).それぞれの細胞におけるタンパク質発現量が蛍光量として検出できると同時に,タンパク質の細胞内局在(膜局在,細胞質局在,DNA局在など)を観察することができた.それぞれの細胞に内在している蛍光に対して単一蛍光分子による規格化を行い,さらに,細胞の自家蛍光による影響を差し引くことによって,それぞれの細胞におけるタンパク質発現数の分布を決定した(図1d).同時に,画像解析によって蛍光分子の細胞内局在(細胞質局在と細胞膜局在との比,点状の局在)をスコア化した(図1e).
この結果,大腸菌のそれぞれの遺伝子の1細胞あたりの平均発現量は,10-1個/細胞から104個/細胞まで,5オーダーにわたって幅広く分布していることがわかった.必須遺伝子の大半が10個/細胞以上の高い発現レベルを示したのに対し,全体ではおおよそ半数の遺伝子が10個/細胞以下の発現レベルを示した.低発現を示すタンパク質のなかには実際に機能していることが示されているものも多く存在しており,これらのタンパク質は10個以下の低分子数でも細胞内で十分に機能することがわかった.このことは,単一細胞レベルの微生物学において,単一分子感度の実験が本質的でありうることを示唆する.
それぞれの細胞におけるタンパク質数の分布を調べたところ,一般に,低発現数を示すタンパク質の分布は単調減少関数,高発現数を示すタンパク質の分布はピークをもった関数になっていた.さまざまなモデルを用いてフィッティングを行い,すべての遺伝子の分布を一般的に記述できる最良の関数を探した結果,1018遺伝子のうち1009遺伝子をガンマ分布によって記述できることをみつけた.大腸菌はガンマ分布というゲノムに共通の構造にそってプロテオームの多様性を生み出しており,その分布はガンマ分布のもつ2つのパラメーターによって一般的に記述できることが明らかになった.
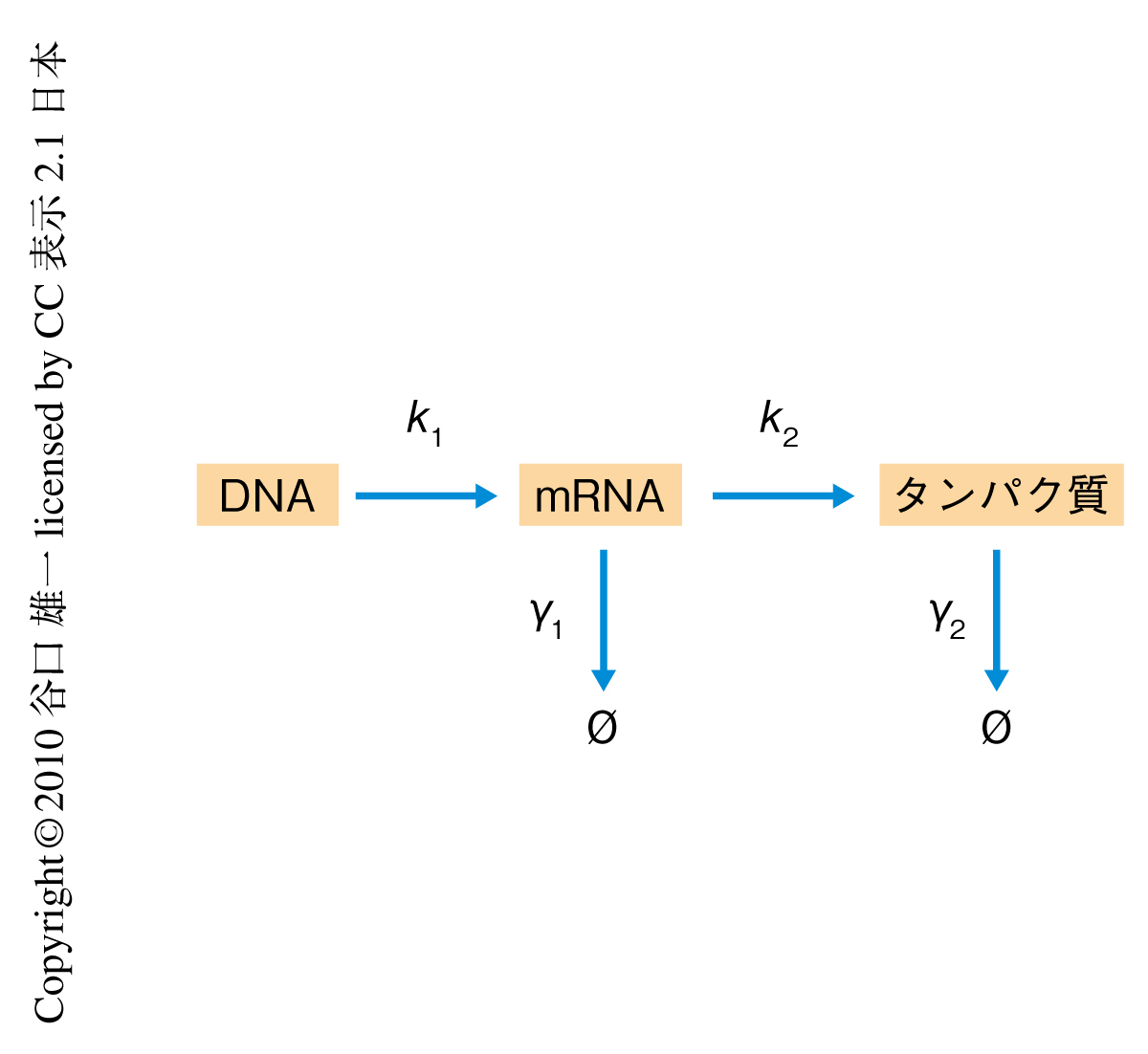
このガンマ分布は,mRNAの転写とタンパク質の翻訳,mRNAの分解とタンパク質の分解が,それぞれ確率的に起こると仮定した場合のタンパク質数の分布に等しい7)(図2).これはつまり,タンパク質数の分布がセントラルドグマの過程の確率的な特性により決定づけられることを示唆している.そこで以降,このガンマ分布を軸として,細胞のタンパク質量を正しく記述するためのモデルをさらに検証した.
タンパク質数の分布のばらつきの大きさ,または,ノイズ(発現数の標準偏差の2乗と発現数の平均の2乗の比と定義される)は,個々の細胞におけるタンパク質量の多様性を表す重要なパラメーターである3).このノイズをそれぞれの遺伝子について求めたところ,つぎに示すような発現量の大きさに応じた二相性のあることをみつけた.
平均発現数が10分子以下の遺伝子は,ほぼすべてがポアソンノイズを下限とする,発現数と反比例した量のノイズをもっていた.このポアソンノイズは一種の量子ノイズであり,遺伝子発現が純粋にランダムに(すなわち,ポアソン過程で)行われた場合のノイズ量を表している.つまり今回の結果は,タンパク質発現のノイズをポアソンノイズ以下に抑えるような遺伝子制御機構は存在しないことを示唆する.実際のノイズがポアソンノイズを上まわるということは,遺伝子の発現が準ランダムに行われていることを表している.実際,ひとつひとつのタンパク質の発現は純粋なランダムではなく,mRNAの発現とともに突発的に複数のタンパク質の発現(バースト)が起こり,mRNAの分解と同時にタンパク質の発現がとまる,といったかたちでバースト的に行われることが報告されている1).筆者らは,複数のライブラリー株をリアルタイム計測することでバーストの観測を行うことにより,バーストの頻度と大きさが細胞集団計測で得られるノイズの大きさに合致することをみつけた.これはつまり,ノイズの大きさがmRNAバーストの性質により決定されていることを表している.
一方で,平均発現数が10分子以上の遺伝子は,ポアソンノイズとは異なる,発現数に依存しない一様なノイズ極限をもっていた.すべての遺伝子はこのノイズ極限よりも大きなノイズをもっていることから,大腸菌に発現するタンパク質は必ず一定割合(30%)以上のノイズをもっていることが示された.
この一様なノイズ極限の起源を調べるため,高発現を示す複数のライブラリー株を無作為に抽出し,これらのタンパク質量の時間的な変化をタイムラプス観測により調べた.高発現タンパク質が一定の確率でランダムに発現している場合,ひとつひとつの細胞に存在するタンパク質の数は短い時間スケールで乱雑に変動し,数分もすればもとあったタンパク質レベルが初期化され,それぞれがまったく別のタンパク質レベルとなるはずである8).これに反して,今回のライブラリー株ではひとつひとつの細胞でのタンパク質レベルの大小が十数世代(1000分間以上)にわたって維持されていることが観測された.これはつまり,細胞ひとつひとつが互いに異なる細胞状態をもっており,さらに,この状態が何世代にもわたって“記憶”されていることを示している.
ノイズ解析で観測された一様なノイズ極限は,こうした細胞状態の不均一性により説明できることがみつけられた.セントラルドグマの過程(図2)において,それぞれの細胞が異なる速度定数をもつとする.この場合,ノイズの値には,発現量に反比例した固有成分にくわえて,発現量に依存しない定数成分が現われるようになる.この定数成分が高発現タンパク質において優勢になることから,一様なノイズ極限が観測されたといえる.つまり,一様なノイズ極限は,細胞内で起こるタンパク質発現のランダム性からではなく,それぞれの細胞の特性のばらつき(たとえば,ポリメラーゼやリボソームの数の不均一性など)から生じたとすることにより説明できた.
さらに,この一様なノイズ極限がポリメラーゼやリボソームなどすべての遺伝子の発現にかかわるグローバルな因子により生み出されていることを突き止めた.これを示すために,複数の2遺伝子の組合せを無作為に抽出し,異なる蛍光タンパク質でラベル化することによって1つの細胞における2つの遺伝子の発現レベルにおける相関関係を調べた.その結果,どの2遺伝子の組合せに関しても正の相関が観察され,細胞状態に応じてすべての遺伝子の発現の大小がひとまとめに制御されていることがわかった.相関解析からこうした“グローバルノイズ”の量は30%と求まり,一様なノイズ極限の値と一致した.
つぎに,タンパク質発現の中間産物であるmRNAの量を単一分子感度・単一細胞分解能でプロファイリングすることを試みた.そのために,蛍光in situハイブリダイゼーション(FISH)法を用いて,ライブラリーの黄色蛍光タンパク質のmRNAに赤色蛍光ヌクレオチドを選択的にハイブリダイゼーションした.この方法ではすべてのライブラリーに対して同じプローブを用いるため,遺伝子ごとのバイアスがほとんどない.レーザー顕微鏡を用いて細胞内の蛍光ヌクレオチドを数えることにより,mRNA数の決定を行った.
mRNA数のノイズを調べた結果,タンパク質の場合とは異なり,ポアソンノイズにもとづくノイズ極限だけがみられた.これは,mRNAの数は少ないためにポアソンノイズが大きくなり,一様なノイズ極限の影響が現われなくなったためであると考えられた.
赤色蛍光ヌクレオチドと黄色蛍光タンパク質の蛍光スペクトルが異なることを利用して,単一細胞におけるmRNA数とタンパク質数を同時に測定しその相関を調べた.137の遺伝子に対して測定を行ったところ,どの遺伝子においてもこれらのあいだには強い相関はなかった.つまり,単一細胞においては内在するmRNA数とタンパク質数とのあいだには相関のないことが判明した.
この非相関性のおもな理由としてmRNAの分解時間の速さがあげられる.RNA-seq法を用いてmRNAの分解時定数を調べたところ,数分以下であった.これに対し,ほとんどのタンパク質の分解時定数は数時間以上であり,タンパク質数の減衰はおもに細胞分裂による希釈効果により起こることが知られている9).したがって,mRNAの数は数分以内に起こった現象を反映するのに対し,タンパク質の数は細胞分裂の時間スケール(150分)のあいだで積み重なった現象を反映することになり,これらの数のあいだに不一致が起こるものと考えられる.
単一細胞におけるmRNA量の高ノイズ性を示す今回の結果は,1細胞レベルでのトランスクリプトーム解析に対してひとつの警告をあたえるものであり,同時に,プロテオーム解析の必要性を表している.
得られた1分子・1細胞レベルでの発現特性が生物学的な機能とどのように相関しているかを統計的に調べた.たとえば,タンパク質発現平均数はコドン使用頻度の指標であるCAI(codon adaptation index)と正の相関をもつのに対し,GC含量やmRNAの分解時間,染色体上の位置との相関はなかった.また,膜トランスポーターの遺伝子は高い膜局在性,転写因子は高い点局在性を示した.また,短い遺伝子は高いタンパク質発現を示すことや,リーディング鎖にある遺伝子からの転写はラギング鎖にある遺伝子からの転写よりも多いことがわかった.さらに,大腸菌のノイズは出芽酵母のノイズと比べ高いことも明らかになった10).
ここで示したのはほんの一例であり,相関解析の全データ,それぞれの遺伝子情報の全データは原著論文のSupporting Online Materialに掲載しているので,参考にしてほしい.
この研究で構築した単一分子・単一細胞プロファイリング技術は,複雑な細胞システムを素子である1分子レベルから理解することを可能とするものであり,1分子・1細胞生物学とシステム生物学とをつなぐ架け橋となりうる.以下,従来のプロファイリングの手法と比べた場合のアドバンテージをまとめる.
1)単一細胞内における遺伝子発現の絶対個数がわかる.
2)細胞を生きたまま解析でき,リアルタイムでの解析が可能.
3)細胞ごとの遺伝子発現量の確率論的なばらつきを解析できる.
4)ごくわずかな割合で存在する異常細胞を発見できる.
5)シグナル増幅が不要であり,遺伝子によるバイアスがきわめて少ない.
6)単一細胞内での2遺伝子の相互作用解析が可能.
7)細胞内におけるタンパク質局在を決定できる.
これらのアドバンテージを利用することで,細胞ひとつひとつの分子数や細胞状態の違いを絶対感度でとらえることが可能となり,さまざまな生命現象をより精密に調べることが可能となる.この研究では,生物特有の性質である個体レベルでの生命活動の“乱雑さ”を直接とらえることを目的としてこの技術を利用し,その一般原理のひとつを明らかにしている.
この研究で得られた大腸菌の単一分子・単一細胞プロファイルは,分子・細胞相互の階層から生物をシステムとして理解するための包括的データリソースとして役立つとともに,生物のもつ乱雑性,多様性を理解するためのひとつの基礎になるものと期待される.
略歴:2006年 大阪大学大学院基礎工学研究科博士課程 修了,同年より米国Harvard大学 ポストドクトラルフェロー.
専門分野:生物物理学,ナノバイオロジー.
キーワード:1分子・1細胞生物学,システム生物学,プロテオミクス,超高感度顕微鏡技術,微細加工技術,生命反応の物理,生物ゆらぎ.
抱負:顕微鏡工学,マイクロ工学,遺伝子工学,コンピューター工学など,さまざまな分野にまたがるさまざまな要素技術を組み合わせて,生命を理解するための新しい画期的な技術をつくるのが仕事です.生物学,物理学,統計学などのあらゆる立場から生命活動の本質を理解し,人々の疾病克服,健康増進に役立てることが目標です.
© 2010 谷口 雄一 Licensed under CC 表示 2.1 日本
(米国Harvard大学Department of Chemistry and Chemical Biology)
email:谷口雄一
DOI: 10.7875/first.author.2010.013
Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells.
Yuichi Taniguchi, Paul J. Choi, Gene-Wei Li, Huiyi Chen, Mohan Babu, Jeremy Hearn, Andrew Emili, X. Sunney Xie
Science, 329, 533-538(2010)
要 約
単一細胞のレベルでは内在するmRNA数とタンパク質数とがたえず乱雑に変動している.このため,ひとつひとつの細胞は,たとえ同じゲノムをもっていても,それぞれが個性的な振る舞いを示す.筆者らは,単一細胞内におけるmRNAとタンパク質の発現プロファイリングを単一分子検出レベルの感度で行うことにより,単一細胞のもつ特性の乱雑さをシステムワイドで定量化し,そこにあるゲノム共通の法則性を明らかにした.そのために,蛍光タンパク質遺伝子をそれぞれの遺伝子のC末端に結合させた大腸菌ライブラリーを1000株以上にわたって作製し,マイクロチップ上で単一分子感度での計測をシステマティックに行うことにより,それぞれの遺伝子におけるmRNAとタンパク質の絶対個数,ばらつき,細胞内局在などの情報を網羅的に取得した.その結果,全体の98%の遺伝子は発現するタンパク質数の分布において特定の共通構造をもっており,それらの分布構造の大きさは量子ノイズやグローバル因子による極限をもつことが判明した.
はじめに
生物は内在するゲノムから数千から数万にわたる種類のタンパク質を生み出すことによって生命活動を行っている.近年,これらの膨大な生物情報を網羅的に取得し,生物を包括的に理解しようとする研究が急速に進展している.2003年にヒトゲノムが完全解読され,現在ではゲノム解読の高速化・低価格化が注目を集める一方で,より直接的に機能レベルの情報を取得する手法として,ゲノム(DNA)の発現産物であるmRNAやタンパク質の発現量を網羅的に調べるトランスクリプトミクスやプロテオミクスに関する研究開発に関心が集まっている.cDNAマイクロアレイ法やRNA-seq法,質量分析法などの技術開発によって発現産物の量をより高感度に探ることが可能となってきているが,いまだ単一分子検出レベルの高感度の実現にはいたっていない.
その一方で,近年のレーザー蛍光顕微鏡技術の発展により,単一細胞内で起こる遺伝子発現を単一分子レベルで検出することが可能になってきた1,2).筆者らは今回,こうした単一分子計測技術を応用することにより,モデル生物である大腸菌(Escherichia coli)について,単一分子・単一細胞レベルでのmRNAとタンパク質の発現プロファイリングをはじめて実現した.
単一分子・単一細胞プロファイリングにおいては,ひとつひとつの細胞に存在するmRNAとタンパク質の絶対個数がそれぞれ決定される.細胞では1つあるいは2つの遺伝子座から確率論的にmRNA,そして,タンパク質の発現が行われているので,ひとつひとつの細胞は同じゲノムをもっていても,内在するmRNAとタンパク質の個数のうちわけには大きな多様性があり,さらにこれは,時々刻々と変化している.つまり,細胞は確率的な遺伝子発現を利用して,表現型の異なる細胞をたえず自発的に生み出しているといえる.こうした乱雑さは生物の大きな特徴であり,これを利用することで細胞の分化や異質化を誘導したり,環境変化に対する生物種の適応度を高めたりしていると考えられている3,4).この研究では,大腸菌について個体レベルでの乱雑さをプロテオームレベルおよびトランスクリプトームレベルで定量化し,そのゲノムに共通する原理を探ることをめざした.
1.大腸菌タンパク質-蛍光タンパク質融合ライブラリーの構築
1分子・1細胞レベルで大腸菌がタンパク質を発現するようすを調べるため,大腸菌染色体内のそれぞれの遺伝子に黄色蛍光タンパク質Venusの遺伝子を導入した大腸菌株ライブラリーを構築した(図1a).このライブラリーは,大腸菌のそれぞれの遺伝子に対応した計1018種類の大腸菌株により構成されており,おのおのの株においては対応する遺伝子のC末端に蛍光タンパク質の遺伝子が挿入されている.遺伝子発現と連動して生じる蛍光タンパク質の蛍光をレーザー顕微鏡により単一分子感度でとらえることによって,遺伝子発現の単一分子観測が可能となる1).
ライブラリーの作製にあたっては,共同研究者であるカナダToronto大学のEmili教授のグループが2006年に作製した,SPA(sequential peptide affinity)ライブラリーを利用した5).このライブラリーでは大腸菌のそれぞれの遺伝子のC末端にタンパク質精製用のSPAタグが挿入されていたが,このタグをλ-Red相同組換え法を用いてVenusの遺伝子に置き換える方法をとることによって,ユニバーサルなプライマーを用いて廉価かつ効率的にライブラリーの作製を行うことができた.
2.ハイスループット解析用のマイクロ流路系の開発
膨大な数のライブラリー株をレーザー顕微鏡によりハイスループットで解析するため,ソフトリソグラフィー技術を用いてシリコン成型したマイクロ流体チップを開発した6)(図1b).このチップは平行に並んだ96のサンプル流路により構成されており,マルチチャネルピペッターを用いてそれぞれに異なるライブラリー株を注入することによって,96のライブラリー株を並列的に2次元配列することができる.チップの底面は薄型カバーガラスになっているためレーザー顕微鏡による高開口数での観察が可能であり,3次元電動ステージを用いてスキャンすることにより多サンプル連続解析が可能となった.チップの3次元スキャン,自動フォーカス,光路の切替え,画像撮影,画像分析など,解析の一連の流れをコンピューターで完全自動化することにより,それぞれのライブラリー株あたり,25秒間に平均4000個の細胞の解析を行うことができた.
3.タンパク質発現数の全ゲノム分布
解析により得られるライブラリー株の位相差像と蛍光像の代表例を表す(図1c).それぞれの細胞におけるタンパク質発現量が蛍光量として検出できると同時に,タンパク質の細胞内局在(膜局在,細胞質局在,DNA局在など)を観察することができた.それぞれの細胞に内在している蛍光に対して単一蛍光分子による規格化を行い,さらに,細胞の自家蛍光による影響を差し引くことによって,それぞれの細胞におけるタンパク質発現数の分布を決定した(図1d).同時に,画像解析によって蛍光分子の細胞内局在(細胞質局在と細胞膜局在との比,点状の局在)をスコア化した(図1e).
この結果,大腸菌のそれぞれの遺伝子の1細胞あたりの平均発現量は,10-1個/細胞から104個/細胞まで,5オーダーにわたって幅広く分布していることがわかった.必須遺伝子の大半が10個/細胞以上の高い発現レベルを示したのに対し,全体ではおおよそ半数の遺伝子が10個/細胞以下の発現レベルを示した.低発現を示すタンパク質のなかには実際に機能していることが示されているものも多く存在しており,これらのタンパク質は10個以下の低分子数でも細胞内で十分に機能することがわかった.このことは,単一細胞レベルの微生物学において,単一分子感度の実験が本質的でありうることを示唆する.
4.タンパク質数分布の普遍的な構造
それぞれの細胞におけるタンパク質数の分布を調べたところ,一般に,低発現数を示すタンパク質の分布は単調減少関数,高発現数を示すタンパク質の分布はピークをもった関数になっていた.さまざまなモデルを用いてフィッティングを行い,すべての遺伝子の分布を一般的に記述できる最良の関数を探した結果,1018遺伝子のうち1009遺伝子をガンマ分布によって記述できることをみつけた.大腸菌はガンマ分布というゲノムに共通の構造にそってプロテオームの多様性を生み出しており,その分布はガンマ分布のもつ2つのパラメーターによって一般的に記述できることが明らかになった.
このガンマ分布は,mRNAの転写とタンパク質の翻訳,mRNAの分解とタンパク質の分解が,それぞれ確率的に起こると仮定した場合のタンパク質数の分布に等しい7)(図2).これはつまり,タンパク質数の分布がセントラルドグマの過程の確率的な特性により決定づけられることを示唆している.そこで以降,このガンマ分布を軸として,細胞のタンパク質量を正しく記述するためのモデルをさらに検証した.
5.タンパク質数のノイズの極限
タンパク質数の分布のばらつきの大きさ,または,ノイズ(発現数の標準偏差の2乗と発現数の平均の2乗の比と定義される)は,個々の細胞におけるタンパク質量の多様性を表す重要なパラメーターである3).このノイズをそれぞれの遺伝子について求めたところ,つぎに示すような発現量の大きさに応じた二相性のあることをみつけた.
平均発現数が10分子以下の遺伝子は,ほぼすべてがポアソンノイズを下限とする,発現数と反比例した量のノイズをもっていた.このポアソンノイズは一種の量子ノイズであり,遺伝子発現が純粋にランダムに(すなわち,ポアソン過程で)行われた場合のノイズ量を表している.つまり今回の結果は,タンパク質発現のノイズをポアソンノイズ以下に抑えるような遺伝子制御機構は存在しないことを示唆する.実際のノイズがポアソンノイズを上まわるということは,遺伝子の発現が準ランダムに行われていることを表している.実際,ひとつひとつのタンパク質の発現は純粋なランダムではなく,mRNAの発現とともに突発的に複数のタンパク質の発現(バースト)が起こり,mRNAの分解と同時にタンパク質の発現がとまる,といったかたちでバースト的に行われることが報告されている1).筆者らは,複数のライブラリー株をリアルタイム計測することでバーストの観測を行うことにより,バーストの頻度と大きさが細胞集団計測で得られるノイズの大きさに合致することをみつけた.これはつまり,ノイズの大きさがmRNAバーストの性質により決定されていることを表している.
一方で,平均発現数が10分子以上の遺伝子は,ポアソンノイズとは異なる,発現数に依存しない一様なノイズ極限をもっていた.すべての遺伝子はこのノイズ極限よりも大きなノイズをもっていることから,大腸菌に発現するタンパク質は必ず一定割合(30%)以上のノイズをもっていることが示された.
6.タンパク質発現量の遅い時間ゆらぎ
この一様なノイズ極限の起源を調べるため,高発現を示す複数のライブラリー株を無作為に抽出し,これらのタンパク質量の時間的な変化をタイムラプス観測により調べた.高発現タンパク質が一定の確率でランダムに発現している場合,ひとつひとつの細胞に存在するタンパク質の数は短い時間スケールで乱雑に変動し,数分もすればもとあったタンパク質レベルが初期化され,それぞれがまったく別のタンパク質レベルとなるはずである8).これに反して,今回のライブラリー株ではひとつひとつの細胞でのタンパク質レベルの大小が十数世代(1000分間以上)にわたって維持されていることが観測された.これはつまり,細胞ひとつひとつが互いに異なる細胞状態をもっており,さらに,この状態が何世代にもわたって“記憶”されていることを示している.
ノイズ解析で観測された一様なノイズ極限は,こうした細胞状態の不均一性により説明できることがみつけられた.セントラルドグマの過程(図2)において,それぞれの細胞が異なる速度定数をもつとする.この場合,ノイズの値には,発現量に反比例した固有成分にくわえて,発現量に依存しない定数成分が現われるようになる.この定数成分が高発現タンパク質において優勢になることから,一様なノイズ極限が観測されたといえる.つまり,一様なノイズ極限は,細胞内で起こるタンパク質発現のランダム性からではなく,それぞれの細胞の特性のばらつき(たとえば,ポリメラーゼやリボソームの数の不均一性など)から生じたとすることにより説明できた.
7.単一細胞における遺伝子発現量のグローバルな相関
さらに,この一様なノイズ極限がポリメラーゼやリボソームなどすべての遺伝子の発現にかかわるグローバルな因子により生み出されていることを突き止めた.これを示すために,複数の2遺伝子の組合せを無作為に抽出し,異なる蛍光タンパク質でラベル化することによって1つの細胞における2つの遺伝子の発現レベルにおける相関関係を調べた.その結果,どの2遺伝子の組合せに関しても正の相関が観察され,細胞状態に応じてすべての遺伝子の発現の大小がひとまとめに制御されていることがわかった.相関解析からこうした“グローバルノイズ”の量は30%と求まり,一様なノイズ極限の値と一致した.
8.mRNAプロファイリング
つぎに,タンパク質発現の中間産物であるmRNAの量を単一分子感度・単一細胞分解能でプロファイリングすることを試みた.そのために,蛍光in situハイブリダイゼーション(FISH)法を用いて,ライブラリーの黄色蛍光タンパク質のmRNAに赤色蛍光ヌクレオチドを選択的にハイブリダイゼーションした.この方法ではすべてのライブラリーに対して同じプローブを用いるため,遺伝子ごとのバイアスがほとんどない.レーザー顕微鏡を用いて細胞内の蛍光ヌクレオチドを数えることにより,mRNA数の決定を行った.
mRNA数のノイズを調べた結果,タンパク質の場合とは異なり,ポアソンノイズにもとづくノイズ極限だけがみられた.これは,mRNAの数は少ないためにポアソンノイズが大きくなり,一様なノイズ極限の影響が現われなくなったためであると考えられた.
9.mRNAレベルとタンパク質レベルとの非相関性
赤色蛍光ヌクレオチドと黄色蛍光タンパク質の蛍光スペクトルが異なることを利用して,単一細胞におけるmRNA数とタンパク質数を同時に測定しその相関を調べた.137の遺伝子に対して測定を行ったところ,どの遺伝子においてもこれらのあいだには強い相関はなかった.つまり,単一細胞においては内在するmRNA数とタンパク質数とのあいだには相関のないことが判明した.
この非相関性のおもな理由としてmRNAの分解時間の速さがあげられる.RNA-seq法を用いてmRNAの分解時定数を調べたところ,数分以下であった.これに対し,ほとんどのタンパク質の分解時定数は数時間以上であり,タンパク質数の減衰はおもに細胞分裂による希釈効果により起こることが知られている9).したがって,mRNAの数は数分以内に起こった現象を反映するのに対し,タンパク質の数は細胞分裂の時間スケール(150分)のあいだで積み重なった現象を反映することになり,これらの数のあいだに不一致が起こるものと考えられる.
単一細胞におけるmRNA量の高ノイズ性を示す今回の結果は,1細胞レベルでのトランスクリプトーム解析に対してひとつの警告をあたえるものであり,同時に,プロテオーム解析の必要性を表している.
10.1分子・1細胞レベルでの発現特性と生物学的機能との相関
得られた1分子・1細胞レベルでの発現特性が生物学的な機能とどのように相関しているかを統計的に調べた.たとえば,タンパク質発現平均数はコドン使用頻度の指標であるCAI(codon adaptation index)と正の相関をもつのに対し,GC含量やmRNAの分解時間,染色体上の位置との相関はなかった.また,膜トランスポーターの遺伝子は高い膜局在性,転写因子は高い点局在性を示した.また,短い遺伝子は高いタンパク質発現を示すことや,リーディング鎖にある遺伝子からの転写はラギング鎖にある遺伝子からの転写よりも多いことがわかった.さらに,大腸菌のノイズは出芽酵母のノイズと比べ高いことも明らかになった10).
ここで示したのはほんの一例であり,相関解析の全データ,それぞれの遺伝子情報の全データは原著論文のSupporting Online Materialに掲載しているので,参考にしてほしい.
おわりに
この研究で構築した単一分子・単一細胞プロファイリング技術は,複雑な細胞システムを素子である1分子レベルから理解することを可能とするものであり,1分子・1細胞生物学とシステム生物学とをつなぐ架け橋となりうる.以下,従来のプロファイリングの手法と比べた場合のアドバンテージをまとめる.
1)単一細胞内における遺伝子発現の絶対個数がわかる.
2)細胞を生きたまま解析でき,リアルタイムでの解析が可能.
3)細胞ごとの遺伝子発現量の確率論的なばらつきを解析できる.
4)ごくわずかな割合で存在する異常細胞を発見できる.
5)シグナル増幅が不要であり,遺伝子によるバイアスがきわめて少ない.
6)単一細胞内での2遺伝子の相互作用解析が可能.
7)細胞内におけるタンパク質局在を決定できる.
これらのアドバンテージを利用することで,細胞ひとつひとつの分子数や細胞状態の違いを絶対感度でとらえることが可能となり,さまざまな生命現象をより精密に調べることが可能となる.この研究では,生物特有の性質である個体レベルでの生命活動の“乱雑さ”を直接とらえることを目的としてこの技術を利用し,その一般原理のひとつを明らかにしている.
この研究で得られた大腸菌の単一分子・単一細胞プロファイルは,分子・細胞相互の階層から生物をシステムとして理解するための包括的データリソースとして役立つとともに,生物のもつ乱雑性,多様性を理解するためのひとつの基礎になるものと期待される.
文 献
- Yu, J., Xiao, J., Ren, X. et al.: Probing gene expression in live cells, one protein molecule at a time. Science, 311, 1600-1603 (2006)[PubMed]
- Golding, I., Paulsson, J., Zawilski, S. M. et al.: Real-time kinetics of gene activity in individual bacteria. Cell, 123, 1025-1036 (2005)[PubMed]
- Elowitz, M. B., Levine, A. J., Siggia, E. D. et al.: Stochastic gene expression in a single cell. Science, 297, 1183-1186 (2002)[PubMed]
- Wernet, M. F., Mazzoni, E. O., Celic, A. et al.: Stochastic spineless expression creates the retinal mosaic for colour vision. Nature, 440, 174-180 (2006)[PubMed]
- Butland, G., Peregrin-Alvarez, J. M., Li, J. et al.: Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature, 433, 531-537 (2005)[PubMed]
- McDonald J. C. & Whitesides. G. M.: Poly(dimethylsiloxane) as a material for fabricating microfluidic devices. Acc. Chem. Res., 35, 491-499 (2002)[PubMed]
- Friedman, N., Cai, L., Xie. X. S.: Linking stochastic dynamics to population distribution: an analytical framework of gene expression. Phys. Rev. Lett., 97, 168302 (2006)[PubMed]
- Rosenfeld, N., Young, J. W., Alon, U.: et al.: Gene regulation at the single-cell level. Science, 307, 1962-1965 (2005)[PubMed]
- Kock, A. L. & Levy. H. R.: Protein turnover in growing cultures of Escherichia coli. J. Biol. Chem., 217, 947-957 (1955)[PubMed]
- Newman, J. R. S., Ghaemmaghami, S., Ihmels, J. et al.: Single-cell proteomic analysis of S. cerevisiae reveals the architecture of biological noise. Nature, 441, 840-846 (2006)[PubMed]
著者プロフィール
略歴:2006年 大阪大学大学院基礎工学研究科博士課程 修了,同年より米国Harvard大学 ポストドクトラルフェロー.
専門分野:生物物理学,ナノバイオロジー.
キーワード:1分子・1細胞生物学,システム生物学,プロテオミクス,超高感度顕微鏡技術,微細加工技術,生命反応の物理,生物ゆらぎ.
抱負:顕微鏡工学,マイクロ工学,遺伝子工学,コンピューター工学など,さまざまな分野にまたがるさまざまな要素技術を組み合わせて,生命を理解するための新しい画期的な技術をつくるのが仕事です.生物学,物理学,統計学などのあらゆる立場から生命活動の本質を理解し,人々の疾病克服,健康増進に役立てることが目標です.
© 2010 谷口 雄一 Licensed under CC 表示 2.1 日本