非対称な結晶構造から明らかになったV1-ATPaseの回転機構
西條慎也1・山登一郎1・村田武士2
(1東京理科大学基礎工学部 生物工学科・2千葉大学大学院理学研究科 生体構造化学研究室)
email:村田武士
DOI: 10.7875/first.author.2013.020
Rotation mechanism of Enterococcus hirae V1-ATPase based on asymmetric crystal structures.
Satoshi Arai, Shinya Saijo, Kano Suzuki, Kenji Mizutani, Yoshimi Kakinuma, Yoshiko Ishizuka-Katsura, Noboru Ohsawa, Takaho Terada, Mikako Shirouzu, Shigeyuki Yokoyama, So Iwata, Ichiro Yamato, Takeshi Murata
Nature, 493, 703-707 (2013)
V-ATPaseは細菌からヒトまで多くの生物の生体膜に存在し,ATPにより駆動されるイオンポンプとして機能している.V-ATPaseは親水性のV1-ATPaseと膜内在性のVo-ATPaseから構成される.V1-ATPaseはATPの加水分解のエネルギーを利用し,ヘテロ六量体からなるA3B3複合体の内部で中心軸であるDF複合体が回転する分子モーターであるが,高分解能の立体構造が得られていなかったため,その詳細な分子機構は不明であった.筆者らは,ヌクレオチド結合型および非結合型のA3B3複合体のX線結晶構造を解明し,ヌクレオチドとの結合により起こる構造変化を明らかにした.さらに,ヌクレオチド結合型および非結合型のV1-ATPase(A3B3DF複合体)のX線結晶構造を解明し,中心軸であるDF複合体の結合により起こるA3B3複合体の構造変化を明らかにし,ATPが分解される部位を推定した.これらの非対称な結晶構造にもとづき,V1-ATPaseの回転機構のモデルを提案した.
ATPは細胞におけるエネルギー通貨であり,おもにATP合成酵素であるF-ATPaseにより合成される1).そして,多数の代謝プロセスがATP加水分解のエネルギーにより駆動されている.V-ATPase(Vacuolar ATPase,液胞型ATPase)はこのエネルギーを利用し,真核生物において酸性オルガネラ膜にてプロトンを輸送し内部の酸性化を担っている.V-ATPaseは細胞膜にも存在し,細胞間マトリックスを酸性化し骨吸収やがんの転移にも関与している2).そのため,V-ATPaseは骨粗鬆症やがんなど,多くの疾病にかかわる創薬ターゲットとしても注目されている.
V-ATPaseはF-ATPaseと共通の祖先から進化してきたと考えられている.ATP分解(合成)を行う親水性のV1部分あるいはF1部分と,イオンポンプである膜内在性のVo部分あるいはFo部分とが,中心軸と周辺固定子により連結された共通の構造をもつ.触媒機能をもつV1-ATPaseおよびF1-ATPaseは回転分子モーターであることが知られている.F1-ATPaseの回転機構については,X線結晶構造解析3) や1分子観察4) により数多くの研究がなされてきた.一方,V1-ATPaseに関しては,ATP合成酵素として機能するThermus thermophilusに由来するものを用いて研究は先導されてきた.A3B3複合体の結晶構造5)(分解能2.8Å)や,V1-ATPase(A3B3DF複合体)の結晶構造6)(分解能4.5~4.8Å),1分子観察7) から,F1-ATPaseとの違いが明らかになってきたが,V1-ATPaseの詳細な回転機構の理解には高分解能での結晶構造の解明が不可欠であった.
筆者らは,腸球菌(Enterococcus hirae)から発見されたV-ATPaseの類縁酵素について,分子生物学的,生化学的,構造生物学的な研究を展開し,この酵素が真核生物のもつV-ATPaseのATPの加水分解に特化したホモログであることを明らかにしてきた8).この研究では,腸球菌のV-ATPaseの回転機構の解明を目的として,V1-ATPaseを構成するサブユニット(A,B,D,F)について,発現系,精製系,再構成系を構築し,得られた複合体のX線結晶構造解析を行った.
筆者らは,1995年以来,V1-ATPaseのX線結晶構造解析をめざし,腸球菌からV1-ATPase(A3B3DF複合体)を精製し結晶化を進めてきた.結晶はすぐに得られたものの,その分解能は6Å程度にとどまりX線結晶構造解析に進むことはできなかった.そののち,大腸菌発現系を用いてAサブユニットおよびBサブユニットをそれぞれ発現させ,精製ののち,A3B3複合体の再構成条件をみつけたが9),大量調製が困難なため良質の結晶を得ることはできなかった.今回,最終的に大腸菌無細胞タンパク質合成系を用いることでA3B3複合体の大量調製に成功し,この高純度のタンパク質試料を用いることにより結晶構造を得ることができた.さらに,A3B3複合体とDF複合体からのV1-ATPase(A3B3DF複合体)の再構成条件を表面プラズモン共鳴法を用いて検討した.その結果,酸性pHやMg2+の存在により安定に精製することができるようになり,この標品を用いることでV1-ATPaseの高分解能X線結晶構造を得ることができた.
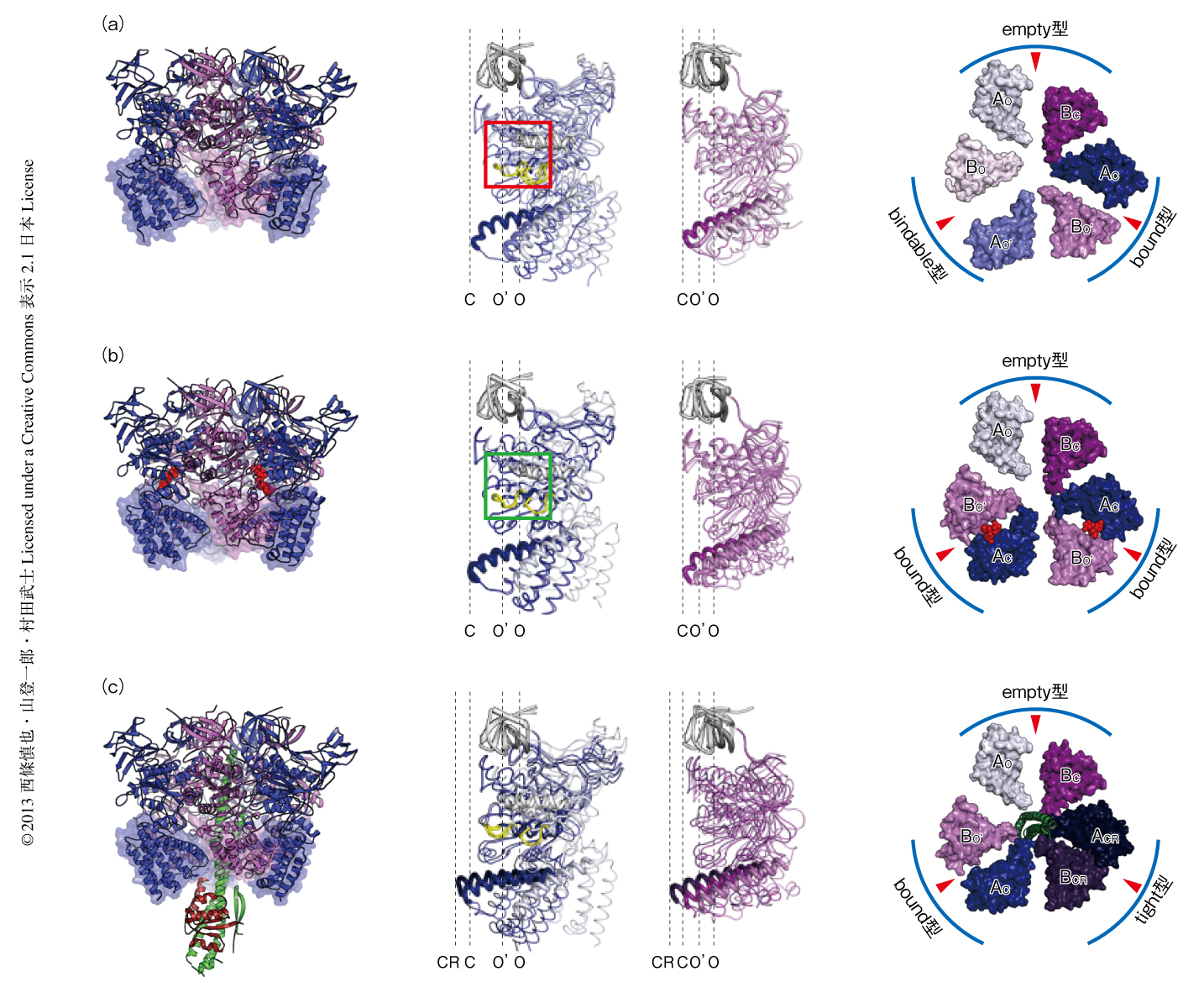
ATPやADPなどのヌクレオチドの非存在下で得られたヌクレオチド非結合型A3B3複合体の結晶構造を分解能2.8Åで決定した(PDB ID:3VR2,図1a).得られた構造は,触媒サブユニットであるAサブユニットと非触媒サブユニットであるBサブユニットがそれぞれ3つ,互い違いに配置したヘテロ六量体リングから構成されていた.どちらのサブユニットもN末端側にあるβバレル,中間にあるα/βドメイン,C末端側にあるヘリカルドメインから構成されているが,ヘテロ六量体リングのなかで固定されているN末端側のβバレルについてそれぞれを重ね合わせたところ,Aサブユニットのうちの1つはA3B3複合体のリングの中心にむかいシフトしているclosed構造(AC)をとり,残り2つのAサブユニットは互いによく似たopen構造(AOおよびAO’)をとっていた.同様に,Bサブユニットも1つはclosed構造(BC)を,2つはopen構造(BOおよびBO’)をとっていた.3箇所あるヌクレオチド結合部位はAOBCペア,AO’BOペア,ACBO’ペアのあいだに位置し,AサブユニットのPループ,Aサブユニットのアーム領域のN末端側に存在するGlu261およびArg262,BサブユニットのArgフィンガーとよばれるArg350から構成されていたが,驚くべきことに,ヌクレオチドと結合していないにもかかわらず,これら3つのヌクレオチド結合部位は異なるコンホメーションを形成していた.
つぎに,ATPアナログであるAMP-PNPの存在下で結晶化させたヌクレオチド結合型A3B3複合体の構造を分解能3.4Åで決定した(PDB ID:3VR3,図1b).3箇所のヌクレオチド結合部位のうち2箇所にAMP-PNPの電子密度が確認された.AMP-PNPと結合していなかったABペアはヌクレオチド非結合型A3B3複合体のAOBCペアにもっとも類似したコンホメーションをとっており,ヌクレオチドに対する親和性が低いと考えられたため,このペアをempty型と名づけた.また,AMP-PNPと結合していた2つのABペアの構造は互いによく似ており,また,ヌクレオチド非結合型A3B3複合体のACBO’ペアとも,AMP-PNPと相互作用していた側鎖の構造のほかはよく似ていた.このACBO’ペアはヌクレオチドを結合している状態の構造であると考えられたのでbound型と名づけた.ヌクレオチド非結合型A3B3複合体のAO’BOペアは,AMP-PNPの存在下ではこれと結合しbound型に構造変化すると考えられた.そこで,このAO’BOペアはヌクレオチドと結合できるという意味でbindable型と名づけた.それでは,bindable型はどのようにしてAMP-PNPを認識しbound型へと構造を変化させるのだろうか? AMP-PNPのγリン酸と相互作用するArg262とArg350との距離は,bindable型とbound型とで類似していた.一方,empty型ではArg262とArg350との距離が近づいており,この違いがヌクレオチドに対する親和性の違いを生んでいる可能性が示唆された.
このように,A3B3複合体はATPと結合できない型(empty型),ATPと結合することができる型(bindable型),ATPと結合している型(bound型)の3つの異なるABペアから構成されていることが明らかになった.ATPの存在下ではbindable型とbound型にATPが結合し2つのbound型ができるが,もともとあったbound型においてATPが分解されると,A3B3複合体ははじめの構造にもどるように変化すると考えられた.つまり,A3B3複合体の構造を120度回転させた構造に変化することになる.以上の考察から,V1-ATPaseがATPのエネルギーを使って一方向に回転するしくみを,A3B3複合体の非対称構造から理解することができた.
ヌクレオチド非結合型のV1-ATPase(A3B3DF複合体)の結晶構造を分解能2.2Åで決定した(PDB ID:3VR4,図1c).A3B3複合体と同様に,非対称な六量体を形成するAサブユニットとBサブユニットの中心の空洞部分に,中心軸であるDサブユニットとFサブユニットが挿入された構造をとっていた.DサブユニットはDF複合体のみの結晶構造10) よりもまっすぐな構造で,コイルドコイル構造のαヘリックスはA3B3複合体の内部にある多くの残基と相互作用していた.V1-ATPaseのもつATPase活性を促進させるのに重要なDサブユニットの短いβヘアピンとFサブユニットのC末端領域は,BサブユニットのC末端ドメインと相互作用していた.
ヌクレオチド非結合型のA3B3複合体とV1-ATPase(A3B3DF複合体)の立体構造を比較することにより,DF複合体との相互作用によりひき起こされる構造変化を理解することができた.ヌクレオチド非結合型A3B3DF複合体には,1つのempty型(AOBCペア)と1つのbound型(ACBO’ペア)が存在していた.しかしながら,empty型どうしの位置を基準とした場合,ヌクレオチド非結合型A3B3複合体のbindable型はDF複合体との相互作用によりA3B3DF複合体ではbound型に変化していた.この構造変化はヌクレオチド結合型A3B3複合体においてAMP-PNPとの結合により起こった変化と非常によく似ていた.残りのABペアはA3B3複合体には存在しない,より閉じたコンホメーションを形成していた.このコンホメーションをcloser構造(ACRおよびBCR),ACRBCRペアをtight型と名づけた.ヌクレオチド非結合型A3B3複合体のbound型がDF複合体との相互作用によりtight型へと変化したものと考えられた.
tight型のヌクレオチド結合部位ではBCRのArgフィンガー(Arg350)がACRのアーム領域のArg262に接近していた.このArg350の構造変化がヌクレオチドとの結合にどのような影響をあたえているかを知るため,ATPアナログであるAMP-PNPと結合したヌクレオチド結合型のV1-ATPase(A3B3DF複合体)の構造を分解能2.7Åで決定した(PDB ID:3VR6).AMP-PNPの電子密度はbound型とtight型の2箇所に存在し,empty型には存在しなかった.ヌクレオチド結合型A3B3複合体での結果と同様に,empty型ではヌクレオチドに対する親和性が低いものと考えられた.AMP-PNPのγリン酸とMg2+はヌクレオチド結合型A3B3複合体と同様に,AサブユニットのLys238,Thr239,Arg262とBサブユニットのArgフィンガー(Arg350)と相互作用していた.ヌクレオチド結合型V1-ATPaseのtight型およびbound型のヌクレオチド結合部位を比較したところ,tight型においてArgフィンガーがγリン酸に対し1.6Å近づき,γリン酸それ自体もGlu261に対し0.7Å移動していた.Glu261は出芽酵母のV1-ATPaseにおいてはATPase活性に必須で11),F1-ATPaseにおいてもγリン酸の酸素原子と水を介し相互作用している12).以上の結果から,DF複合体との相互作用により起こるArgフィンガーの動きがATP加水分解のきっかけとなることが示唆され,今回,得られたヌクレオチド結合型V1-ATPaseの結晶構造はATP加水分解待ちの中間状態をとらえているものと考えられた.
今回,得られた結晶構造をもとに,V1-ATPaseの回転機構のモデルを提案する(図2).ヌクレオチド結合型V1-ATPaseではbound型とtight型にATPが結合している.tight型に結合したATPはArgフィンガーがγリン酸に近づいた分解待ちの状態であり,このATPが加水分解することにより反応はスタートする.ATPが加水分解されるとA3B3複合体には定常状態であるヌクレオチド非結合型の構造にもどるような構造変化が促進されると考えられる.つまり,tight型はempty型へと構造変化し,ヌクレオチドに対する親和性が低下した結果,ADPとリン酸の遊離が起こる.一方,empty型はATPとの結合の可能なbindable型へと構造変化を起こし,A3B3複合体はヌクレオチド非結合型の状態になる方向へと進む.しかしながら,tight型とDF複合体は強く結合しているため,A3B3複合体の構造変化は抑制されると考えられ,実際には,これらの中間状態に変化すると予想している.つぎに,bindable型あるいは中間状態のABペアとATPとの結合によりA3B3複合体はヌクレオチド結合型の構造に変化し,2つのbound型にATPが結合し,これによりDF複合体は回転する.最後に,もともとあったbound型がDF複合体との相互作用によりtight型へと構造変化し,Argフィンガーがγリン酸に近づいて,はじめから120度回転したヌクレオチド結合型V1-ATPaseの構造にもどる.
今回,腸球菌に由来するV-ATPaseについて,ヌクレオチド非結合型およびヌクレオチド結合型のA3B3複合体およびV1-ATPase(A3B3DF複合体)の結晶構造を明らかにした.
ヌクレオチド非結合型A3B3複合体の構造それ自体が非対称性をもち,その非対称性が回転の方向性を決定していることや,DF複合体との結合によりtight型への構造変化が起こりATP加水分解のトリガーとなることなどが示唆され,V1-ATPaseの新しい回転機構のモデルを提案することができた.これにより,ほかの分子モーターを含む生体エネルギー変換機構の原理の解明につながることが期待される.さらに現在,より詳細な回転機構の解明にむけ,反応中間体の結晶構造解析や分子動力学シミュレーション,1分子観察などの相関構造解析を進めている.
東京理科大学基礎工学部 助教.
山登 一郎(Ichiro Yamato)
東京理科大学基礎工学部 教授.
村田 武士(Takeshi Murata)
千葉大学大学院理学研究科 特任准教授.
研究室URL:http://murata-lab.matrix.jp/
© 2013 西條慎也・山登一郎・村田武士 Licensed under CC 表示 2.1 日本
(1東京理科大学基礎工学部 生物工学科・2千葉大学大学院理学研究科 生体構造化学研究室)
email:村田武士
DOI: 10.7875/first.author.2013.020
Rotation mechanism of Enterococcus hirae V1-ATPase based on asymmetric crystal structures.
Satoshi Arai, Shinya Saijo, Kano Suzuki, Kenji Mizutani, Yoshimi Kakinuma, Yoshiko Ishizuka-Katsura, Noboru Ohsawa, Takaho Terada, Mikako Shirouzu, Shigeyuki Yokoyama, So Iwata, Ichiro Yamato, Takeshi Murata
Nature, 493, 703-707 (2013)
要 約
V-ATPaseは細菌からヒトまで多くの生物の生体膜に存在し,ATPにより駆動されるイオンポンプとして機能している.V-ATPaseは親水性のV1-ATPaseと膜内在性のVo-ATPaseから構成される.V1-ATPaseはATPの加水分解のエネルギーを利用し,ヘテロ六量体からなるA3B3複合体の内部で中心軸であるDF複合体が回転する分子モーターであるが,高分解能の立体構造が得られていなかったため,その詳細な分子機構は不明であった.筆者らは,ヌクレオチド結合型および非結合型のA3B3複合体のX線結晶構造を解明し,ヌクレオチドとの結合により起こる構造変化を明らかにした.さらに,ヌクレオチド結合型および非結合型のV1-ATPase(A3B3DF複合体)のX線結晶構造を解明し,中心軸であるDF複合体の結合により起こるA3B3複合体の構造変化を明らかにし,ATPが分解される部位を推定した.これらの非対称な結晶構造にもとづき,V1-ATPaseの回転機構のモデルを提案した.
はじめに
ATPは細胞におけるエネルギー通貨であり,おもにATP合成酵素であるF-ATPaseにより合成される1).そして,多数の代謝プロセスがATP加水分解のエネルギーにより駆動されている.V-ATPase(Vacuolar ATPase,液胞型ATPase)はこのエネルギーを利用し,真核生物において酸性オルガネラ膜にてプロトンを輸送し内部の酸性化を担っている.V-ATPaseは細胞膜にも存在し,細胞間マトリックスを酸性化し骨吸収やがんの転移にも関与している2).そのため,V-ATPaseは骨粗鬆症やがんなど,多くの疾病にかかわる創薬ターゲットとしても注目されている.
V-ATPaseはF-ATPaseと共通の祖先から進化してきたと考えられている.ATP分解(合成)を行う親水性のV1部分あるいはF1部分と,イオンポンプである膜内在性のVo部分あるいはFo部分とが,中心軸と周辺固定子により連結された共通の構造をもつ.触媒機能をもつV1-ATPaseおよびF1-ATPaseは回転分子モーターであることが知られている.F1-ATPaseの回転機構については,X線結晶構造解析3) や1分子観察4) により数多くの研究がなされてきた.一方,V1-ATPaseに関しては,ATP合成酵素として機能するThermus thermophilusに由来するものを用いて研究は先導されてきた.A3B3複合体の結晶構造5)(分解能2.8Å)や,V1-ATPase(A3B3DF複合体)の結晶構造6)(分解能4.5~4.8Å),1分子観察7) から,F1-ATPaseとの違いが明らかになってきたが,V1-ATPaseの詳細な回転機構の理解には高分解能での結晶構造の解明が不可欠であった.
筆者らは,腸球菌(Enterococcus hirae)から発見されたV-ATPaseの類縁酵素について,分子生物学的,生化学的,構造生物学的な研究を展開し,この酵素が真核生物のもつV-ATPaseのATPの加水分解に特化したホモログであることを明らかにしてきた8).この研究では,腸球菌のV-ATPaseの回転機構の解明を目的として,V1-ATPaseを構成するサブユニット(A,B,D,F)について,発現系,精製系,再構成系を構築し,得られた複合体のX線結晶構造解析を行った.
1.大腸菌無細胞タンパク質合成系を用いた発現と結晶化
筆者らは,1995年以来,V1-ATPaseのX線結晶構造解析をめざし,腸球菌からV1-ATPase(A3B3DF複合体)を精製し結晶化を進めてきた.結晶はすぐに得られたものの,その分解能は6Å程度にとどまりX線結晶構造解析に進むことはできなかった.そののち,大腸菌発現系を用いてAサブユニットおよびBサブユニットをそれぞれ発現させ,精製ののち,A3B3複合体の再構成条件をみつけたが9),大量調製が困難なため良質の結晶を得ることはできなかった.今回,最終的に大腸菌無細胞タンパク質合成系を用いることでA3B3複合体の大量調製に成功し,この高純度のタンパク質試料を用いることにより結晶構造を得ることができた.さらに,A3B3複合体とDF複合体からのV1-ATPase(A3B3DF複合体)の再構成条件を表面プラズモン共鳴法を用いて検討した.その結果,酸性pHやMg2+の存在により安定に精製することができるようになり,この標品を用いることでV1-ATPaseの高分解能X線結晶構造を得ることができた.
2.A3B3複合体の非対称な結晶構造
ATPやADPなどのヌクレオチドの非存在下で得られたヌクレオチド非結合型A3B3複合体の結晶構造を分解能2.8Åで決定した(PDB ID:3VR2,図1a).得られた構造は,触媒サブユニットであるAサブユニットと非触媒サブユニットであるBサブユニットがそれぞれ3つ,互い違いに配置したヘテロ六量体リングから構成されていた.どちらのサブユニットもN末端側にあるβバレル,中間にあるα/βドメイン,C末端側にあるヘリカルドメインから構成されているが,ヘテロ六量体リングのなかで固定されているN末端側のβバレルについてそれぞれを重ね合わせたところ,Aサブユニットのうちの1つはA3B3複合体のリングの中心にむかいシフトしているclosed構造(AC)をとり,残り2つのAサブユニットは互いによく似たopen構造(AOおよびAO’)をとっていた.同様に,Bサブユニットも1つはclosed構造(BC)を,2つはopen構造(BOおよびBO’)をとっていた.3箇所あるヌクレオチド結合部位はAOBCペア,AO’BOペア,ACBO’ペアのあいだに位置し,AサブユニットのPループ,Aサブユニットのアーム領域のN末端側に存在するGlu261およびArg262,BサブユニットのArgフィンガーとよばれるArg350から構成されていたが,驚くべきことに,ヌクレオチドと結合していないにもかかわらず,これら3つのヌクレオチド結合部位は異なるコンホメーションを形成していた.
つぎに,ATPアナログであるAMP-PNPの存在下で結晶化させたヌクレオチド結合型A3B3複合体の構造を分解能3.4Åで決定した(PDB ID:3VR3,図1b).3箇所のヌクレオチド結合部位のうち2箇所にAMP-PNPの電子密度が確認された.AMP-PNPと結合していなかったABペアはヌクレオチド非結合型A3B3複合体のAOBCペアにもっとも類似したコンホメーションをとっており,ヌクレオチドに対する親和性が低いと考えられたため,このペアをempty型と名づけた.また,AMP-PNPと結合していた2つのABペアの構造は互いによく似ており,また,ヌクレオチド非結合型A3B3複合体のACBO’ペアとも,AMP-PNPと相互作用していた側鎖の構造のほかはよく似ていた.このACBO’ペアはヌクレオチドを結合している状態の構造であると考えられたのでbound型と名づけた.ヌクレオチド非結合型A3B3複合体のAO’BOペアは,AMP-PNPの存在下ではこれと結合しbound型に構造変化すると考えられた.そこで,このAO’BOペアはヌクレオチドと結合できるという意味でbindable型と名づけた.それでは,bindable型はどのようにしてAMP-PNPを認識しbound型へと構造を変化させるのだろうか? AMP-PNPのγリン酸と相互作用するArg262とArg350との距離は,bindable型とbound型とで類似していた.一方,empty型ではArg262とArg350との距離が近づいており,この違いがヌクレオチドに対する親和性の違いを生んでいる可能性が示唆された.
このように,A3B3複合体はATPと結合できない型(empty型),ATPと結合することができる型(bindable型),ATPと結合している型(bound型)の3つの異なるABペアから構成されていることが明らかになった.ATPの存在下ではbindable型とbound型にATPが結合し2つのbound型ができるが,もともとあったbound型においてATPが分解されると,A3B3複合体ははじめの構造にもどるように変化すると考えられた.つまり,A3B3複合体の構造を120度回転させた構造に変化することになる.以上の考察から,V1-ATPaseがATPのエネルギーを使って一方向に回転するしくみを,A3B3複合体の非対称構造から理解することができた.
3.V1-ATPaseの結晶構造
ヌクレオチド非結合型のV1-ATPase(A3B3DF複合体)の結晶構造を分解能2.2Åで決定した(PDB ID:3VR4,図1c).A3B3複合体と同様に,非対称な六量体を形成するAサブユニットとBサブユニットの中心の空洞部分に,中心軸であるDサブユニットとFサブユニットが挿入された構造をとっていた.DサブユニットはDF複合体のみの結晶構造10) よりもまっすぐな構造で,コイルドコイル構造のαヘリックスはA3B3複合体の内部にある多くの残基と相互作用していた.V1-ATPaseのもつATPase活性を促進させるのに重要なDサブユニットの短いβヘアピンとFサブユニットのC末端領域は,BサブユニットのC末端ドメインと相互作用していた.
ヌクレオチド非結合型のA3B3複合体とV1-ATPase(A3B3DF複合体)の立体構造を比較することにより,DF複合体との相互作用によりひき起こされる構造変化を理解することができた.ヌクレオチド非結合型A3B3DF複合体には,1つのempty型(AOBCペア)と1つのbound型(ACBO’ペア)が存在していた.しかしながら,empty型どうしの位置を基準とした場合,ヌクレオチド非結合型A3B3複合体のbindable型はDF複合体との相互作用によりA3B3DF複合体ではbound型に変化していた.この構造変化はヌクレオチド結合型A3B3複合体においてAMP-PNPとの結合により起こった変化と非常によく似ていた.残りのABペアはA3B3複合体には存在しない,より閉じたコンホメーションを形成していた.このコンホメーションをcloser構造(ACRおよびBCR),ACRBCRペアをtight型と名づけた.ヌクレオチド非結合型A3B3複合体のbound型がDF複合体との相互作用によりtight型へと変化したものと考えられた.
tight型のヌクレオチド結合部位ではBCRのArgフィンガー(Arg350)がACRのアーム領域のArg262に接近していた.このArg350の構造変化がヌクレオチドとの結合にどのような影響をあたえているかを知るため,ATPアナログであるAMP-PNPと結合したヌクレオチド結合型のV1-ATPase(A3B3DF複合体)の構造を分解能2.7Åで決定した(PDB ID:3VR6).AMP-PNPの電子密度はbound型とtight型の2箇所に存在し,empty型には存在しなかった.ヌクレオチド結合型A3B3複合体での結果と同様に,empty型ではヌクレオチドに対する親和性が低いものと考えられた.AMP-PNPのγリン酸とMg2+はヌクレオチド結合型A3B3複合体と同様に,AサブユニットのLys238,Thr239,Arg262とBサブユニットのArgフィンガー(Arg350)と相互作用していた.ヌクレオチド結合型V1-ATPaseのtight型およびbound型のヌクレオチド結合部位を比較したところ,tight型においてArgフィンガーがγリン酸に対し1.6Å近づき,γリン酸それ自体もGlu261に対し0.7Å移動していた.Glu261は出芽酵母のV1-ATPaseにおいてはATPase活性に必須で11),F1-ATPaseにおいてもγリン酸の酸素原子と水を介し相互作用している12).以上の結果から,DF複合体との相互作用により起こるArgフィンガーの動きがATP加水分解のきっかけとなることが示唆され,今回,得られたヌクレオチド結合型V1-ATPaseの結晶構造はATP加水分解待ちの中間状態をとらえているものと考えられた.
4.V1-ATPaseの回転機構
今回,得られた結晶構造をもとに,V1-ATPaseの回転機構のモデルを提案する(図2).ヌクレオチド結合型V1-ATPaseではbound型とtight型にATPが結合している.tight型に結合したATPはArgフィンガーがγリン酸に近づいた分解待ちの状態であり,このATPが加水分解することにより反応はスタートする.ATPが加水分解されるとA3B3複合体には定常状態であるヌクレオチド非結合型の構造にもどるような構造変化が促進されると考えられる.つまり,tight型はempty型へと構造変化し,ヌクレオチドに対する親和性が低下した結果,ADPとリン酸の遊離が起こる.一方,empty型はATPとの結合の可能なbindable型へと構造変化を起こし,A3B3複合体はヌクレオチド非結合型の状態になる方向へと進む.しかしながら,tight型とDF複合体は強く結合しているため,A3B3複合体の構造変化は抑制されると考えられ,実際には,これらの中間状態に変化すると予想している.つぎに,bindable型あるいは中間状態のABペアとATPとの結合によりA3B3複合体はヌクレオチド結合型の構造に変化し,2つのbound型にATPが結合し,これによりDF複合体は回転する.最後に,もともとあったbound型がDF複合体との相互作用によりtight型へと構造変化し,Argフィンガーがγリン酸に近づいて,はじめから120度回転したヌクレオチド結合型V1-ATPaseの構造にもどる.
おわりに
今回,腸球菌に由来するV-ATPaseについて,ヌクレオチド非結合型およびヌクレオチド結合型のA3B3複合体およびV1-ATPase(A3B3DF複合体)の結晶構造を明らかにした.
ヌクレオチド非結合型A3B3複合体の構造それ自体が非対称性をもち,その非対称性が回転の方向性を決定していることや,DF複合体との結合によりtight型への構造変化が起こりATP加水分解のトリガーとなることなどが示唆され,V1-ATPaseの新しい回転機構のモデルを提案することができた.これにより,ほかの分子モーターを含む生体エネルギー変換機構の原理の解明につながることが期待される.さらに現在,より詳細な回転機構の解明にむけ,反応中間体の結晶構造解析や分子動力学シミュレーション,1分子観察などの相関構造解析を進めている.
文 献
- Walker, J. E.: ATP synthesis by rotary catalysis (Nobel Lecture). Angew. Chem. Int. Ed. Engl., 37, 2308-2319 (1998)
- Forgac, M.: Vacuolar ATPases: rotary proton pumps in physiology and pathophysiology. Nat. Rev. Mol. Cell Biol., 8, 917-929 (2007)[PubMed]
- Abrahams, J. P., Leslie, A. G., Lutter, R. et al.: Structure at 2.8 Å resolution of F1-ATPase from bovine heart mitochondria. Nature, 370, 621-628 (1994)[PubMed]
- Noji, H., Yasuda, R., Yoshida, M. et al.: Direct observation of the rotation of F1-ATPase. Nature 386, 299-302 (1997)[PubMed]
- Maher, M. J., Akimoto, S., Iwata, M. et al.: Crystal structure of A3B3 complex of V-ATPase from Thermus thermophilus. EMBO J., 28, 3771-3779 (2009)[PubMed]
- Numoto, N., Hasegawa, Y., Takeda, K. et al.: Inter-subunit interaction and quaternary rearrangement defined by the central stalk of prokaryotic V1-ATPase. EMBO Rep., 10, 1228-1234 (2009)[PubMed]
- Imamura, H., Takeda, M., Funamoto, S. et al.: Rotation scheme of V1-motor is different from that of F1-motor. Proc. Natl. Acad. Sci. USA, 102, 17929-17933 (2005)[PubMed]
- Murata, T., Igarashi, K., Kakinuma, Y. et al.: Na+ binding of V-type Na+-ATPase in Enterococcus hirae. J. Biol. Chem., 275, 13415-13419 (2000)[PubMed]
- Arai, S., Yamato, I., Shiokawa, A. et al.: Reconstitution in vitro of the catalytic portion (NtpA3-B3-D-G complex) of Enterococcus hirae V-type Na+-ATPase. Biochem. Biophys. Res. Commun., 390, 698-702 (2009)[PubMed]
- Saijo, S., Arai, S., Hossain K. M. M. et al.: Crystal structure of the central axis DF complex of the prokaryotic V-ATPase. Proc. Natl. Acad. Sci. USA, 108, 19955-19960 (2011)[PubMed]
- Liu, Q., Leng, X. H., Newman P. R. et al.: Site-directed mutagenesis of the yeast V-ATPase A subunit. J. Biol. Chem., 272, 11750-11756 (1997)[PubMed]
- Bowler, M. W., Montgomery, M. G., Leslie, A. G. W. et al.: Ground state structure of F1-ATPase from bovine heart mitochondria at 1.9 Å resolution. J. Biol. Chem., 282, 14238-14242 (2007)[PubMed]
著者プロフィール
東京理科大学基礎工学部 助教.
山登 一郎(Ichiro Yamato)
東京理科大学基礎工学部 教授.
村田 武士(Takeshi Murata)
千葉大学大学院理学研究科 特任准教授.
研究室URL:http://murata-lab.matrix.jp/
© 2013 西條慎也・山登一郎・村田武士 Licensed under CC 表示 2.1 日本