タンパク質合成に必須なtRNA修飾の化学構造が決定された
宮内健常・鈴木 勉
(東京大学大学院工学系研究科 化学生命工学専攻)
email:宮内健常,鈴木 勉
DOI: 10.7875/first.author.2013.007
A cyclic form of N6-threonylcarbamoyladenosine as a widely distributed tRNA hypermodification.
Kenjyo Miyauchi, Satoshi Kimura, Tsutomu Suzuki
Nature Chemical Biology, 9, 105-111 (2013)
N6-スレオニルカルバモイルアデノシン(t6A)はANNコドンを解読するtRNAの37位に存在する修飾塩基で,ほぼすべての生物に保存されており多くの生物の生育に必須であって,コドンの認識における正確さや効率を維持するのに非常に大きな役割をはたしていることが知られていた.筆者らは,t6Aは大腸菌や出芽酵母において,さらに脱水環化してオキサゾロン環をもつ活性エステルであるサイクリックt6A(ct6A)を形成していることを発見した.この新規の修飾塩基は加水分解をうけやすいため,これまでの解析手法では検出できなかったが,試料の調製法を最適化することによりその検出が可能になった.とくに大腸菌にはt6Aはほとんど存在しないことが明らかになり,tRNAにはct6Aが形成されていることが判明した.ct6Aは細菌,酵母,菌類,植物,原生生物などさまざまな生物種に広く分布していた.さらに,比較ゲノム解析などによりct6Aの生合成に必須な修飾酵素TcdAを発見し,ct6Aの試験管内合成にも成功した.出芽酵母においては2つのtcdA遺伝子ホモログ,TCD1遺伝子とTCD2遺伝子がct6Aの生合成に必須であり,これらの遺伝子は非発酵性の炭素源による生育に必須なことも判明した.ルシフェラーゼを用いたレポーターアッセイにより,ct6AはtRNALysにおいてコドン認識能を補助するはたらきのあることが示された.また,リボソームA部位におけるコドンとアンチコドンとの対合の構造モデルから,ct6Aの側鎖がコドン1字目のAと水素結合している可能性が示された.t6Aは1969年に発見され,おもに大腸菌や出芽酵母を用いて研究されてきたが,これらの研究はct6Aが加水分解されたアーティファクトを研究してきたことになり,筆者らの研究は,これまでのまちがった化学構造を前提とした40年来の研究の見直しをせまるものである.
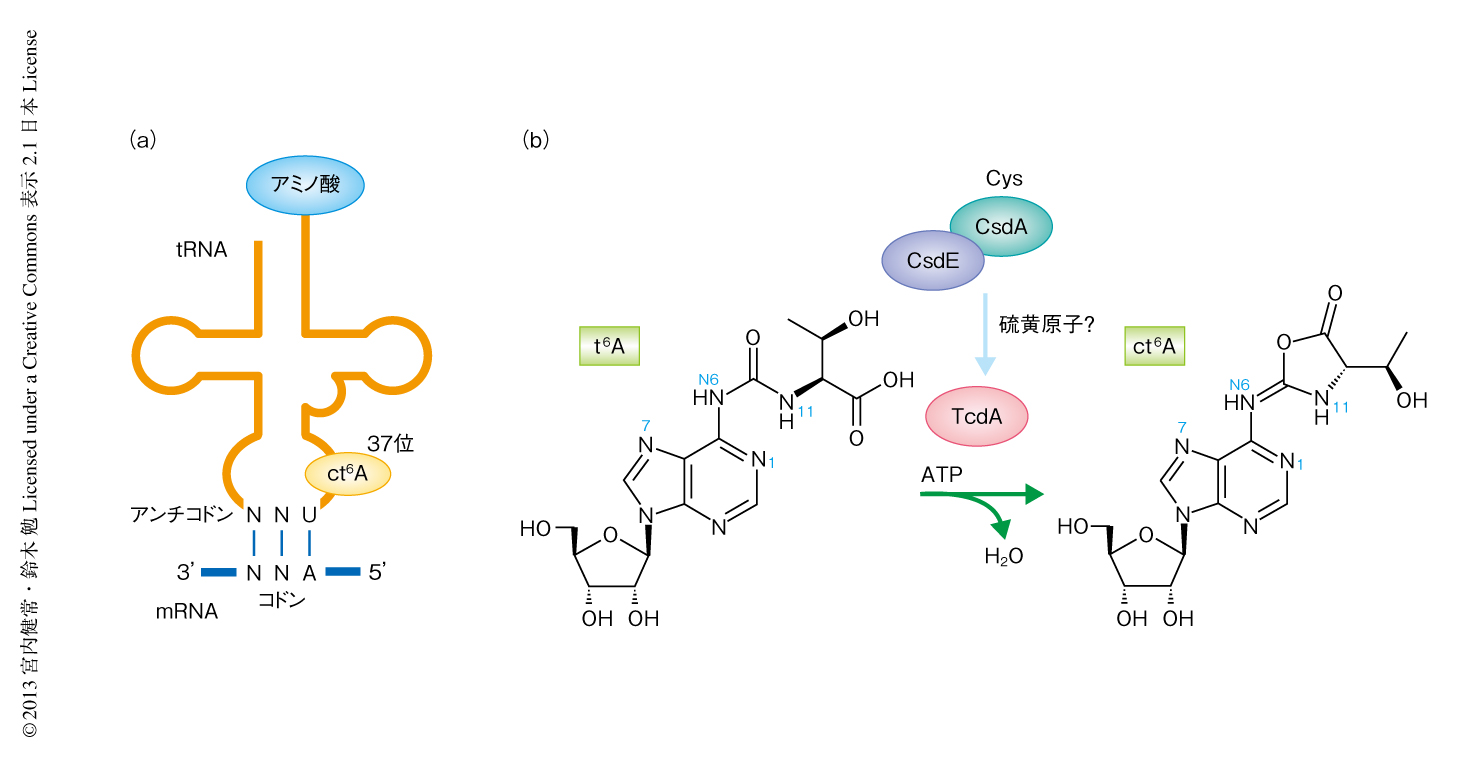
tRNAは4つの塩基により書き込まれたDNAの遺伝情報をタンパク質のアミノ酸配列へと変換するアダプター分子である.アンチコドン配列を使いmRNAの相補的なコドンと対合することにより,3’末端に結合したアミノ酸を正しく対応させている(図1a).tRNAにはさまざまな転写後修飾が施されている1).これらの修飾はtRNAのフォールディングや安定性に必要なだけでなく,遺伝暗号の解読を正確かつ効率よく行うため重要な役割を担っている2).N6-スレオニルカルバモイルアデノシン(t6A)はアデノシンの誘導体であり,アデニンのN6位にカルボニル基を介してスレオニンの結合した化学構造をもつ(図1b).1969年に,出芽酵母の全RNAから単離され化学構造が決定された3).それ以来,t6Aはさまざまな生物種から発見され,構造的にも機能的にも,もっともよく研究された修飾塩基のひとつである.t6AはANNコドンを解読するtRNAの37位(アンチコドンの3’側に隣接した部位)に存在する4).真正細菌,古細菌,真核生物のほか,ミトコンドリアや葉緑体など独自のゲノムをもつオルガネラも含め,ほぼすべての生物がもち,多くの生物の生育において必須であることが知られている.t6AはtRNAのアミノアシル化,転座反応,コドンの正確な認識,読み枠の維持など,タンパク質合成のさまざまな段階において重要な役割を担うことが明らかになっている.t6Aのかさ高い側鎖はアンチコドンループにおけるU33-A37対合を防止し,アンチコドンループが正しい構造をとるために必要である.また,リボソーム30SサブユニットのA部位におけるコドンとアンチコドンとの対合の結晶構造から,t6AはアデニンN1と側鎖N11とのあいだで分子内水素結合を形成し,これにより延長された平面構造がコドン1文字目のAおよびA38(t6Aのとなりの塩基)とスタッキングすることにより,コドンとアンチコドンとの対合を安定化している5).t6Aの研究は,40年以上ものあいだ,おもに大腸菌や出芽酵母を用いて行われてきたが,その化学構造について疑われたことはなかった.
今回の筆者らの発見を説明するまえに,1970年代に報告されたt6Aの誘導体についてふれておきたい.1972年,大腸菌のtRNAにはt6Aのマイナーな誘導体が存在することが報告された6).単離して構造を解析したところ,t6Aの側鎖にトリスヒドロキシメチルアミノメタン(Tris)がアミド結合したものであることが判明した7).この論文では,大腸菌からRNAを抽出する際に使用した緩衝液に由来するTrisが,大腸菌に存在する酵素によりt6Aに結合したものであろうと考察している.したがって,この研究を教訓として,tRNAの研究者のあいだでは昔から,tRNAを抽出する際にはTris緩衝液を用いてはならないといわれてきた.しかし筆者らは,本当にそのような酵素が大腸菌に存在するのか疑問を感じていた.
筆者らは,さまざまな生物種からtRNAや非コードRNAを単離精製し,高感度な質量分析法を用いた転写後修飾の解析を行ってきた8,9).単離した大腸菌のtRNAをRNase T1により消化し,キャピラリーLC/nanoESI質量分析法により解析した.t6Aを含むRNA断片を観測したところ,予測された質量の断片にくわえ,予測より18 Da小さい断片が見い出された.衝突誘起解離(collision induced dissociation:CID)法を用いた詳細な解析により,この18 Daは37位のt6Aから失われていることがわかった.ヌクレオシドの質量は394 Daであるため,この塩基を仮にN394とよぶことにした.精密質量の解析から18 DaはH2O 1分子の質量であることが判明し,N394はt6Aから脱水したものであると考えられた.つぎに,全RNAをヌクレオシドまで消化しN394の直接的な検出を試みたが,t6Aは感度よく検出されるのに対し,N394はまったく検出されなかった.N394はt6Aが脱水したものだとすると,全RNAの抽出の過程,あるいは,ヌクレオシドの消化の過程においてN394が分解した可能性があると考えられた.従来から一般的に行われているヌクレオシド消化法では10),ヌクレアーゼP1により消化したのち,アルカリ性条件(pH 8.2)においてアルカリホスファターゼを用い脱リン酸化する.この条件において脱リン酸の過程でN394が加水分解する可能性を考え,ヌクレオシド消化を中性条件(pH 7.0),かつ,できるだけ短い時間(1時間)で行った.その結果,驚いたことに,t6Aはほとんど検出されず,逆に,N394が効率よく検出された.このことは,大腸菌にはt6Aがほとんど存在せず,実際にはN394のかたちで存在していることを示していた.これまでN394がまったくみつからなかった原因は,一般的に広く用いられ確立されたヌクレオシド調製法に誰も疑問をもたなかったことにあった.
N394を安定同位体により標識した水H218Oにより加水分解すると,質量が2 Da増加したt6Aが検出された.この結果から,実際にN394が加水分解されることによりt6Aは生じることが証明された.さらに,18O標識されたt6Aのタンデム質量分析法による解析により,水の酸素原子はt6Aの側鎖のカルボン酸の部分に入ることが判明した.また,N394をTris緩衝液(pH 8.5)において保温したところ,t6Aのカルボキシル基にTrisがアミド結合により付加した化合物が生じた.したがって,1970年代に報告されたt6AのTris付加体は7),Tris緩衝液によりtRNAを抽出した際,N394にTrisが非酵素的に付加したものであると思われた.さらに,Trisのほかにも,エタノールアミンのような1級アミンとN394を混合するだけで同様の付加体の生じることが確認された.
安定同位体により標識したスレオニン[18O]Thrおよび[2H2]Thrを用い,大腸菌を培養することによりN394の代謝標識を行った.カルボキシル基が18O標識されたスレオニンの取り込み効率の低さから,N394が形成されるときにt6Aの側鎖のカルボキシル基から酸素原子が抜けていることが示唆された.さらに,標識されたN394の衝突誘起解離法による解析を行い,プロダクトイオンの帰属により化学構造の推定を行った.その結果,N394はt6Aのカルボニル基とカルボキシル基が脱水縮合してオキサゾロン環を形成した構造であると予測され,これをサイクリックt6A(ct6A)と名づけた(図1b).
N394がct6Aであることを確認するため,ct6Aの化学合成を試みた.t6Aにイミダゾールの存在(pH 6.0)のもと水溶性カルボジイミドを反応させることにより比較的効率よくct6Aを化学合成することができた.HPLCにより精製したのち,NMRにより1H-1H COSYスペクトル,1H-13C HSQCスペクトル,HMBCスペクトルを測定し目的物であることを確認した.大腸菌から単離したct6Aについても1H-1H COSYスペクトルを測定した.微量であったためシグナルは弱かったが,化学合成した標品と同一のクロスピークが得られた.さらに,大腸菌の全ヌクレオシドと化学合成したct6AとをLC/MS/MSに同時に注入したところ,溶出位置やMS/MSパターンが同一であったことから,N394はct6Aであることが確認された.ct6Aの構造についてはいくつかの互変異性体が可能であるが,11位にプロトンが観測され,N6位や1位にプロトンは観測されなかった.ct6Aは活性エステルの一種でありアルカリ性では容易に水と反応しt6Aを生じることから,従来の研究ではct6Aが加水分解して生じたアーティファクトを観測していたものと思われた.また,アルカリ条件においてTrisなどのアミンと容易に反応し付加体を生じる理由も判明した.
ct6Aの生合成にかかわる遺伝子を同定するため,まず,さまざまな生物におけるct6Aの有無を調べた.大腸菌,枯草菌,出芽酵母,分裂酵母,ホウレンソウにおいてct6Aが検出されたが,Mycoplasma mobile(真正細菌),Acidimicrobium ferrooxidans(真正細菌),Haloferax volcanii(古細菌),Thermoplasma acidophilum(古細菌),HeLa細胞,マウスの肝臓にはct6Aは存在しなかった.トリパノソーマにおいて2-メチルチオt6AのTris付加体の報告のあることから11),トリパノソーマなどの一部の原生生物にもct6Aがあると予想された.これらの知見をもとに比較ゲノム解析を行ったところ,大腸菌においてct6Aの生合成にかかわる候補遺伝子として82個の遺伝子が絞り込まれた.また筆者らはこれまでの研究により8,12),約2000遺伝子をカバーする大腸菌の広範囲ゲノム欠損株のヌクレオシド解析データを保持しており,ct6Aの有無を詳細に見直した結果,大腸菌株AA787においてct6Aの欠損が見い出された.この株はrelA遺伝子からargA遺伝子までの32個のORFを欠損しており,さきに述べた比較ゲノム解析により絞り込まれた候補との集合をとることにより,csdL遺伝子が第1の候補遺伝子として浮上した.実際に,csdL遺伝子破壊株ではct6Aは完全に欠損しており,ct6Aの生合成にはcsdL遺伝子が必須であることが明らかになった.CsdLはユビキチン活性化酵素E1様のタンパク質であるが明確な機能については報告がなかった.システイン脱硫酵素であるCsdAがCys残基から引き抜いた硫黄原子をCsdEを介しCsdLへと受け渡すことが知られていたため,CsdAおよびCsdEの関与を調べる目的でcsdE遺伝子破壊株およびcsdA遺伝子破壊株のヌクレオシド解析を行った.その結果,csdE遺伝子破壊株ではct6Aは2%に減少し,csdA遺伝子破壊株では12%に減少していたが,csdL遺伝子破壊株とは異なり両方の株ともct6Aは完全には欠損していなかった.したがって,CsdAやCsdEは硫黄原子の受け渡しをつうじCsdLの活性化などにかかわっていると考えられた(図1b).csdL遺伝子のホモログは,真正細菌においてはプロテオバクテリアの多くやファーミキューテスの約半分などさまざまな種に存在し,真核生物においては菌類と植物,一部の原生生物に分布していることから,ct6Aはこれらの生物種に広く分布していると考えられる.
CsdLの組換えタンパク質によりct6Aの試験管内合成を行った.基質として,csdL遺伝子破壊株から単離したtRNAAsn(37位がt6A)を用いた.CsdLはユビキチン活性化酵素E1様タンパク質であるためATPを消費すると考えた.実際に,ATP存在のもとCsdLはtRNAにあるt6Aをct6Aに変換した.したがって,CsdLはATPに依存的にt6Aを脱水しct6Aを生成する酵素であったことから,CsdLを“tRNA threonylcarbamoyladenosine dehydratase A”(TcdA)と改名した(図1b).
出芽酵母には2つのtcdA遺伝子ホモログ,TCD1遺伝子(YHR003C)とTCD2遺伝子(YKL027W)がある.それぞれの遺伝子欠損株を解析したところ,ct6Aが完全に失われていたことから,この2つの遺伝子の両方がct6Aの生合成に必須であることが判明した.また,これらの遺伝子は非発酵性の炭素源による生育に必須であることが判明し,ct6Aそれ自体,あるいは,TCD1やTCD2が別の経路において,出芽酵母の呼吸に重要なはたらきをもつことも明らかになった.
ct6Aの機能を解析するためtcdA遺伝子とほかのtRNA修飾酵素遺伝子との二重欠損株を作製したところ,tRNAの34位の2-チオウリジン修飾酵素をコードするmnmA遺伝子とtcdA遺伝子との二重欠損株において合成的な生育阻害がみられた.2-チオウリジンとct6Aの両方の修飾をもつtRNAはtRNALysだけであることから,ct6AはtRNALysの機能にかかわるものと考えられた.そこで,tRNALysのタンパク質合成におけるコドン認識能を評価するため,ルシフェラーゼを用いたレポーターアッセイを行った.ホタルルシフェラーゼの529位のLys残基は活性に必須であることが知られているため,この位置をAGAコドン(Arg)あるいはUAGコドン(終止)へと置換した変異体を作製した.これらのコンストラクトを大腸菌の野生株(ct6Aをもつ)およびtcdA遺伝子破壊株(t6Aをもつ)に導入しルシフェラーゼの酵素活性を測定することにより,リジンが529位へ誤って取り込まれる効率を評価することができる.その結果,いずれのコンストラクトにおいても野生株と比較して,tcdA遺伝子破壊株におけるリジンの取り込みは顕著に低下することが判明した.すなわち,ct6AはtRNALysのコドン認識能をさらに向上させる効果のあることが示された.実際に,大腸菌においては11種類のtRNAにct6Aが存在しており,ct6AはほかのtRNAにおいても効率のよいコドン認識に関与しているのではないかと考えられる.
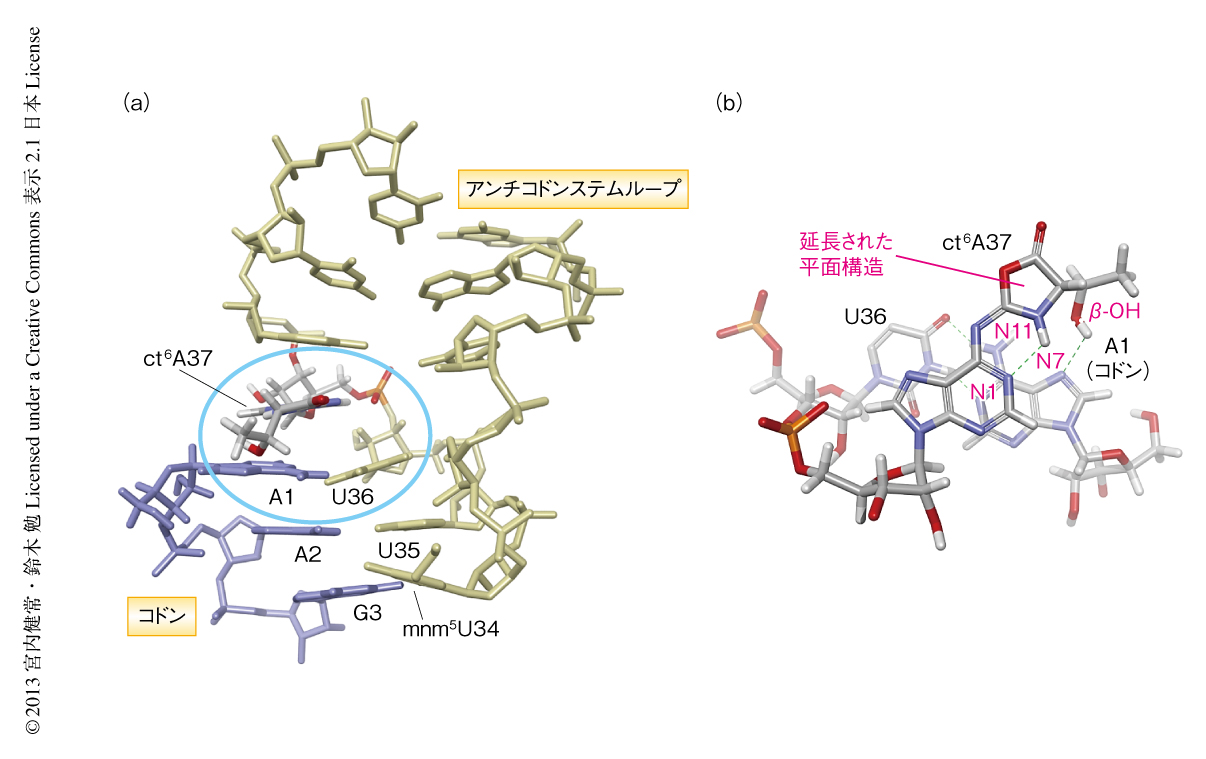
ct6Aがどのような機構によりtRNAによるコドン認識に関与しているかを理解するため,リボソームA部位におけるct6Aの構造モデルを構築した(図2).好熱菌Thermus thermophilusのリボソーム30Sサブユニットにおけるアンチコドンステムループ(t6Aをもつ)とコドンの結晶構造をもとに5),t6Aをct6Aに置換した構造モデルを構築した(図2a).t6AにみられるN1とN11とのあいだの分子内水素結合(図1b)はct6Aにおいても保存されていると考えられたこと,N6位に二重結合があることから,アデニン環からオキサゾロン環までの大きな平面構造がより安定に形成されていると予測された(図2b).これにより,コドン1字目のAとのスタッキングがより強化されると考えられた.また,スレオニンがL体であることを考慮すると,側鎖のヒドロキシエチル基がオキサゾロン環からアキシアル方向(コドン1文字目の方向)へと延び,β-OH基がコドン1字目AのN7位と水素結合する可能性が示唆された(図2b).延長した平面構造によるスタッキング相互作用にくわえ,コドン1文字目との追加的な水素結合が,ct6Aによるコドン認識を補助するはたらきの分子基盤であると考えられた.
この研究は,40年間にわたり信じられてきたRNA修飾の化学構造が,実は生体においてはまったく異なる構造をとっていたことを示したものであり,これまでのt6Aの構造を前提とした関連研究の見直しをせまるものである.また,生体にはしばしば化学的に不安定な分子種が存在し,抽出方法や解析方法によってはまちがった構造をもとに研究が進められている例があることを示した.生命を正しく理解するためには,生体を構成する分子の化学構造を正しく把握しなければならず,ふだん何気なく使用している汎用的な実験手法やプロトコールに問題がないか疑ってみることも,ときには必要なのかもしれない.
略歴:2007年 東京大学大学院新領域創成科学研究科博士課程 修了,2006年 バイオ産業情報化コンソーシアム 特別研究員を経て,2010年 東京大学大学院工学系研究科 特任研究員.
研究テーマ:RNA修飾,RNAの単離精製法.
関心事:細胞における生体分子の真の構造を明らかにしたい.
鈴木 勉(Tsutomu Suzuki)
東京大学大学院工学系研究科 教授.
研究室URL:http://rna.chem.t.u-tokyo.ac.jp/index_j.html
© 2013 宮内健常・鈴木 勉 Licensed under CC 表示 2.1 日本
(東京大学大学院工学系研究科 化学生命工学専攻)
email:宮内健常,鈴木 勉
DOI: 10.7875/first.author.2013.007
A cyclic form of N6-threonylcarbamoyladenosine as a widely distributed tRNA hypermodification.
Kenjyo Miyauchi, Satoshi Kimura, Tsutomu Suzuki
Nature Chemical Biology, 9, 105-111 (2013)
要 約
N6-スレオニルカルバモイルアデノシン(t6A)はANNコドンを解読するtRNAの37位に存在する修飾塩基で,ほぼすべての生物に保存されており多くの生物の生育に必須であって,コドンの認識における正確さや効率を維持するのに非常に大きな役割をはたしていることが知られていた.筆者らは,t6Aは大腸菌や出芽酵母において,さらに脱水環化してオキサゾロン環をもつ活性エステルであるサイクリックt6A(ct6A)を形成していることを発見した.この新規の修飾塩基は加水分解をうけやすいため,これまでの解析手法では検出できなかったが,試料の調製法を最適化することによりその検出が可能になった.とくに大腸菌にはt6Aはほとんど存在しないことが明らかになり,tRNAにはct6Aが形成されていることが判明した.ct6Aは細菌,酵母,菌類,植物,原生生物などさまざまな生物種に広く分布していた.さらに,比較ゲノム解析などによりct6Aの生合成に必須な修飾酵素TcdAを発見し,ct6Aの試験管内合成にも成功した.出芽酵母においては2つのtcdA遺伝子ホモログ,TCD1遺伝子とTCD2遺伝子がct6Aの生合成に必須であり,これらの遺伝子は非発酵性の炭素源による生育に必須なことも判明した.ルシフェラーゼを用いたレポーターアッセイにより,ct6AはtRNALysにおいてコドン認識能を補助するはたらきのあることが示された.また,リボソームA部位におけるコドンとアンチコドンとの対合の構造モデルから,ct6Aの側鎖がコドン1字目のAと水素結合している可能性が示された.t6Aは1969年に発見され,おもに大腸菌や出芽酵母を用いて研究されてきたが,これらの研究はct6Aが加水分解されたアーティファクトを研究してきたことになり,筆者らの研究は,これまでのまちがった化学構造を前提とした40年来の研究の見直しをせまるものである.
はじめに
tRNAは4つの塩基により書き込まれたDNAの遺伝情報をタンパク質のアミノ酸配列へと変換するアダプター分子である.アンチコドン配列を使いmRNAの相補的なコドンと対合することにより,3’末端に結合したアミノ酸を正しく対応させている(図1a).tRNAにはさまざまな転写後修飾が施されている1).これらの修飾はtRNAのフォールディングや安定性に必要なだけでなく,遺伝暗号の解読を正確かつ効率よく行うため重要な役割を担っている2).N6-スレオニルカルバモイルアデノシン(t6A)はアデノシンの誘導体であり,アデニンのN6位にカルボニル基を介してスレオニンの結合した化学構造をもつ(図1b).1969年に,出芽酵母の全RNAから単離され化学構造が決定された3).それ以来,t6Aはさまざまな生物種から発見され,構造的にも機能的にも,もっともよく研究された修飾塩基のひとつである.t6AはANNコドンを解読するtRNAの37位(アンチコドンの3’側に隣接した部位)に存在する4).真正細菌,古細菌,真核生物のほか,ミトコンドリアや葉緑体など独自のゲノムをもつオルガネラも含め,ほぼすべての生物がもち,多くの生物の生育において必須であることが知られている.t6AはtRNAのアミノアシル化,転座反応,コドンの正確な認識,読み枠の維持など,タンパク質合成のさまざまな段階において重要な役割を担うことが明らかになっている.t6Aのかさ高い側鎖はアンチコドンループにおけるU33-A37対合を防止し,アンチコドンループが正しい構造をとるために必要である.また,リボソーム30SサブユニットのA部位におけるコドンとアンチコドンとの対合の結晶構造から,t6AはアデニンN1と側鎖N11とのあいだで分子内水素結合を形成し,これにより延長された平面構造がコドン1文字目のAおよびA38(t6Aのとなりの塩基)とスタッキングすることにより,コドンとアンチコドンとの対合を安定化している5).t6Aの研究は,40年以上ものあいだ,おもに大腸菌や出芽酵母を用いて行われてきたが,その化学構造について疑われたことはなかった.
1.t6Aに緩衝液に由来するTrisが結合するという奇妙な話
今回の筆者らの発見を説明するまえに,1970年代に報告されたt6Aの誘導体についてふれておきたい.1972年,大腸菌のtRNAにはt6Aのマイナーな誘導体が存在することが報告された6).単離して構造を解析したところ,t6Aの側鎖にトリスヒドロキシメチルアミノメタン(Tris)がアミド結合したものであることが判明した7).この論文では,大腸菌からRNAを抽出する際に使用した緩衝液に由来するTrisが,大腸菌に存在する酵素によりt6Aに結合したものであろうと考察している.したがって,この研究を教訓として,tRNAの研究者のあいだでは昔から,tRNAを抽出する際にはTris緩衝液を用いてはならないといわれてきた.しかし筆者らは,本当にそのような酵素が大腸菌に存在するのか疑問を感じていた.
2.新規の修飾塩基N394の発見とその反応性
筆者らは,さまざまな生物種からtRNAや非コードRNAを単離精製し,高感度な質量分析法を用いた転写後修飾の解析を行ってきた8,9).単離した大腸菌のtRNAをRNase T1により消化し,キャピラリーLC/nanoESI質量分析法により解析した.t6Aを含むRNA断片を観測したところ,予測された質量の断片にくわえ,予測より18 Da小さい断片が見い出された.衝突誘起解離(collision induced dissociation:CID)法を用いた詳細な解析により,この18 Daは37位のt6Aから失われていることがわかった.ヌクレオシドの質量は394 Daであるため,この塩基を仮にN394とよぶことにした.精密質量の解析から18 DaはH2O 1分子の質量であることが判明し,N394はt6Aから脱水したものであると考えられた.つぎに,全RNAをヌクレオシドまで消化しN394の直接的な検出を試みたが,t6Aは感度よく検出されるのに対し,N394はまったく検出されなかった.N394はt6Aが脱水したものだとすると,全RNAの抽出の過程,あるいは,ヌクレオシドの消化の過程においてN394が分解した可能性があると考えられた.従来から一般的に行われているヌクレオシド消化法では10),ヌクレアーゼP1により消化したのち,アルカリ性条件(pH 8.2)においてアルカリホスファターゼを用い脱リン酸化する.この条件において脱リン酸の過程でN394が加水分解する可能性を考え,ヌクレオシド消化を中性条件(pH 7.0),かつ,できるだけ短い時間(1時間)で行った.その結果,驚いたことに,t6Aはほとんど検出されず,逆に,N394が効率よく検出された.このことは,大腸菌にはt6Aがほとんど存在せず,実際にはN394のかたちで存在していることを示していた.これまでN394がまったくみつからなかった原因は,一般的に広く用いられ確立されたヌクレオシド調製法に誰も疑問をもたなかったことにあった.
N394を安定同位体により標識した水H218Oにより加水分解すると,質量が2 Da増加したt6Aが検出された.この結果から,実際にN394が加水分解されることによりt6Aは生じることが証明された.さらに,18O標識されたt6Aのタンデム質量分析法による解析により,水の酸素原子はt6Aの側鎖のカルボン酸の部分に入ることが判明した.また,N394をTris緩衝液(pH 8.5)において保温したところ,t6Aのカルボキシル基にTrisがアミド結合により付加した化合物が生じた.したがって,1970年代に報告されたt6AのTris付加体は7),Tris緩衝液によりtRNAを抽出した際,N394にTrisが非酵素的に付加したものであると思われた.さらに,Trisのほかにも,エタノールアミンのような1級アミンとN394を混合するだけで同様の付加体の生じることが確認された.
3.サイクリックt6Aの発見
安定同位体により標識したスレオニン[18O]Thrおよび[2H2]Thrを用い,大腸菌を培養することによりN394の代謝標識を行った.カルボキシル基が18O標識されたスレオニンの取り込み効率の低さから,N394が形成されるときにt6Aの側鎖のカルボキシル基から酸素原子が抜けていることが示唆された.さらに,標識されたN394の衝突誘起解離法による解析を行い,プロダクトイオンの帰属により化学構造の推定を行った.その結果,N394はt6Aのカルボニル基とカルボキシル基が脱水縮合してオキサゾロン環を形成した構造であると予測され,これをサイクリックt6A(ct6A)と名づけた(図1b).
N394がct6Aであることを確認するため,ct6Aの化学合成を試みた.t6Aにイミダゾールの存在(pH 6.0)のもと水溶性カルボジイミドを反応させることにより比較的効率よくct6Aを化学合成することができた.HPLCにより精製したのち,NMRにより1H-1H COSYスペクトル,1H-13C HSQCスペクトル,HMBCスペクトルを測定し目的物であることを確認した.大腸菌から単離したct6Aについても1H-1H COSYスペクトルを測定した.微量であったためシグナルは弱かったが,化学合成した標品と同一のクロスピークが得られた.さらに,大腸菌の全ヌクレオシドと化学合成したct6AとをLC/MS/MSに同時に注入したところ,溶出位置やMS/MSパターンが同一であったことから,N394はct6Aであることが確認された.ct6Aの構造についてはいくつかの互変異性体が可能であるが,11位にプロトンが観測され,N6位や1位にプロトンは観測されなかった.ct6Aは活性エステルの一種でありアルカリ性では容易に水と反応しt6Aを生じることから,従来の研究ではct6Aが加水分解して生じたアーティファクトを観測していたものと思われた.また,アルカリ条件においてTrisなどのアミンと容易に反応し付加体を生じる理由も判明した.
4.ct6Aはt6A脱水酵素TcdAにより生合成される
ct6Aの生合成にかかわる遺伝子を同定するため,まず,さまざまな生物におけるct6Aの有無を調べた.大腸菌,枯草菌,出芽酵母,分裂酵母,ホウレンソウにおいてct6Aが検出されたが,Mycoplasma mobile(真正細菌),Acidimicrobium ferrooxidans(真正細菌),Haloferax volcanii(古細菌),Thermoplasma acidophilum(古細菌),HeLa細胞,マウスの肝臓にはct6Aは存在しなかった.トリパノソーマにおいて2-メチルチオt6AのTris付加体の報告のあることから11),トリパノソーマなどの一部の原生生物にもct6Aがあると予想された.これらの知見をもとに比較ゲノム解析を行ったところ,大腸菌においてct6Aの生合成にかかわる候補遺伝子として82個の遺伝子が絞り込まれた.また筆者らはこれまでの研究により8,12),約2000遺伝子をカバーする大腸菌の広範囲ゲノム欠損株のヌクレオシド解析データを保持しており,ct6Aの有無を詳細に見直した結果,大腸菌株AA787においてct6Aの欠損が見い出された.この株はrelA遺伝子からargA遺伝子までの32個のORFを欠損しており,さきに述べた比較ゲノム解析により絞り込まれた候補との集合をとることにより,csdL遺伝子が第1の候補遺伝子として浮上した.実際に,csdL遺伝子破壊株ではct6Aは完全に欠損しており,ct6Aの生合成にはcsdL遺伝子が必須であることが明らかになった.CsdLはユビキチン活性化酵素E1様のタンパク質であるが明確な機能については報告がなかった.システイン脱硫酵素であるCsdAがCys残基から引き抜いた硫黄原子をCsdEを介しCsdLへと受け渡すことが知られていたため,CsdAおよびCsdEの関与を調べる目的でcsdE遺伝子破壊株およびcsdA遺伝子破壊株のヌクレオシド解析を行った.その結果,csdE遺伝子破壊株ではct6Aは2%に減少し,csdA遺伝子破壊株では12%に減少していたが,csdL遺伝子破壊株とは異なり両方の株ともct6Aは完全には欠損していなかった.したがって,CsdAやCsdEは硫黄原子の受け渡しをつうじCsdLの活性化などにかかわっていると考えられた(図1b).csdL遺伝子のホモログは,真正細菌においてはプロテオバクテリアの多くやファーミキューテスの約半分などさまざまな種に存在し,真核生物においては菌類と植物,一部の原生生物に分布していることから,ct6Aはこれらの生物種に広く分布していると考えられる.
CsdLの組換えタンパク質によりct6Aの試験管内合成を行った.基質として,csdL遺伝子破壊株から単離したtRNAAsn(37位がt6A)を用いた.CsdLはユビキチン活性化酵素E1様タンパク質であるためATPを消費すると考えた.実際に,ATP存在のもとCsdLはtRNAにあるt6Aをct6Aに変換した.したがって,CsdLはATPに依存的にt6Aを脱水しct6Aを生成する酵素であったことから,CsdLを“tRNA threonylcarbamoyladenosine dehydratase A”(TcdA)と改名した(図1b).
出芽酵母には2つのtcdA遺伝子ホモログ,TCD1遺伝子(YHR003C)とTCD2遺伝子(YKL027W)がある.それぞれの遺伝子欠損株を解析したところ,ct6Aが完全に失われていたことから,この2つの遺伝子の両方がct6Aの生合成に必須であることが判明した.また,これらの遺伝子は非発酵性の炭素源による生育に必須であることが判明し,ct6Aそれ自体,あるいは,TCD1やTCD2が別の経路において,出芽酵母の呼吸に重要なはたらきをもつことも明らかになった.
5.ct6AはtRNALysの翻訳効率を向上させる
ct6Aの機能を解析するためtcdA遺伝子とほかのtRNA修飾酵素遺伝子との二重欠損株を作製したところ,tRNAの34位の2-チオウリジン修飾酵素をコードするmnmA遺伝子とtcdA遺伝子との二重欠損株において合成的な生育阻害がみられた.2-チオウリジンとct6Aの両方の修飾をもつtRNAはtRNALysだけであることから,ct6AはtRNALysの機能にかかわるものと考えられた.そこで,tRNALysのタンパク質合成におけるコドン認識能を評価するため,ルシフェラーゼを用いたレポーターアッセイを行った.ホタルルシフェラーゼの529位のLys残基は活性に必須であることが知られているため,この位置をAGAコドン(Arg)あるいはUAGコドン(終止)へと置換した変異体を作製した.これらのコンストラクトを大腸菌の野生株(ct6Aをもつ)およびtcdA遺伝子破壊株(t6Aをもつ)に導入しルシフェラーゼの酵素活性を測定することにより,リジンが529位へ誤って取り込まれる効率を評価することができる.その結果,いずれのコンストラクトにおいても野生株と比較して,tcdA遺伝子破壊株におけるリジンの取り込みは顕著に低下することが判明した.すなわち,ct6AはtRNALysのコドン認識能をさらに向上させる効果のあることが示された.実際に,大腸菌においては11種類のtRNAにct6Aが存在しており,ct6AはほかのtRNAにおいても効率のよいコドン認識に関与しているのではないかと考えられる.
6.リボソームA部位におけるct6Aの構造モデル
ct6Aがどのような機構によりtRNAによるコドン認識に関与しているかを理解するため,リボソームA部位におけるct6Aの構造モデルを構築した(図2).好熱菌Thermus thermophilusのリボソーム30Sサブユニットにおけるアンチコドンステムループ(t6Aをもつ)とコドンの結晶構造をもとに5),t6Aをct6Aに置換した構造モデルを構築した(図2a).t6AにみられるN1とN11とのあいだの分子内水素結合(図1b)はct6Aにおいても保存されていると考えられたこと,N6位に二重結合があることから,アデニン環からオキサゾロン環までの大きな平面構造がより安定に形成されていると予測された(図2b).これにより,コドン1字目のAとのスタッキングがより強化されると考えられた.また,スレオニンがL体であることを考慮すると,側鎖のヒドロキシエチル基がオキサゾロン環からアキシアル方向(コドン1文字目の方向)へと延び,β-OH基がコドン1字目AのN7位と水素結合する可能性が示唆された(図2b).延長した平面構造によるスタッキング相互作用にくわえ,コドン1文字目との追加的な水素結合が,ct6Aによるコドン認識を補助するはたらきの分子基盤であると考えられた.
おわりに
この研究は,40年間にわたり信じられてきたRNA修飾の化学構造が,実は生体においてはまったく異なる構造をとっていたことを示したものであり,これまでのt6Aの構造を前提とした関連研究の見直しをせまるものである.また,生体にはしばしば化学的に不安定な分子種が存在し,抽出方法や解析方法によってはまちがった構造をもとに研究が進められている例があることを示した.生命を正しく理解するためには,生体を構成する分子の化学構造を正しく把握しなければならず,ふだん何気なく使用している汎用的な実験手法やプロトコールに問題がないか疑ってみることも,ときには必要なのかもしれない.
文 献
- Juhling, F., Morl, M., Hartmann, R. K. et al.: tRNAdb 2009: compilation of tRNA sequences and tRNA genes. Nucleic Acids Res., 37, D159-D162 (2009)[PubMed]
- Suzuki, T.: Biosynthesis and function of tRNA wobble modifications. in Fine-Tuning of RNA Functions by Modification and Editing (Grosjean, H. ed.). pp. 24-69, Springer-Verlag, New York (2005)
- Schweizer, M. P., Chheda, G. B., Baczynskyj, L. et al.: Aminoacyl nucleosides. VII. N-(purin-6-ylcarbamoyl)threonine. A new component of transfer ribonucleic acid. Biochemistry, 8, 3283-3289 (1969)[PubMed]
- El Yacoubi, B., Bailly, M. & de Crecy-Lagard, V.: Biosynthesis and function of posttranscriptional modifications of transfer RNAs. Annu. Rev. Genet., 46, 69-95 (2012)[PubMed]
- Murphy, F. V., Ramakrishnan, V., Malkiewicz, A. et al.: The role of modifications in codon discrimination by tRNALysUUU. Nat. Struct. Mol. Biol., 11, 1186-1191 (2004)[PubMed]
- Nishimura, S.: Minor components in transfer RNA: their characterization, location, and function. Prog. Nucleic Acid Res. Mol. Biol., 12, 49-85 (1972)[PubMed]
- Kasai, H., Murao, K., Nishimura, S. et al.: Structure determination of a modified nucleoside isolated from Escherichia coli transfer ribonucleic acid N-[N-[(9-β-D-ribofuranosylpurin-6-yl)carbamoyl]threonyl]2-amido-2-hydroxymethylpropane-1,3-diol. Eur. J. Biochem., 69, 435-444 (1976)
- Suzuki, T., Ikeuchi, Y., Noma, A. et al.: Mass spectrometric identification and characterization of RNA-modifying enzymes. Methods Enzymol., 425, 211-229 (2007)[PubMed]
- Ikeuchi, Y., Kimura, S., Numata, T. et al.: Agmatine-conjugated cytidine in a tRNA anticodon is essential for AUA decoding in archaea. Nat. Chem. Biol., 6, 277-282 (2010)[PubMed]
- Crain, P. F.: Preparation and enzymatic hydrolysis of DNA and RNA for mass spectrometry. Methods Enzymol., 193, 782-790 (1990)[PubMed]
- Krog, J. S.: 3-(3-amino-3-carboxypropyl)-5,6-dihydrouridine is one of two novel post-transcriptional modifications in tRNALys(UUU) from Trypanosoma brucei. FEBS J., 278, 4782-4796 (2011)[PubMed]
- Ikeuchi, Y., Shigi, N., Kato, J. et al.: Mechanistic insights into sulfur relay by multiple sulfur mediators involved in thiouridine biosynthesis at tRNA wobble positions. Mol. Cell, 21, 97-108 (2006)[PubMed]
著者プロフィール
略歴:2007年 東京大学大学院新領域創成科学研究科博士課程 修了,2006年 バイオ産業情報化コンソーシアム 特別研究員を経て,2010年 東京大学大学院工学系研究科 特任研究員.
研究テーマ:RNA修飾,RNAの単離精製法.
関心事:細胞における生体分子の真の構造を明らかにしたい.
鈴木 勉(Tsutomu Suzuki)
東京大学大学院工学系研究科 教授.
研究室URL:http://rna.chem.t.u-tokyo.ac.jp/index_j.html
© 2013 宮内健常・鈴木 勉 Licensed under CC 表示 2.1 日本