ユビキチン様タンパク質活性化酵素Atg7は2つのユビキチン様タンパク質結合酵素Atg3とAtg10とを区別しない
山口雅也1・野田展生2・稲垣冬彦1
(1北海道大学大学院先端生命科学研究院 次世代ポストゲノム研究センター,2微生物化学研究会微生物化学研究所 分子構造解析部)
email:山口雅也,野田展生,稲垣冬彦
DOI: 10.7875/first.author.2012.148
Noncanonical recognition and UBL loading of distinct E2s by autophagy-essential Atg7.
Masaya Yamaguchi, Kazuaki Matoba, Ryoko Sawada, Yuko Fujioka, Hitoshi Nakatogawa, Hayashi Yamamoto, Yoshihiro Kobashigawa, Hisashi Hoshida, Rinji Akada, Yoshinori Ohsumi, Nobuo N Noda, Fuyuhiko Inagaki
Nature Structural & Molecular Biology, 19, 1250-1256 (2012)
オートファジーにはAtg8結合系とAtg12結合系の2つのユビキチン様タンパク質結合系が必須である.この2つの結合系では,オートファジーにおける唯一のユビキチン様タンパク質活性化酵素であるAtg7が共通して機能する.Atg7はAtg8結合系におけるAtg3とAtg12結合系におけるAtg10の,2つのユビキチン様タンパク質結合酵素を認識しなければならない.しかし,実際にAtg7がいかにしてこの2つのユビキチン様タンパク質結合酵素に正しい組合せのユビキチン様タンパク質を受け渡すのかについては明らかにされていなかった.今回,筆者らは,Atg7のN末端ドメインとAtg10との複合体,および,Atg7のN末端ドメインとAtg3との複合体の結晶構造を決定した.これらの構造をAtg7ホモ二量体の結晶構造と重ね合わせることにより,ユビキチン様タンパク質活性化酵素Atg7はユビキチン様タンパク質Atg8とAtg12をユビキチン様タンパク質結合酵素Atg3とAtg10に,トランス機構により受け渡す可能性を見い出し,これをin vitroにおける実験により確認した.さらに,in vitroにおいてAtg7は,Atg8とAtg12をAtg3とAtg10にランダムに受け渡すことが示された.
オートファジーは真核生物に広く保存された細胞内タンパク質分解経路である.オートファジーでは飢餓に応答して細胞質に忽然と出現した隔離膜がタンパク質,細胞質成分,オルガネラ,細胞に侵入した細菌などさまざまな対象をとりかこみ,二重膜からなる構造体であるオートファゴソームを形成する.オートファゴソームは最終的に,酵母や植物では液胞と,哺乳細胞ではリソソームと,外膜において膜融合し,内容物を内膜ごと液胞あるいはリソソームへと放出し分解をうける1).このオートファゴソームの形成過程にはAtg8結合系とAtg12結合系の2つのユビキチン様タンパク質結合系が必要である2,3).オートファジーに特異的なユビキチン様タンパク質活性化酵素(E1様酵素)であるAtg7はATPに依存的にユビキチン様タンパク質であるAtg8とAtg12を,オートファジーに特異的なユビキチン様タンパク質結合酵素(E2様酵素)であるAtg3とAtg10にそれぞれ受け渡す.Atg8とAtg12は最終的にホスファチジルエタノールアミンおよびAtg5とそれぞれ結合体を形成し,オートファジーにおいて重要な役割をはたす4,5)(出芽酵母におけるオートファジー研究については,荒木保弘・大隅良典, 領域融合レビュー, 1, e005, 2012 も参照されたい).
2011年,筆者らを含む複数のグループにより,出芽酵母Atg7全長の結晶構造,Atg7とAtg8との複合体の結晶構造,Atg7とAtg3フレキシブル領域との複合体の結晶構造が報告された6-8).Atg7はおもにN末端ドメインとC末端ドメインの2つのドメインから構成されており,Atg7はN末端ドメインを用いてAtg3を,C末端ドメインを用いてAtg8を認識することが明らかになった.また,Atg7はC末端ドメインにおいてホモ二量体を形成することから,2つのユビキチン様タンパク質および2つのE2様酵素と相互作用することができると考えられている.in vitroにおける生化学的な実験から,Atg8結合系において,Atg7はホモ二量体における同一の分子においてAtg8をAtg3へ受け渡すシス機構ではなく,Atg8をホモ二量体においてもうひとつの別の分子と結合したAtg3へ受け渡すトランス機構により進行することが示された.しかしながら,Atg7がどのようにAtg3を認識してAtg8を受け渡すのかについては明らかになっておらず,さらに,Atg12結合系におけるAtg7の役割についても不明な点が多い.
筆者らは,Atg7のN末端ドメインとAtg10との複合体,および,Atg7のN末端ドメインとAtg3との複合体の結晶構造を決定し,構造情報にもとづき種々の生化学的な実験を行った結果,Atg7とAtg10との相互作用およびAtg7とAtg3との相互作用は既知のE1様酵素とE2様酵素との相互作用とはまったく異なるものであること,in vitroにおいて,E1様酵素Atg7はE2様酵素Atg3とAtg10とを厳密に区別せずにユビキチン様タンパク質Atg8とAtg12を受け渡すことを明らかにした.
E1様酵素Atg7のN末端ドメインはAtg8結合系におけるAtg8のAtg7からAtg3への受け渡し,および,Atg12結合系におけるAtg12のAtg7からAtg10への受け渡しに必要であるかどうか,耐熱性酵母Kluyveromyces marxianusに由来するKmAtg7,KmAtg10,KmAtg5,および,シロイヌナズナに由来するAtAtg12bを利用したin vitroにおけるAtg12結合系の再構成系により検討した9).その結果,KmAtg7のC末端ドメインはAtAtg12bの活性化に十分であること,KmAtg7のN末端ドメインはAtAtg12bのKmAtg10へのトランス機構によるチオエステル化反応に必要であることが示唆された.このことから,E1様酵素Atg7のN末端ドメインとC末端ドメインの役割はAtg8結合系とAtg12結合系とで共通であることが示された.
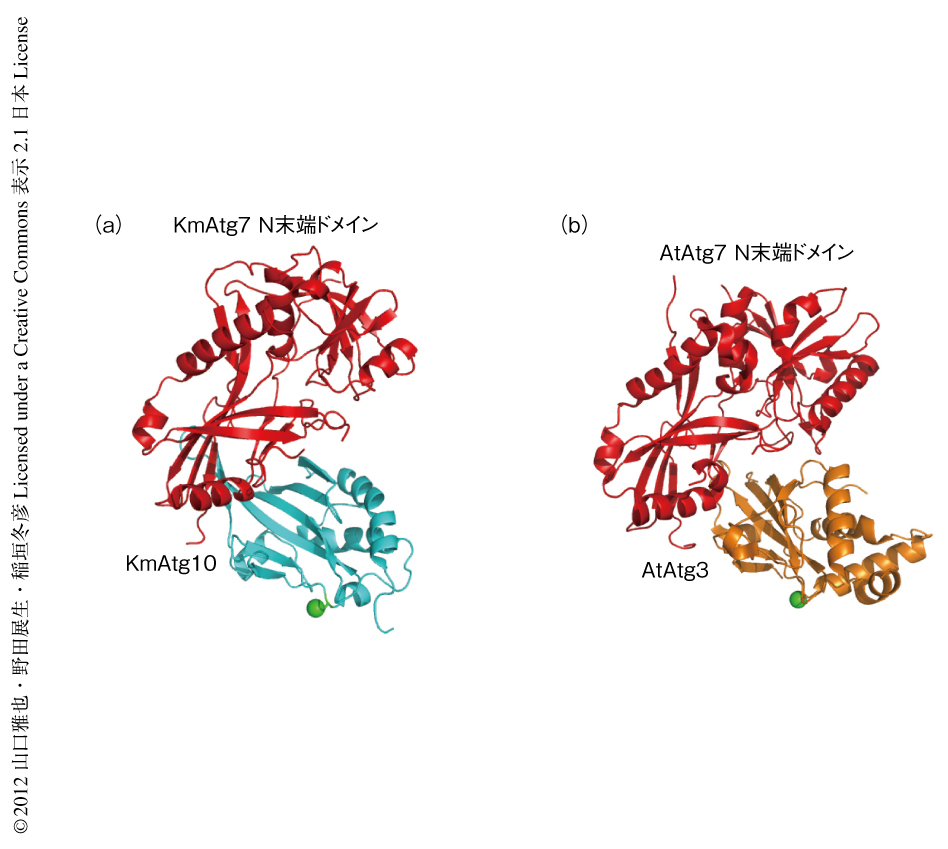
KmAtg7のN末端ドメインとKmAtg10全長との複合体の結晶構造を分解能3.2Åで決定した(図1a,PDB ID:3VX7).KmAtg7のN末端ドメインはすでに構造の決定されている出芽酵母Atg7のN末端ドメインとほとんど同一の構造を保持しており,15本のβストランドと6本のαヘリックスから構成されていた.KmAtg10はKmAtg7 N末端ドメインのC末端側の領域と結合しておりL字型の構造を形成していた.おもな相互作用はKmAtg7 N末端ドメインのβ15とKmAtg10のβ4’とのあいだの分子間βシートであった.E1様酵素とE2様酵素との古典的な相互作用では,E1様酵素のユビキチン様ドメインとE2様酵素のN末端側にあるαヘリックスおよびβ1とβ2とのあいだのループ領域とが相互作用する.したがって,KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造でみられたE1様酵素-E2様酵素相互作用は,古典的なE1様酵素-E2様酵素相互作用とはまったく異なることがわかった.
KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造にもとづき11種のKmAtg7変異体と7種のKmAtg10変異体を作製し,KmAtg7からKmAtg10へのAtAtg12bのトランス機構によるチオエステル化反応への影響を評価した.その結果,野生型を用いた場合と比較して,ほぼすべての変異体においてトランスチオエステル化反応の効率は低下した.とくに,KmAtg7のβ15に変異を導入した場合にトランスチオエステル化反応の効率は大きく減弱した.以上の結果より,KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造においてみられた相互作用は,実際にAtg12結合系におけるE1様酵素KmAtg7からE2様酵素KmAtg10へのユビキチン様タンパク質AtAtg12bの受け渡しに重要であることが示された.
E1様酵素Atg7はAtg10にくわえもうひとつのE2様酵素であるAtg3を認識する.Atg10とAtg3とのあいだの配列相同性は低いことから,Atg7のN末端ドメインはどのようにして2つのE2様酵素を認識するのかは不明であった.これまでに,Atg7のN末端ドメインとAtg3のフレキシブル領域の一部のペプチドとの複合体の結晶構造が報告されていたが,この相互作用のみではトランスチオエステル化反応におけるAtg7とAtg3の活性化システイン残基どうしの適切な配向は決まらないと考えられた7).
そこで,Atg8結合系におけるトランスチオエステル化反応の際のAtg3とAtg7との直接の相互作用を知るため,AtAtg7のN末端ドメインとAtAtg3との複合体の結晶構造を分解能3.1Åで決定した(図1b,PDB ID:3VX8).予測に反して,出芽酵母Atg7のN末端ドメインと出芽酵母Atg3のフレキシブル領域との複合体の結晶構造においてみられた相互作用はみられなかった.しかしながら,AtAtg7のN末端ドメインにおいて表面の別の領域になんらかの電子密度が確認され,また,in vitroにおけるプルダウンアッセイによりAtAtg3のフレキシブル領域はAtAtg7とAtAtg3との相互作用に必要であることが確認された.このことから,AtAtg3もAtAtg7のN末端ドメインと直接的な相互作用を形成すると考えられた.
興味深いことに,AtAtg3のコア領域はAtAtg7のN末端ドメインのC末端側の領域と結合しており,KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造と同様にL字型の構造を形成していた.AtAtg7のN末端ドメインとAtAtg3のコア領域とのおもな相互作用もKmAtg7 N末端ドメイン-KmAtg10相互作用と類似しており,AtAtg7のN末端ドメインのβ15とAtAtg3のβ4のうしろのループ(β4ループ)とが相互作用していた.しかし,この相互作用はKmAtg7 N末端ドメイン-KmAtg10複合体でみられた分子間βシートではなく,側鎖を介した疎水性相互作用からなっていた.この相互作用にくわえ,AtAtg7のN末端ドメインのβ1およびβ3の手前のループとAtAtg3のAsn77およびArg219とのあいだにも相互作用がみられた.
これらの相互作用の重要性を確認するため,AtAtg7のN末端ドメインのβ15とAtAtg3のβ4ループの三点変異体とAtAtg3のAsn77点変異体を作製した.in vitroにおけるプルダウンアッセイの結果,これらの変異体を用いた場合にはAtAtg7とAtAtg3とのあいだの結合が弱くなっていることが確認された.さらに,AtAtg7からAtAtg3へのAtAtg8aのトランス機構によるチオエステル化反応への影響を検討した結果,すべての変異体において反応の効率は低下していた.以上の結果から,AtAtg7 N末端ドメイン-AtAtg3複合体の結晶構造でみられた相互作用は,Atg8結合系におけるE1様酵素AtAtg7とE1様酵素AtAtg3との結合,ならびに,AtAtg7からAtAtg3へのユビキチン様タンパク質AtAtg8aの受け渡しにおいて重要であることが示された.
KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造とAtAtg7 N末端ドメイン-AtAtg3複合体の結晶構造を出芽酵母Atg7全長の結晶構造と重ね合わせることにより,出芽酵母Atg7とKmAtg10との複合体,および,出芽酵母Atg7とAtAtg3との複合体の構造モデルを作製した(図2).これらのモデルにおいて,Atg7のホモ二量体のうちKmAtg10もしくはAtAtg3と結合している分子をAtg7cis,結合していない分子をAtg7transとよぶ.これらの構造モデルでは,それぞれのE2様酵素KmAtg10およびAtAtg3は,E1様酵素Atg7のN末端ドメインとC末端ドメインとのあいだの空間に位置しており,KmAtg10の活性化システイン残基Cys116あるいはAtAtg3の活性化システイン残基Cys258はAtg7transの活性化システイン残基Cys507transと向かい合うよう配置していた.KmAtg10あるいはAtAtg3の活性化システイン残基とAtg7cisの活性化システイン残基Cys507cisとのあいだの距離は50Åあるいは58Åであるのに対し,Atg7transの活性化システイン残基とのとのあいだの距離は17Åあるいは24Åとはるかに近いものであった.Atg7の活性化システイン残基はクロスオーバーループの中央に位置していたが,Atg8との結合によりこのクロスオーバーループはダイナミックな構造変化を起こすことが報告されているため,Atg8と結合しているAtg7のC末端ドメインの結晶構造を出芽酵母Atg7-KmAtg10複合体および出芽酵母Atg7-AtAtg3複合体の構造モデルと重ね合わせた6,8).その結果,Atg8との結合によりクロスオーバーループは移動し,Atg7transの活性化システイン残基はKmAtg10あるいはAtAtg3の活性化システイン残基から13Åあるいは21Åとより接近することが示唆された.一方,Atg7のクロスオーバーループが大きくコンフォメーション変化を起こしても,KmAtg10あるいはAtAtg3の活性化システイン残基がAtg7cisの活性化システイン残基に近づくのはむずかしいことが示唆された.
出芽酵母Atg7-AtAtg3複合体の構造モデルは,Atg8結合系においてE1様酵素Atg7はトランス機構によりユビキチン様タンパク質Atg8をE2様酵素Atg3へと受け渡すことを支持した.出芽酵母Atg7-KmAtg10複合体の構造モデルもまた,Atg12結合系においてAtg7はトランス機構によりユビキチン様タンパク質Atg12をE2様酵素Atg10へと受け渡すことを支持していた.この仮説を実証するため,トランス機構でしかAtg12をAtg10へと受け渡せないKmAtg7ヘテロ二量体KmAtg7transと,シス機構でしかAtg12をAtg10へと受け渡せないKmAtg7ヘテロ二量体KmAtg7cisを作製し,KmAtg10-AtAtg12bチオエステル中間体の形成反応について調べた.その結果,KmAtg7transを用いた場合のみKmAtg10-AtAtg12bチオエステル中間体が形成されたことから,Atg12結合系のトランスチオエステル化反応もAtg8結合系と同様にトランス機構により進行することが明確に示された.
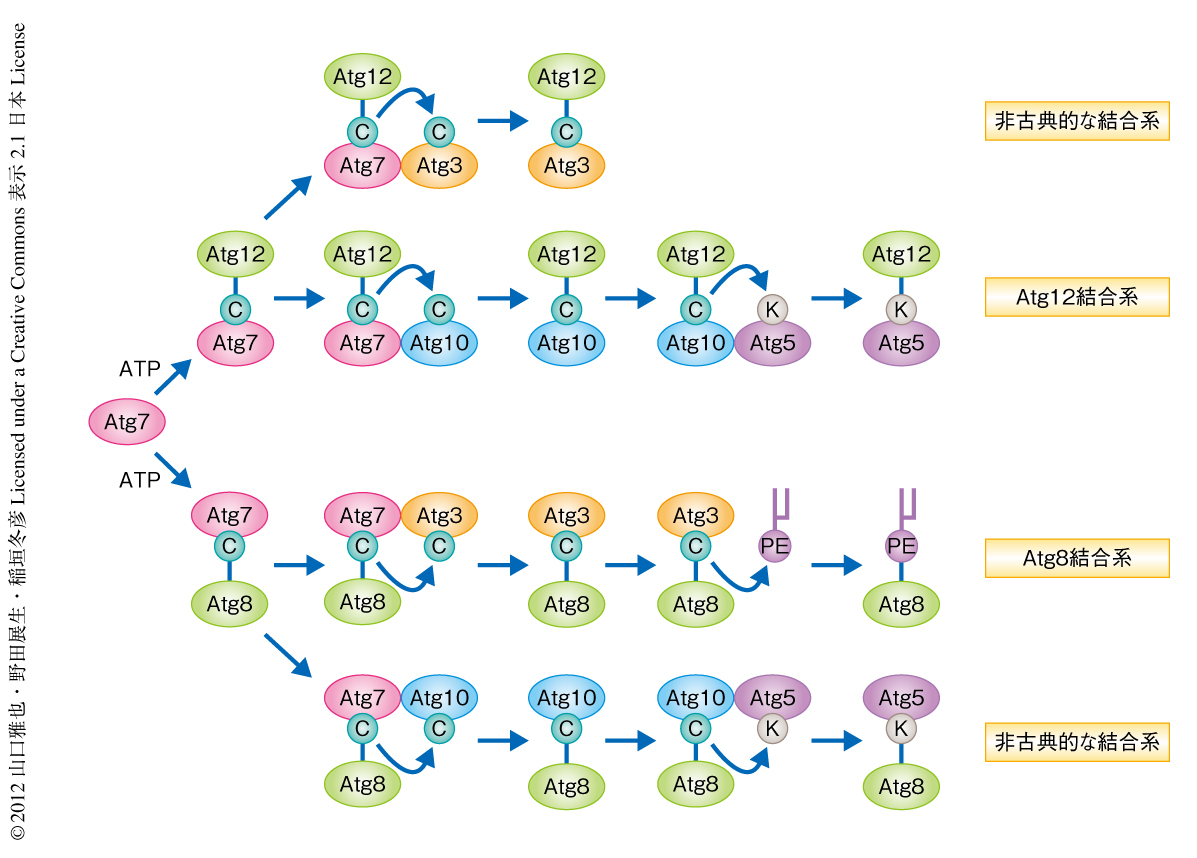
構造解析と生化学的な実験の結果は,E1様酵素Atg7はE2様酵素Atg10とAtg3とを同様の様式により認識することを示唆した.このため,Atg7はどのようにしてAtg10とAtg3に正しいペアのユビキチン様タンパク質であるAtg12とAtg8をそれぞれ受け渡すのだろうかという疑問が提示された.そこで,in vitroにおけるAtg12結合系の再構成系とAtg8結合系の再構成系を用いて,Atg7がどのくらい正しくユビキチン様タンパク質を受け渡しているのかを検討した.まず,Atg12結合系の再構成系において,KmAtg3の存在のもとKmAtg10-AtAtg12bチオエステル中間体の形成反応について調べた.その結果,予想に反して,KmAtg3-AtAtg12bチオエステル中間体に相当するバンドがKmAtg3の濃度に依存的に検出された.逆に,KmAtg10-AtAtg12bチオエステル中間体はKmAtg3の濃度に依存的に減少し,10μMのKmAtg10に対し5μMのKmAtg3の存在のもとでは確認できなくなった.この新たに検出されたバンドはジチオトレイトールの添加によるチオエステル結合の還元,もしくは,KmAtg3のCys235のAla置換変異体を用いた場合に消失したことから,KmAtg3-AtAtg12bチオエステル中間体であることが強く示唆された.すなわち,in vitroにおいて,E1様酵素KmAtg7はE2様酵素としてKmAtg10よりもKmAtg3に効率的にユビキチン様タンパク質AtAtg12bを受け渡すことが示された(図3).哺乳細胞においては,Atg3-Atg12チオエステル中間体が形成されたのち,同一のAtg3分子のLys側鎖にAtg12が自動的に受け渡されAtg12-Atg3結合体が形成されること,また,このAtg12-Atg3結合体はミトコンドリアの恒常性の維持に貢献していることが報告されている.Atg12-Atg3結合体は酵母においては報告されていないが,このことは,ユビキチン様タンパク質Atg12とE2様酵素Atg3とがチオエステル結合されうることを支持している.
Atg8結合系の再構成系において,KmAtg10の存在のもとKmAtg3-KmAtg8チオエステル中間体の形成反応についても調べた.その結果,さきほどと同様に,KmAtg10-KmAtg8チオエステル中間体に相当する新たなバンドがKmAtg10の濃度に依存的に検出された.このKmAtg3-KmAtg8チオエステル中間体は1μMのKmAtg3に対し50μMのKmAtg10が存在したとき顕著に低下した.この新たに検出されたバンドはジチオトレイトールの添加によるチオエステル結合の還元,もしくは,KmAtg10のCys116のAla置換変異体を用いた場合に消失したことから,KmAtg10-KmAtg8チオエステル中間体であることが示唆された.すなわち,in vitroにおいて,E1様酵素KmAtg7はE2様酵素としてKmAtg10よりもKmAtg3に対しより効率的にユビキチン様タンパク質KmAtg8を受け渡すことが示された(図3).以上の結果から,これまで受け入れられてきた考えに反して,E1様酵素Atg7はin vitroにおいてはE2様酵素としてAtg10とAtg3とを区別しておらず,Atg7に結合しているユビキチン様タンパク質をE2様酵素へとランダムに受け渡すことが示唆された.Atg7がAtg10よりもAtg3へ好んでユビキチン様タンパク質を受け渡すのは,Atg7とAtg3とのあいだの高い親和性に起因すると考えられた.
E2様酵素Atg10は特定のユビキチン様タンパク質リガーゼを必要とせず直接にAtg5を認識してユビキチン様タンパク質Atg12をAtg5へと受け渡し,共有結合によりAtg12-Atg5結合体を形成する.そこで,Atg10とチオエステル結合を形成したユビキチン様タンパク質Atg8はAtg5へと受け渡されるのかどうか検討した.KmAtg10-KmAtg8チオエステル中間体の形成ののちHis6標識したKmAtg5をくわえたところ,約45 kDaに新たなバンドが検出された.この新たなバンドは抗His抗体と抗Atg8抗体の両方により検出されたこと,ジチオトレイトールの添加による還元に耐性をもっていたこと,KmAtg5のLys145のAla置換変異体を用いた場合に消失したことから,KmAtg8-KmAtg5結合体の形成されていることが示唆された(図3).
Atg7-Atg10複合体およびAtg7-Atg3複合体の結晶構造においてみられたE1様酵素-E2様酵素相互作用は,ほかのユビキチン様タンパク質結合系におけるE1様酵素-E2様酵素相互作用とはまったく異なるものであることが明らかになった.これまで,ユビキチン様タンパク質Atg8のE2様酵素Atg3への受け渡しと,ユビキチン様タンパク質Atg12のE2様酵素Atg10への受け渡しは,E1様酵素Atg7により厳密に制御されているものと考えられてきた.しかし,この研究により,少なくともin vitroにおいては,Atg7はE2様酵素としてAtg3とAtg10とを区別せずにユビキチン様タンパク質Atg8もしくはAtg12へと受け渡すことが示唆された.今後は,in vivoにおいて,E1様酵素Atg7はどのようにして正しいE2様酵素とユビキチン様タンパク質のペアを選んでトランスチオエステル化反応を行っているのか明らかにしていくことが課題である.
略歴:2011年 北海道大学大学院生命科学院博士課程 修了,同年 同 博士研究員を経て,2012年より米国St. Jude Children’s Research Hospital博士研究員.
研究テーマ:ユビキチン様修飾系の分子機構.
関心事:アメリカの文化と人.
野田 展生(Nobuo N. Noda)
微生物化学研究会微生物化学研究所 主席研究員.
稲垣 冬彦(Fuyuhiko Inagaki)
北海道大学大学院先端生命科学研究院 特任教授.
© 2012 山口雅也・野田展生・稲垣冬彦 Licensed under CC 表示 2.1 日本
(1北海道大学大学院先端生命科学研究院 次世代ポストゲノム研究センター,2微生物化学研究会微生物化学研究所 分子構造解析部)
email:山口雅也,野田展生,稲垣冬彦
DOI: 10.7875/first.author.2012.148
Noncanonical recognition and UBL loading of distinct E2s by autophagy-essential Atg7.
Masaya Yamaguchi, Kazuaki Matoba, Ryoko Sawada, Yuko Fujioka, Hitoshi Nakatogawa, Hayashi Yamamoto, Yoshihiro Kobashigawa, Hisashi Hoshida, Rinji Akada, Yoshinori Ohsumi, Nobuo N Noda, Fuyuhiko Inagaki
Nature Structural & Molecular Biology, 19, 1250-1256 (2012)
要 約
オートファジーにはAtg8結合系とAtg12結合系の2つのユビキチン様タンパク質結合系が必須である.この2つの結合系では,オートファジーにおける唯一のユビキチン様タンパク質活性化酵素であるAtg7が共通して機能する.Atg7はAtg8結合系におけるAtg3とAtg12結合系におけるAtg10の,2つのユビキチン様タンパク質結合酵素を認識しなければならない.しかし,実際にAtg7がいかにしてこの2つのユビキチン様タンパク質結合酵素に正しい組合せのユビキチン様タンパク質を受け渡すのかについては明らかにされていなかった.今回,筆者らは,Atg7のN末端ドメインとAtg10との複合体,および,Atg7のN末端ドメインとAtg3との複合体の結晶構造を決定した.これらの構造をAtg7ホモ二量体の結晶構造と重ね合わせることにより,ユビキチン様タンパク質活性化酵素Atg7はユビキチン様タンパク質Atg8とAtg12をユビキチン様タンパク質結合酵素Atg3とAtg10に,トランス機構により受け渡す可能性を見い出し,これをin vitroにおける実験により確認した.さらに,in vitroにおいてAtg7は,Atg8とAtg12をAtg3とAtg10にランダムに受け渡すことが示された.
はじめに
オートファジーは真核生物に広く保存された細胞内タンパク質分解経路である.オートファジーでは飢餓に応答して細胞質に忽然と出現した隔離膜がタンパク質,細胞質成分,オルガネラ,細胞に侵入した細菌などさまざまな対象をとりかこみ,二重膜からなる構造体であるオートファゴソームを形成する.オートファゴソームは最終的に,酵母や植物では液胞と,哺乳細胞ではリソソームと,外膜において膜融合し,内容物を内膜ごと液胞あるいはリソソームへと放出し分解をうける1).このオートファゴソームの形成過程にはAtg8結合系とAtg12結合系の2つのユビキチン様タンパク質結合系が必要である2,3).オートファジーに特異的なユビキチン様タンパク質活性化酵素(E1様酵素)であるAtg7はATPに依存的にユビキチン様タンパク質であるAtg8とAtg12を,オートファジーに特異的なユビキチン様タンパク質結合酵素(E2様酵素)であるAtg3とAtg10にそれぞれ受け渡す.Atg8とAtg12は最終的にホスファチジルエタノールアミンおよびAtg5とそれぞれ結合体を形成し,オートファジーにおいて重要な役割をはたす4,5)(出芽酵母におけるオートファジー研究については,荒木保弘・大隅良典, 領域融合レビュー, 1, e005, 2012 も参照されたい).
2011年,筆者らを含む複数のグループにより,出芽酵母Atg7全長の結晶構造,Atg7とAtg8との複合体の結晶構造,Atg7とAtg3フレキシブル領域との複合体の結晶構造が報告された6-8).Atg7はおもにN末端ドメインとC末端ドメインの2つのドメインから構成されており,Atg7はN末端ドメインを用いてAtg3を,C末端ドメインを用いてAtg8を認識することが明らかになった.また,Atg7はC末端ドメインにおいてホモ二量体を形成することから,2つのユビキチン様タンパク質および2つのE2様酵素と相互作用することができると考えられている.in vitroにおける生化学的な実験から,Atg8結合系において,Atg7はホモ二量体における同一の分子においてAtg8をAtg3へ受け渡すシス機構ではなく,Atg8をホモ二量体においてもうひとつの別の分子と結合したAtg3へ受け渡すトランス機構により進行することが示された.しかしながら,Atg7がどのようにAtg3を認識してAtg8を受け渡すのかについては明らかになっておらず,さらに,Atg12結合系におけるAtg7の役割についても不明な点が多い.
筆者らは,Atg7のN末端ドメインとAtg10との複合体,および,Atg7のN末端ドメインとAtg3との複合体の結晶構造を決定し,構造情報にもとづき種々の生化学的な実験を行った結果,Atg7とAtg10との相互作用およびAtg7とAtg3との相互作用は既知のE1様酵素とE2様酵素との相互作用とはまったく異なるものであること,in vitroにおいて,E1様酵素Atg7はE2様酵素Atg3とAtg10とを厳密に区別せずにユビキチン様タンパク質Atg8とAtg12を受け渡すことを明らかにした.
1.Atg7のN末端ドメインとAtg10との複合体の結晶構造
E1様酵素Atg7のN末端ドメインはAtg8結合系におけるAtg8のAtg7からAtg3への受け渡し,および,Atg12結合系におけるAtg12のAtg7からAtg10への受け渡しに必要であるかどうか,耐熱性酵母Kluyveromyces marxianusに由来するKmAtg7,KmAtg10,KmAtg5,および,シロイヌナズナに由来するAtAtg12bを利用したin vitroにおけるAtg12結合系の再構成系により検討した9).その結果,KmAtg7のC末端ドメインはAtAtg12bの活性化に十分であること,KmAtg7のN末端ドメインはAtAtg12bのKmAtg10へのトランス機構によるチオエステル化反応に必要であることが示唆された.このことから,E1様酵素Atg7のN末端ドメインとC末端ドメインの役割はAtg8結合系とAtg12結合系とで共通であることが示された.
KmAtg7のN末端ドメインとKmAtg10全長との複合体の結晶構造を分解能3.2Åで決定した(図1a,PDB ID:3VX7).KmAtg7のN末端ドメインはすでに構造の決定されている出芽酵母Atg7のN末端ドメインとほとんど同一の構造を保持しており,15本のβストランドと6本のαヘリックスから構成されていた.KmAtg10はKmAtg7 N末端ドメインのC末端側の領域と結合しておりL字型の構造を形成していた.おもな相互作用はKmAtg7 N末端ドメインのβ15とKmAtg10のβ4’とのあいだの分子間βシートであった.E1様酵素とE2様酵素との古典的な相互作用では,E1様酵素のユビキチン様ドメインとE2様酵素のN末端側にあるαヘリックスおよびβ1とβ2とのあいだのループ領域とが相互作用する.したがって,KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造でみられたE1様酵素-E2様酵素相互作用は,古典的なE1様酵素-E2様酵素相互作用とはまったく異なることがわかった.
KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造にもとづき11種のKmAtg7変異体と7種のKmAtg10変異体を作製し,KmAtg7からKmAtg10へのAtAtg12bのトランス機構によるチオエステル化反応への影響を評価した.その結果,野生型を用いた場合と比較して,ほぼすべての変異体においてトランスチオエステル化反応の効率は低下した.とくに,KmAtg7のβ15に変異を導入した場合にトランスチオエステル化反応の効率は大きく減弱した.以上の結果より,KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造においてみられた相互作用は,実際にAtg12結合系におけるE1様酵素KmAtg7からE2様酵素KmAtg10へのユビキチン様タンパク質AtAtg12bの受け渡しに重要であることが示された.
2.Atg7のN末端ドメインとAtg3との複合体の結晶構造
E1様酵素Atg7はAtg10にくわえもうひとつのE2様酵素であるAtg3を認識する.Atg10とAtg3とのあいだの配列相同性は低いことから,Atg7のN末端ドメインはどのようにして2つのE2様酵素を認識するのかは不明であった.これまでに,Atg7のN末端ドメインとAtg3のフレキシブル領域の一部のペプチドとの複合体の結晶構造が報告されていたが,この相互作用のみではトランスチオエステル化反応におけるAtg7とAtg3の活性化システイン残基どうしの適切な配向は決まらないと考えられた7).
そこで,Atg8結合系におけるトランスチオエステル化反応の際のAtg3とAtg7との直接の相互作用を知るため,AtAtg7のN末端ドメインとAtAtg3との複合体の結晶構造を分解能3.1Åで決定した(図1b,PDB ID:3VX8).予測に反して,出芽酵母Atg7のN末端ドメインと出芽酵母Atg3のフレキシブル領域との複合体の結晶構造においてみられた相互作用はみられなかった.しかしながら,AtAtg7のN末端ドメインにおいて表面の別の領域になんらかの電子密度が確認され,また,in vitroにおけるプルダウンアッセイによりAtAtg3のフレキシブル領域はAtAtg7とAtAtg3との相互作用に必要であることが確認された.このことから,AtAtg3もAtAtg7のN末端ドメインと直接的な相互作用を形成すると考えられた.
興味深いことに,AtAtg3のコア領域はAtAtg7のN末端ドメインのC末端側の領域と結合しており,KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造と同様にL字型の構造を形成していた.AtAtg7のN末端ドメインとAtAtg3のコア領域とのおもな相互作用もKmAtg7 N末端ドメイン-KmAtg10相互作用と類似しており,AtAtg7のN末端ドメインのβ15とAtAtg3のβ4のうしろのループ(β4ループ)とが相互作用していた.しかし,この相互作用はKmAtg7 N末端ドメイン-KmAtg10複合体でみられた分子間βシートではなく,側鎖を介した疎水性相互作用からなっていた.この相互作用にくわえ,AtAtg7のN末端ドメインのβ1およびβ3の手前のループとAtAtg3のAsn77およびArg219とのあいだにも相互作用がみられた.
これらの相互作用の重要性を確認するため,AtAtg7のN末端ドメインのβ15とAtAtg3のβ4ループの三点変異体とAtAtg3のAsn77点変異体を作製した.in vitroにおけるプルダウンアッセイの結果,これらの変異体を用いた場合にはAtAtg7とAtAtg3とのあいだの結合が弱くなっていることが確認された.さらに,AtAtg7からAtAtg3へのAtAtg8aのトランス機構によるチオエステル化反応への影響を検討した結果,すべての変異体において反応の効率は低下していた.以上の結果から,AtAtg7 N末端ドメイン-AtAtg3複合体の結晶構造でみられた相互作用は,Atg8結合系におけるE1様酵素AtAtg7とE1様酵素AtAtg3との結合,ならびに,AtAtg7からAtAtg3へのユビキチン様タンパク質AtAtg8aの受け渡しにおいて重要であることが示された.
3.Atg7のトランス機構によるチオエステル化反応の構造基盤
KmAtg7 N末端ドメイン-KmAtg10複合体の結晶構造とAtAtg7 N末端ドメイン-AtAtg3複合体の結晶構造を出芽酵母Atg7全長の結晶構造と重ね合わせることにより,出芽酵母Atg7とKmAtg10との複合体,および,出芽酵母Atg7とAtAtg3との複合体の構造モデルを作製した(図2).これらのモデルにおいて,Atg7のホモ二量体のうちKmAtg10もしくはAtAtg3と結合している分子をAtg7cis,結合していない分子をAtg7transとよぶ.これらの構造モデルでは,それぞれのE2様酵素KmAtg10およびAtAtg3は,E1様酵素Atg7のN末端ドメインとC末端ドメインとのあいだの空間に位置しており,KmAtg10の活性化システイン残基Cys116あるいはAtAtg3の活性化システイン残基Cys258はAtg7transの活性化システイン残基Cys507transと向かい合うよう配置していた.KmAtg10あるいはAtAtg3の活性化システイン残基とAtg7cisの活性化システイン残基Cys507cisとのあいだの距離は50Åあるいは58Åであるのに対し,Atg7transの活性化システイン残基とのとのあいだの距離は17Åあるいは24Åとはるかに近いものであった.Atg7の活性化システイン残基はクロスオーバーループの中央に位置していたが,Atg8との結合によりこのクロスオーバーループはダイナミックな構造変化を起こすことが報告されているため,Atg8と結合しているAtg7のC末端ドメインの結晶構造を出芽酵母Atg7-KmAtg10複合体および出芽酵母Atg7-AtAtg3複合体の構造モデルと重ね合わせた6,8).その結果,Atg8との結合によりクロスオーバーループは移動し,Atg7transの活性化システイン残基はKmAtg10あるいはAtAtg3の活性化システイン残基から13Åあるいは21Åとより接近することが示唆された.一方,Atg7のクロスオーバーループが大きくコンフォメーション変化を起こしても,KmAtg10あるいはAtAtg3の活性化システイン残基がAtg7cisの活性化システイン残基に近づくのはむずかしいことが示唆された.
出芽酵母Atg7-AtAtg3複合体の構造モデルは,Atg8結合系においてE1様酵素Atg7はトランス機構によりユビキチン様タンパク質Atg8をE2様酵素Atg3へと受け渡すことを支持した.出芽酵母Atg7-KmAtg10複合体の構造モデルもまた,Atg12結合系においてAtg7はトランス機構によりユビキチン様タンパク質Atg12をE2様酵素Atg10へと受け渡すことを支持していた.この仮説を実証するため,トランス機構でしかAtg12をAtg10へと受け渡せないKmAtg7ヘテロ二量体KmAtg7transと,シス機構でしかAtg12をAtg10へと受け渡せないKmAtg7ヘテロ二量体KmAtg7cisを作製し,KmAtg10-AtAtg12bチオエステル中間体の形成反応について調べた.その結果,KmAtg7transを用いた場合のみKmAtg10-AtAtg12bチオエステル中間体が形成されたことから,Atg12結合系のトランスチオエステル化反応もAtg8結合系と同様にトランス機構により進行することが明確に示された.
4.Atg7によるAtg10とAtg3への非選択的なユビキチン様タンパク質の受け渡し
構造解析と生化学的な実験の結果は,E1様酵素Atg7はE2様酵素Atg10とAtg3とを同様の様式により認識することを示唆した.このため,Atg7はどのようにしてAtg10とAtg3に正しいペアのユビキチン様タンパク質であるAtg12とAtg8をそれぞれ受け渡すのだろうかという疑問が提示された.そこで,in vitroにおけるAtg12結合系の再構成系とAtg8結合系の再構成系を用いて,Atg7がどのくらい正しくユビキチン様タンパク質を受け渡しているのかを検討した.まず,Atg12結合系の再構成系において,KmAtg3の存在のもとKmAtg10-AtAtg12bチオエステル中間体の形成反応について調べた.その結果,予想に反して,KmAtg3-AtAtg12bチオエステル中間体に相当するバンドがKmAtg3の濃度に依存的に検出された.逆に,KmAtg10-AtAtg12bチオエステル中間体はKmAtg3の濃度に依存的に減少し,10μMのKmAtg10に対し5μMのKmAtg3の存在のもとでは確認できなくなった.この新たに検出されたバンドはジチオトレイトールの添加によるチオエステル結合の還元,もしくは,KmAtg3のCys235のAla置換変異体を用いた場合に消失したことから,KmAtg3-AtAtg12bチオエステル中間体であることが強く示唆された.すなわち,in vitroにおいて,E1様酵素KmAtg7はE2様酵素としてKmAtg10よりもKmAtg3に効率的にユビキチン様タンパク質AtAtg12bを受け渡すことが示された(図3).哺乳細胞においては,Atg3-Atg12チオエステル中間体が形成されたのち,同一のAtg3分子のLys側鎖にAtg12が自動的に受け渡されAtg12-Atg3結合体が形成されること,また,このAtg12-Atg3結合体はミトコンドリアの恒常性の維持に貢献していることが報告されている.Atg12-Atg3結合体は酵母においては報告されていないが,このことは,ユビキチン様タンパク質Atg12とE2様酵素Atg3とがチオエステル結合されうることを支持している.
Atg8結合系の再構成系において,KmAtg10の存在のもとKmAtg3-KmAtg8チオエステル中間体の形成反応についても調べた.その結果,さきほどと同様に,KmAtg10-KmAtg8チオエステル中間体に相当する新たなバンドがKmAtg10の濃度に依存的に検出された.このKmAtg3-KmAtg8チオエステル中間体は1μMのKmAtg3に対し50μMのKmAtg10が存在したとき顕著に低下した.この新たに検出されたバンドはジチオトレイトールの添加によるチオエステル結合の還元,もしくは,KmAtg10のCys116のAla置換変異体を用いた場合に消失したことから,KmAtg10-KmAtg8チオエステル中間体であることが示唆された.すなわち,in vitroにおいて,E1様酵素KmAtg7はE2様酵素としてKmAtg10よりもKmAtg3に対しより効率的にユビキチン様タンパク質KmAtg8を受け渡すことが示された(図3).以上の結果から,これまで受け入れられてきた考えに反して,E1様酵素Atg7はin vitroにおいてはE2様酵素としてAtg10とAtg3とを区別しておらず,Atg7に結合しているユビキチン様タンパク質をE2様酵素へとランダムに受け渡すことが示唆された.Atg7がAtg10よりもAtg3へ好んでユビキチン様タンパク質を受け渡すのは,Atg7とAtg3とのあいだの高い親和性に起因すると考えられた.
E2様酵素Atg10は特定のユビキチン様タンパク質リガーゼを必要とせず直接にAtg5を認識してユビキチン様タンパク質Atg12をAtg5へと受け渡し,共有結合によりAtg12-Atg5結合体を形成する.そこで,Atg10とチオエステル結合を形成したユビキチン様タンパク質Atg8はAtg5へと受け渡されるのかどうか検討した.KmAtg10-KmAtg8チオエステル中間体の形成ののちHis6標識したKmAtg5をくわえたところ,約45 kDaに新たなバンドが検出された.この新たなバンドは抗His抗体と抗Atg8抗体の両方により検出されたこと,ジチオトレイトールの添加による還元に耐性をもっていたこと,KmAtg5のLys145のAla置換変異体を用いた場合に消失したことから,KmAtg8-KmAtg5結合体の形成されていることが示唆された(図3).
おわりに
Atg7-Atg10複合体およびAtg7-Atg3複合体の結晶構造においてみられたE1様酵素-E2様酵素相互作用は,ほかのユビキチン様タンパク質結合系におけるE1様酵素-E2様酵素相互作用とはまったく異なるものであることが明らかになった.これまで,ユビキチン様タンパク質Atg8のE2様酵素Atg3への受け渡しと,ユビキチン様タンパク質Atg12のE2様酵素Atg10への受け渡しは,E1様酵素Atg7により厳密に制御されているものと考えられてきた.しかし,この研究により,少なくともin vitroにおいては,Atg7はE2様酵素としてAtg3とAtg10とを区別せずにユビキチン様タンパク質Atg8もしくはAtg12へと受け渡すことが示唆された.今後は,in vivoにおいて,E1様酵素Atg7はどのようにして正しいE2様酵素とユビキチン様タンパク質のペアを選んでトランスチオエステル化反応を行っているのか明らかにしていくことが課題である.
文 献
- Mizushima, N., Yoshimori, T. & Ohsumi, Y.: The role of Atg proteins in autophagosome formation. Annu. Rev. Cell Dev. Biol., 27, 107-132 (2011)[PubMed]
- Nakatogawa, H., Suzuki, K., Kamada, Y. et al.: Dynamics and diversity in autophagy mechanisms: lessons from yeast. Nat. Rev. Mol. Cell Biol., 10, 458-467 (2009)[PubMed]
- Noda, N. N., Ohsumi, Y. & Inagaki, F.: ATG systems from the protein structural point of view. Chem. Rev., 109, 1587-1598 (2009)[PubMed]
- Ichimura, Y., Kirisako, T., Takao, T. et al.: A ubiquitin like system mediates protein lipidation. Nature, 408, 488-192 (2000)[PubMed]
- Mizushima, N., Noda, T., Yoshimori, T. et al.: A protein conjugation system essential for autophagy. Nature, 395, 395-398 (1998)[PubMed]
- Noda, N. N., Satoo, K., Fujioka, Y. et al.: Structural basis of Atg8 activation by a homodimeric E1, Atg7. Mol. Cell, 44, 462-475 (2011)[PubMed] [新着論文レビュー]
- Taherbhoy, A. M., Tait, S. W., Kaiser, S. E. et al.: Atg8 transfer from Atg7 to Atg3: a distinctive E1-E2 architecture and mechanism in the autophagy pathway. Mol. Cell, 44, 451-461 (2011)[PubMed]
- Hong, S. B., Kim, B. W., Lee, K. E. et al.: Insights into noncanonical E1 enzyme activation from the structure of autophagic E1 Atg7 with Atg8. Nat. Struct. Mol. Biol., 18, 1323-1330 (2011)[PubMed]
- Yamaguchi, M., Noda, N. N., Yamamoto, H. et al.: Structural insights into atg10-mediated formation of the autophagy essential Atg12-Atg5 conjugate. Structure, 20, 1244-1254 (2012)[PubMed]
著者プロフィール
略歴:2011年 北海道大学大学院生命科学院博士課程 修了,同年 同 博士研究員を経て,2012年より米国St. Jude Children’s Research Hospital博士研究員.
研究テーマ:ユビキチン様修飾系の分子機構.
関心事:アメリカの文化と人.
野田 展生(Nobuo N. Noda)
微生物化学研究会微生物化学研究所 主席研究員.
稲垣 冬彦(Fuyuhiko Inagaki)
北海道大学大学院先端生命科学研究院 特任教授.
© 2012 山口雅也・野田展生・稲垣冬彦 Licensed under CC 表示 2.1 日本