出芽酵母のDicerによるsiRNA二本鎖の新しい生成機構
中西 孝太郎
(米国Memorial Sloan-Kettering Cancer Center,Structural Biology Program)
email:中西孝太郎
DOI: 10.7875/first.author.2011.124
The inside-out mechanism of dicers from budding yeasts.
David E. Weinberg, Kotaro Nakanishi, Dinshaw J. Patel, David P. Bartel
Cell, 146, 262-276 (2011)
モデル生物Saccharomyces cerevisiaeに代表される出芽酵母にはRNAi現象はないものと考えられてきた.しかし,最近,S. cerevisiae以外の数種の出芽酵母にはRNAi現象のあることが明らかになった.そこでの大きなブレイクスルーは,まったく新しいDicerの発見であった.従来から知られているヒトやショウジョウバエのもつDicerでは,二本鎖RNAの末端を認識するPAZドメインと,その反対側でRNAを切断するRNase IIIドメインとの距離が分子的な定規としてはたらくことで,生成されるsiRNA二本鎖の長さを決定している.一方,出芽酵母のもつDicerはPAZドメインをもたないにもかかわらず,特定の長さのsiRNA二本鎖を生成することができる.しかし,その機構は明らかにされていなかった.今回,筆者らは,X線結晶構造解析と生化学的な手法とを用い,出芽酵母のもつDicerが長い二本鎖RNAのうえに協調的に結合し活性部位が23塩基長の間隔で並ぶことにより二本鎖RNAが特定の長さに切断されるという,まったく新しいsiRNA二本鎖の生成機構を明らかにした.
RNAi(RNA interference,RNA干渉)はRNA鎖に相補的なmRNAが分解されるという真核生物に特有の転写後遺伝子抑制現象である1).その中心としてはたらくタンパク質Dicerは長い二本鎖RNAからはsiRNA二本鎖(siRNA:small interfering RNA)を生成し,また,ステムループ構造をとるmiRNA前駆体からはmiRNA二本鎖(miRNA:microRNA)を生成する.別の重要なタンパク質Argonauteはそれら二本鎖RNAと結合したのち,一方のRNA鎖を切断し,残った一本鎖RNAをガイド鎖として標的となるmRNAに結合する2).
このように,RNAiにおいてはDicerとArgonauteの両方のタンパク質を必要とするが,モデル生物Saccharomyces cerevisiaeではゲノムにそれらをコードする遺伝子がみつからない.また,S. cerevisiae以外の数種の出芽酵母,Saccharomyces castellii,Kluyveromyces polysporus,Candida albicansでは,Argonauteの遺伝子は同定されているがDicerの遺伝子がみつかっていない.そのため,出芽酵母はRNAi現象のない例外的な真核生物であると考えられてきた.しかしながら最近,Argonauteをもつこれら3種の出芽酵母にはRNAi現象のあることが明らかになった3).ブレイクスルーは新しいDicerの発見であった.
これまで,出芽酵母においてDicerが発見されてこなかったことには理由がある.Dicerのドメイン構造は生物種のあいだで異なるが,2塩基の突出した3’側の末端を認識するPAZドメインと2つの連続したRNase IIIドメインをもつことがDicerのアイデンティティとされてきた.実際,このドメインセットのみで構成されるランブル鞭毛虫(Giardia intestinalis)に由来するDicerは25~27塩基長の二本鎖RNAを生成できる4).また,ランブル鞭毛虫に由来するDicerの結晶構造は,PAZドメインがRNA鎖の末端を固定し,そこからRNase IIIドメインの活性部位までの長さに相当するsiRNA二本鎖が生成されるというモデルをよく説明した4,5).したがって,ゲノムにおいてDicerの遺伝子を探す際には,1つのPAZドメインと2つの連続したRNase IIIドメインをもつ遺伝子がその候補となった.しかし,そのようなドメインセットをもつ遺伝子は出芽酵母のゲノムには存在しなかったのである.そこで,Dicerの遺伝子を探す際の指標を“少なくとも1つのRNase IIIドメインをもつこと”に緩和することが,出芽酵母に特有なDicer(Dcr1)の発見につながった3).このDcr1は長い二本鎖RNAから特定の塩基長のsiRNA二本鎖を生成できるが,従来のDicerのもつドメイン構造とは大きく異なり,PAZドメインをもたずRNase IIIドメインも1つしかない.出芽酵母のもつDcr1がどのような機構でRNAの長さを測定しているかについてはまったくの謎であった.
出芽酵母のもつDcr1は,N末端ドメイン,RNase IIIドメイン,そして,2つの二本鎖RNA結合ドメイン(dsRBD1,dsRBD2)から構成される.当初,K. polysporusに由来するDcr1の全長構造の決定を試みたが,核酸が強固に結合しており全長を高純度に精製することができなかった.そこで,核酸への結合能が低下しつつもDicing活性(特定の塩基長のsiRNA二本鎖を生成する活性)を維持するコンストラクトを模索した.その結果,N末端ドメインからdsRBD1までの領域を含むフラグメント(Dcr1ΔC)が全長構造と同じ程度のDicing活性をもつことがわかった4).出芽酵母のもつDcr1に特徴的なC末端側のdsRBD2こそが特定の長さのsiRNA二本鎖を生成するために必須であろうと考えていたので,この結果は予想外であった.全長構造に比べるとその量は少ないが,Dcr1ΔCには依然として大量の核酸が強固に結合していた.筆者が所属する研究室ではRNaseの使用が全面的に禁止されているため,最終的に5種類のカラムを用いることにより夾雑する核酸を完全に除くことに成功し,Dcr1ΔCの結晶構造を2.3Åの分解能で決定した(図1,PDB ID:3RV0).
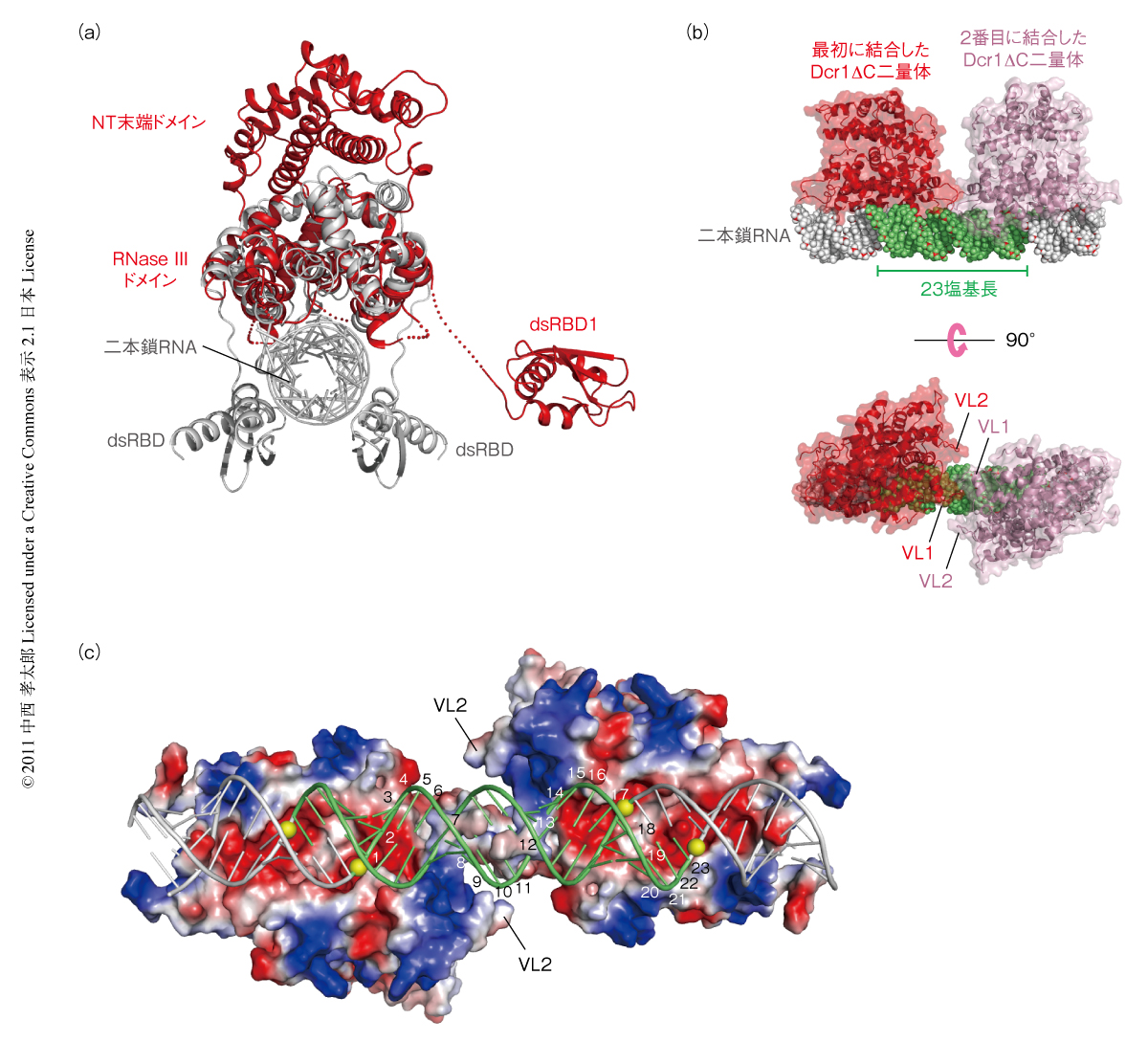
Dcr1ΔCの立体構造は,N末端ドメインとRNase IIIドメインがそれぞれ二量体化したコアパートと,溶液において自由に動くと思われるdsRBD1から構成されていた(今回の結晶構造では,片方のdsRBD1のみ電子密度が明瞭であった).二量体化したRNase IIIドメインから両側にdsRBD1の伸びるようすは,細菌のRNase IIIの立体構造と類似していた6)(図2a).RNAプロセシングにかかわる細菌のRNase IIIは,2つのdsRBDにより二本鎖RNAを抱き込んだのち,RNase IIIドメインにある2カ所の活性部位が二本鎖RNAをそれぞれ同時に切断する.今回のDcr1ΔCの立体構造からN末端ドメインがRNase IIIドメインの活性部位の反対側で二量体化することがわかり,Dcr1ΔCは細菌のRNase IIIと同様に二本鎖RNAを結合することが示唆された(図2a).
従来のDicerは,PAZドメインが二本鎖RNAの3’末端を認識しRNase IIIドメインがその反対側を切断することでsiRNA二本鎖を生成する4,5).その際,PAZドメインとRNase IIIドメインとの間隔がsiRNA二本鎖の長さを決定する.これに対し,Dcr1ΔCは二量体化することで二本鎖RNAと結合しこれを切断できるが,PAZドメインに代わり二本鎖RNAの末端を認識できるようなドメインがない.しかしながら,Dcr1ΔCのRNase IIIドメインから両側に突き出たループ,VL1とVL2が列車の連結器のように相補的な形状をもつことに気づいた(図2b).実際,Dcr1ΔC二量体を二本鎖RNAのうえで23塩基長の間隔をもって並べると,Dcr1ΔCどうしは立体障害なく連結することができた(図2 b,c).このことから,出芽酵母のもつDcr1によるsiRNA二本鎖の生成は,末端からではなく内部からはじまるのではないかと考えた.もし,Dcr1二量体が列車の車両のように二本鎖RNAのうえに並ぶことでsiRNA二本鎖を生成しているのであれば,野生型のDcr1ΔCに対し切断活性を失った変異型のDcr1ΔCを混ぜることにより,23塩基の倍数の長さのsiRNA二本鎖が生成されてくるに違いないと予想した.そして予想どおり,切断活性を失った変異型Dcr1ΔCの割合が増えるにつれ,46塩基長,69塩基長,92塩基長のsiRNA二本鎖が生成された.
さきに述べた機構によりsiRNA二本鎖が連続的に生成されるためには,最初に二本鎖RNAと結合したDcr1二量体がRNAを切断してこれをはなすまえに,つぎのDcr1二量体がそのとなりに結合する必要がある.70塩基長の二本鎖RNAを基質としDcr1ΔCが複数回の反応を触媒する多重代謝回転キネティクス解析を行ったところ,前定常状態におけるsiRNA二本鎖の生成速度はDcr1ΔCの濃度に比例して上昇するが,定常状態におけるsiRNA二本鎖の生成速度はDcr1ΔCの濃度によらず著しく遅かった.この結果から,1回目の切断反応ののちもDcr1ΔCは二本鎖RNAと結合したままでいるために,2回目の切断反応が起こらないことが示唆された.
in vitro切断実験においては基質として完全に相補的な500塩基対の二本鎖RNAを用いた.二本鎖RNAに対するDcr1の結合は配列に非特異的であるため,siRNA二本鎖の生成は二本鎖RNAの任意の箇所からはじまる.一方,生体においてDcr1の基質となるRNAは特定の箇所で切断されることがわかっている3).生体における基質RNAは完全に相補的な二本鎖RNAではなく,ミスマッチ塩基対やバルジなどを含み塩基配列に特異的な2次構造をもつであろう.それらが立体障害となりDcr1が結合できる場所を制限することで,生体では特定の箇所からsiRNA二本鎖の生成がはじまるものと考えている.
これまで,RNase IIIファミリーは分子量とドメイン構造により,クラス1:RNase III(細菌),クラスII:Drosha(真核生物),クラスIII:Dicer(真核生物)の3つのグループに分類されてきた7).RNase IIIファミリーのすべてに共通な特徴として,触媒活性に必須なMg2+の配位に2つのアスパラギン酸残基と2つのグルタミン酸残基が直接または水分子を介しかかわっていることがあげられる.今回,Dcr1ΔCの活性部位において明瞭な八面体の電子密度を確認することができた.結晶化の条件にMg2+を含むことから,この電子密度の中心にはMg2+があるものと考えられた.この活性部位では,さきの保存された4残基にくわえ,アスパラギン残基とリジン残基とがMg2+の配位にくわわっており,それらのアラニン残基への置換体ではいずれもDicing活性の低下が観察された.特筆すべきは,この新たにみつかった2残基はクラスI RNase IIIファミリーでは保存されておらず,クラスIIとクラスIIIのみで保存されていることであった.つまり,出芽酵母のもつDcr1はその立体構造こそ細菌のもつクラスIに似ているものの,触媒残基としては真核生物のもつクラスIIおよびクラスIIIと同じセットをもつハイブリッド酵素であった.この事実は,細菌のRNase IIIが出芽酵母Dcr1をへて真核生物のDroshaやDicerへと進化していった可能性を連想させた.
筆者らは,出芽酵母のもつDcr1が二本鎖RNAのうえでとなりあって結合することにより活性部位が一定の間隔で並び,特定の塩基長のsiRNA二本鎖を生成することを明らかにした.この機構は,二本鎖RNAの末端を必要とする従来のDicerによるsiRNA二本鎖の生成機構とはまったく異なることから,出芽酵母のもつDcr1と従来のDicerとは機能収斂のよい例といえよう.また,今回の研究では,RNAiという枠をこえ,分子定規の新しい概念をその実験的な証拠とともに提示できたのではないかと考えている.
略歴:2007年 東京工業大学大学院生命理工学研究科 修了,同年 同 博士研究員,2008年 米国Memorial Sloan-Kettering Cancer Centerポスドク研究員を経て,2011年より同 リサーチアソシエート.
研究テーマ:ノンコーディングRNAによる制御機構の解明.
抱負:立体構造からみえてくる新しい知見をもとに生命現象の謎を解き明かしたい.
© 2011 中西 孝太郎 Licensed under CC 表示 2.1 日本
(米国Memorial Sloan-Kettering Cancer Center,Structural Biology Program)
email:中西孝太郎
DOI: 10.7875/first.author.2011.124
The inside-out mechanism of dicers from budding yeasts.
David E. Weinberg, Kotaro Nakanishi, Dinshaw J. Patel, David P. Bartel
Cell, 146, 262-276 (2011)
要 約
モデル生物Saccharomyces cerevisiaeに代表される出芽酵母にはRNAi現象はないものと考えられてきた.しかし,最近,S. cerevisiae以外の数種の出芽酵母にはRNAi現象のあることが明らかになった.そこでの大きなブレイクスルーは,まったく新しいDicerの発見であった.従来から知られているヒトやショウジョウバエのもつDicerでは,二本鎖RNAの末端を認識するPAZドメインと,その反対側でRNAを切断するRNase IIIドメインとの距離が分子的な定規としてはたらくことで,生成されるsiRNA二本鎖の長さを決定している.一方,出芽酵母のもつDicerはPAZドメインをもたないにもかかわらず,特定の長さのsiRNA二本鎖を生成することができる.しかし,その機構は明らかにされていなかった.今回,筆者らは,X線結晶構造解析と生化学的な手法とを用い,出芽酵母のもつDicerが長い二本鎖RNAのうえに協調的に結合し活性部位が23塩基長の間隔で並ぶことにより二本鎖RNAが特定の長さに切断されるという,まったく新しいsiRNA二本鎖の生成機構を明らかにした.
はじめに
RNAi(RNA interference,RNA干渉)はRNA鎖に相補的なmRNAが分解されるという真核生物に特有の転写後遺伝子抑制現象である1).その中心としてはたらくタンパク質Dicerは長い二本鎖RNAからはsiRNA二本鎖(siRNA:small interfering RNA)を生成し,また,ステムループ構造をとるmiRNA前駆体からはmiRNA二本鎖(miRNA:microRNA)を生成する.別の重要なタンパク質Argonauteはそれら二本鎖RNAと結合したのち,一方のRNA鎖を切断し,残った一本鎖RNAをガイド鎖として標的となるmRNAに結合する2).
このように,RNAiにおいてはDicerとArgonauteの両方のタンパク質を必要とするが,モデル生物Saccharomyces cerevisiaeではゲノムにそれらをコードする遺伝子がみつからない.また,S. cerevisiae以外の数種の出芽酵母,Saccharomyces castellii,Kluyveromyces polysporus,Candida albicansでは,Argonauteの遺伝子は同定されているがDicerの遺伝子がみつかっていない.そのため,出芽酵母はRNAi現象のない例外的な真核生物であると考えられてきた.しかしながら最近,Argonauteをもつこれら3種の出芽酵母にはRNAi現象のあることが明らかになった3).ブレイクスルーは新しいDicerの発見であった.
これまで,出芽酵母においてDicerが発見されてこなかったことには理由がある.Dicerのドメイン構造は生物種のあいだで異なるが,2塩基の突出した3’側の末端を認識するPAZドメインと2つの連続したRNase IIIドメインをもつことがDicerのアイデンティティとされてきた.実際,このドメインセットのみで構成されるランブル鞭毛虫(Giardia intestinalis)に由来するDicerは25~27塩基長の二本鎖RNAを生成できる4).また,ランブル鞭毛虫に由来するDicerの結晶構造は,PAZドメインがRNA鎖の末端を固定し,そこからRNase IIIドメインの活性部位までの長さに相当するsiRNA二本鎖が生成されるというモデルをよく説明した4,5).したがって,ゲノムにおいてDicerの遺伝子を探す際には,1つのPAZドメインと2つの連続したRNase IIIドメインをもつ遺伝子がその候補となった.しかし,そのようなドメインセットをもつ遺伝子は出芽酵母のゲノムには存在しなかったのである.そこで,Dicerの遺伝子を探す際の指標を“少なくとも1つのRNase IIIドメインをもつこと”に緩和することが,出芽酵母に特有なDicer(Dcr1)の発見につながった3).このDcr1は長い二本鎖RNAから特定の塩基長のsiRNA二本鎖を生成できるが,従来のDicerのもつドメイン構造とは大きく異なり,PAZドメインをもたずRNase IIIドメインも1つしかない.出芽酵母のもつDcr1がどのような機構でRNAの長さを測定しているかについてはまったくの謎であった.
1.出芽酵母のもつDcr1の全体構造
出芽酵母のもつDcr1は,N末端ドメイン,RNase IIIドメイン,そして,2つの二本鎖RNA結合ドメイン(dsRBD1,dsRBD2)から構成される.当初,K. polysporusに由来するDcr1の全長構造の決定を試みたが,核酸が強固に結合しており全長を高純度に精製することができなかった.そこで,核酸への結合能が低下しつつもDicing活性(特定の塩基長のsiRNA二本鎖を生成する活性)を維持するコンストラクトを模索した.その結果,N末端ドメインからdsRBD1までの領域を含むフラグメント(Dcr1ΔC)が全長構造と同じ程度のDicing活性をもつことがわかった4).出芽酵母のもつDcr1に特徴的なC末端側のdsRBD2こそが特定の長さのsiRNA二本鎖を生成するために必須であろうと考えていたので,この結果は予想外であった.全長構造に比べるとその量は少ないが,Dcr1ΔCには依然として大量の核酸が強固に結合していた.筆者が所属する研究室ではRNaseの使用が全面的に禁止されているため,最終的に5種類のカラムを用いることにより夾雑する核酸を完全に除くことに成功し,Dcr1ΔCの結晶構造を2.3Åの分解能で決定した(図1,PDB ID:3RV0).
Dcr1ΔCの立体構造は,N末端ドメインとRNase IIIドメインがそれぞれ二量体化したコアパートと,溶液において自由に動くと思われるdsRBD1から構成されていた(今回の結晶構造では,片方のdsRBD1のみ電子密度が明瞭であった).二量体化したRNase IIIドメインから両側にdsRBD1の伸びるようすは,細菌のRNase IIIの立体構造と類似していた6)(図2a).RNAプロセシングにかかわる細菌のRNase IIIは,2つのdsRBDにより二本鎖RNAを抱き込んだのち,RNase IIIドメインにある2カ所の活性部位が二本鎖RNAをそれぞれ同時に切断する.今回のDcr1ΔCの立体構造からN末端ドメインがRNase IIIドメインの活性部位の反対側で二量体化することがわかり,Dcr1ΔCは細菌のRNase IIIと同様に二本鎖RNAを結合することが示唆された(図2a).
2.Dcr1は二本鎖RNAのうえで連結する
従来のDicerは,PAZドメインが二本鎖RNAの3’末端を認識しRNase IIIドメインがその反対側を切断することでsiRNA二本鎖を生成する4,5).その際,PAZドメインとRNase IIIドメインとの間隔がsiRNA二本鎖の長さを決定する.これに対し,Dcr1ΔCは二量体化することで二本鎖RNAと結合しこれを切断できるが,PAZドメインに代わり二本鎖RNAの末端を認識できるようなドメインがない.しかしながら,Dcr1ΔCのRNase IIIドメインから両側に突き出たループ,VL1とVL2が列車の連結器のように相補的な形状をもつことに気づいた(図2b).実際,Dcr1ΔC二量体を二本鎖RNAのうえで23塩基長の間隔をもって並べると,Dcr1ΔCどうしは立体障害なく連結することができた(図2 b,c).このことから,出芽酵母のもつDcr1によるsiRNA二本鎖の生成は,末端からではなく内部からはじまるのではないかと考えた.もし,Dcr1二量体が列車の車両のように二本鎖RNAのうえに並ぶことでsiRNA二本鎖を生成しているのであれば,野生型のDcr1ΔCに対し切断活性を失った変異型のDcr1ΔCを混ぜることにより,23塩基の倍数の長さのsiRNA二本鎖が生成されてくるに違いないと予想した.そして予想どおり,切断活性を失った変異型Dcr1ΔCの割合が増えるにつれ,46塩基長,69塩基長,92塩基長のsiRNA二本鎖が生成された.
3.Dcr1は切断ののちもsiRNA二本鎖と結合したままである
さきに述べた機構によりsiRNA二本鎖が連続的に生成されるためには,最初に二本鎖RNAと結合したDcr1二量体がRNAを切断してこれをはなすまえに,つぎのDcr1二量体がそのとなりに結合する必要がある.70塩基長の二本鎖RNAを基質としDcr1ΔCが複数回の反応を触媒する多重代謝回転キネティクス解析を行ったところ,前定常状態におけるsiRNA二本鎖の生成速度はDcr1ΔCの濃度に比例して上昇するが,定常状態におけるsiRNA二本鎖の生成速度はDcr1ΔCの濃度によらず著しく遅かった.この結果から,1回目の切断反応ののちもDcr1ΔCは二本鎖RNAと結合したままでいるために,2回目の切断反応が起こらないことが示唆された.
4.Dcr1による切断部位は二本鎖RNAの2次構造により決定されている
in vitro切断実験においては基質として完全に相補的な500塩基対の二本鎖RNAを用いた.二本鎖RNAに対するDcr1の結合は配列に非特異的であるため,siRNA二本鎖の生成は二本鎖RNAの任意の箇所からはじまる.一方,生体においてDcr1の基質となるRNAは特定の箇所で切断されることがわかっている3).生体における基質RNAは完全に相補的な二本鎖RNAではなく,ミスマッチ塩基対やバルジなどを含み塩基配列に特異的な2次構造をもつであろう.それらが立体障害となりDcr1が結合できる場所を制限することで,生体では特定の箇所からsiRNA二本鎖の生成がはじまるものと考えている.
5.新たにみつかった触媒残基にみるRNase IIIファミリーの進化の過程
これまで,RNase IIIファミリーは分子量とドメイン構造により,クラス1:RNase III(細菌),クラスII:Drosha(真核生物),クラスIII:Dicer(真核生物)の3つのグループに分類されてきた7).RNase IIIファミリーのすべてに共通な特徴として,触媒活性に必須なMg2+の配位に2つのアスパラギン酸残基と2つのグルタミン酸残基が直接または水分子を介しかかわっていることがあげられる.今回,Dcr1ΔCの活性部位において明瞭な八面体の電子密度を確認することができた.結晶化の条件にMg2+を含むことから,この電子密度の中心にはMg2+があるものと考えられた.この活性部位では,さきの保存された4残基にくわえ,アスパラギン残基とリジン残基とがMg2+の配位にくわわっており,それらのアラニン残基への置換体ではいずれもDicing活性の低下が観察された.特筆すべきは,この新たにみつかった2残基はクラスI RNase IIIファミリーでは保存されておらず,クラスIIとクラスIIIのみで保存されていることであった.つまり,出芽酵母のもつDcr1はその立体構造こそ細菌のもつクラスIに似ているものの,触媒残基としては真核生物のもつクラスIIおよびクラスIIIと同じセットをもつハイブリッド酵素であった.この事実は,細菌のRNase IIIが出芽酵母Dcr1をへて真核生物のDroshaやDicerへと進化していった可能性を連想させた.
おわりに
筆者らは,出芽酵母のもつDcr1が二本鎖RNAのうえでとなりあって結合することにより活性部位が一定の間隔で並び,特定の塩基長のsiRNA二本鎖を生成することを明らかにした.この機構は,二本鎖RNAの末端を必要とする従来のDicerによるsiRNA二本鎖の生成機構とはまったく異なることから,出芽酵母のもつDcr1と従来のDicerとは機能収斂のよい例といえよう.また,今回の研究では,RNAiという枠をこえ,分子定規の新しい概念をその実験的な証拠とともに提示できたのではないかと考えている.
文 献
- Tomari, Y. & Zamore, P. D.: Perspective: machines for RNAi. Genes Dev., 19, 517-529 (2005)[PubMed]
- Siomi, H. & Siomi, M. C.: On the road to reading the RNA-interference code. Nature, 457, 396-404 (2009)[PubMed]
- Drinnenberg, I. A., Weinberg, D. E., Xie, K. T. et al.: RNAi in budding yeast. Science, 326, 544-550 (2009)[PubMed]
- Macrae, I. J., Zhou, K., Li, F. et al.: Structural basis for double-stranded RNA processing by Dicer. Science, 311, 195-198 (2006)[PubMed]
- Zhang, H., Kolb, F. A., Jaskiewicz, L. et al.: Single processing center models for human Dicer and bacterial RNase III. Cell, 118, 57-68 (2004)[PubMed]
- Gan, J., Tropea, J., Austin, B. P. et al.: Structural insight into the mechanism of double-stranded RNA processing by ribonuclease III. Cell, 124, 355-366 (2006)[PubMed]
- Du, Z., Lee, J. K., Tjhen, R. et al.: Structural and biochemical insights into the dicing mechanism of mouse Dicer: a conserved lysine is critical for dsRNA cleavage. Proc. Natl. Acad. Sci. USA, 105, 2391-2396 (2008)[PubMed]
著者プロフィール
略歴:2007年 東京工業大学大学院生命理工学研究科 修了,同年 同 博士研究員,2008年 米国Memorial Sloan-Kettering Cancer Centerポスドク研究員を経て,2011年より同 リサーチアソシエート.
研究テーマ:ノンコーディングRNAによる制御機構の解明.
抱負:立体構造からみえてくる新しい知見をもとに生命現象の謎を解き明かしたい.
© 2011 中西 孝太郎 Licensed under CC 表示 2.1 日本