リベロマイシンAにおけるスピロアセタール構造の形成機構の解明
高橋俊二・長田裕之
(理化学研究所基幹研究所 ケミカルバイオロジー研究基盤施設)
email:高橋俊二,長田裕之
DOI: 10.7875/first.author.2011.099
Reveromycin A biosynthesis uses RevG and RevJ for stereospecific spiroacetal formation.
Shunji Takahashi, Atsushi Toyoda, Yasuyo Sekiyama, Hiroshi Takagi, Toshihiko Nogawa, Masakazu Uramoto, Ryuichiro Suzuki, Hiroyuki Koshino, Takuto Kumano, Suresh Panthee, Tohru Dairi, Jun Ishikawa, Haruo Ikeda, Yoshiyuki Sakaki, Hiroyuki Osada
Nature Chemical Biology, 7, 461-481 (2011)
スピロアセタール化合物は自然界に広く存在し,その立体特異的な構造による広範囲の薬理活性が知られている.立体特異的なスピロアセタール構造の生合成機構を解明することは化学合成が困難な化合物の効率的な生産につながる.筆者らは,破骨細胞に選択的にアポトーシスを誘導することで,骨吸収の抑制活性やがんの骨転移の抑制活性をもつスピロアセタール化合物リベロマイシンAの生合成遺伝子クラスターの同定に成功した.遺伝子およびタンパク質の機能を詳細に解析した結果,長いあいだ非酵素的に進行すると考えられてきた脱水環化による立体特異的なスピロアセタール構造の形成には,ジヒドロキシケトン合成酵素とスピロアセタール合成酵素が関与していることを見い出した.
微生物は臨床的に重要な薬剤として使われるさまざまなポリケチド化合物(エリスロマイシン,エポチロン,ラパマイシン,ロバスタチンなど)を生産する.ポリケチド化合物の構造の多様化にかかわる酵素反応を解明することは,新しい薬剤の開発に必要な生合成の理論構築につながる.筆者らは,細菌,渦鞭毛藻,海綿動物から広く見い出されているポリケチド化合物のなかで,さまざまな生理活性(イオノホア,プロテインホスファターゼ阻害,駆虫作用,抗菌作用など)をもつことで知られ立体特異的な構造の形成機構に不明な点の多いスピロアセタール化合物に着目した.
1991年,がん細胞の増殖を阻害する物質として放線菌(Streptomyces sp. SN-593)からリベロマイシンAが見い出された1).リベロマイシンAの細胞内での標的はイソロイシルtRNA合成酵素であり2),破骨細胞に選択的にアポトーシスを誘導し骨吸収を阻害する活性をもつ3).破骨細胞に選択的に作用することで肺がんや前立腺がんにより誘導される骨転移を阻害することも明らかになっている4,5).リベロマイシンAのスピロアセタール立体構造の構築は難題であったが有機化学者を魅了し,多段階のステップをへて有機合成が達成された6).一方,生合成に関しては酵素や不安定な生合成中間体の同定が困難であったことから知見がなかった.立体特異的なスピロアセタール構造は生理活性に重要であることから,その遺伝子クラスターを同定し生合成機構を解明することは効率的な化合物の創製手法に道を開くものと考えられている.
もっとも研究の進んでいるポリエーテル化合物であるモネンシンAはスピロアセタール構造をもちエポキシド-ケトン中間体を経由する環化反応が知られている.トウトマイシンおよびエバーメクチンの生合成では脱水によるスピロアセタール構造の環化機構が予想されている.これらの環化反応は生合成遺伝子クラスターが同定されているにもかかわらず環化にかかわる遺伝子が見い出されていないことから,反応は非酵素的に進行すると考えられてきた.一方,スピランジエンAの生合成においては環化反応の過程にP450の関与が予想されているが,詳細な機構は明らかにされていない.また,リベロマイシンAの環化機構については知見がまったく存在しない.このように,広範な解析にもかかわらず脱水環化によるスピロアセタール構造の形成にかかわる生化学的な機構はまったく証明されていなかった.そこで,筆者らは,これらの問題に答えるため,化学的,分子生物学的,生化学的な手法を用いてリベロマイシンAの生合成機構を包括的に解析した.
放線菌2次代謝産物の生合成遺伝子はクラスターを形成している.遺伝子クラスターをクローニングするには,特徴的な化学構造の形成に必須の遺伝子をプローブとしてハイブリダイゼーションにより類似遺伝子を検出する手法が一般的である.リベロマイシンA(図1)の場合,スピロアセタール構造の形成やサクシニル基の転移にかかわる遺伝子が不明であり,関与が容易に予想できるP450およびポリケチド合成酵素は放線菌のゲノムに相同性の高い遺伝子が多数存在するため,特異的なDNAプローブをデザインすることが困難であった.そこで,高生産培地を開発しリベロマイシン生合成遺伝子を選択的に増幅する手法を考えた.
通常,Streptomyces sp. SN-593によるリベロマイシンの生産量は少ないが,長年,検討されてきた培養液にトマトエキスをくわえると生産性が飛躍的に向上する.そこで,生産培地と非生産培地での遺伝子発現の差を利用すれば,リベロマイシンを生産するときに特異的に発現するポリケチド合成酵素遺伝子の同定が可能である.RT-PCR法により特異的に発現が増大する遺伝子を選別し,さらに,これをプローブとしてホスミドライブラリーから遺伝子の再探索を行った.この研究では同時にStreptomyces. sp. SN-593のゲノム配列のドラフト解析も行ったが,ポリケチド合成酵素の遺伝子配列の連結が困難であることから,発現遺伝子の情報とホスミドクローンの情報とを統合することで遺伝子配列を確定する必要があった.以上の結果,リベロマイシン生合成にかかわるポリケチド合成酵素遺伝子群revA,revB,revC,revDと,その近傍に存在する制御遺伝子や修飾酵素遺伝子を含む約91 kbにおよぶ遺伝子クラスターを見い出すことに成功した.
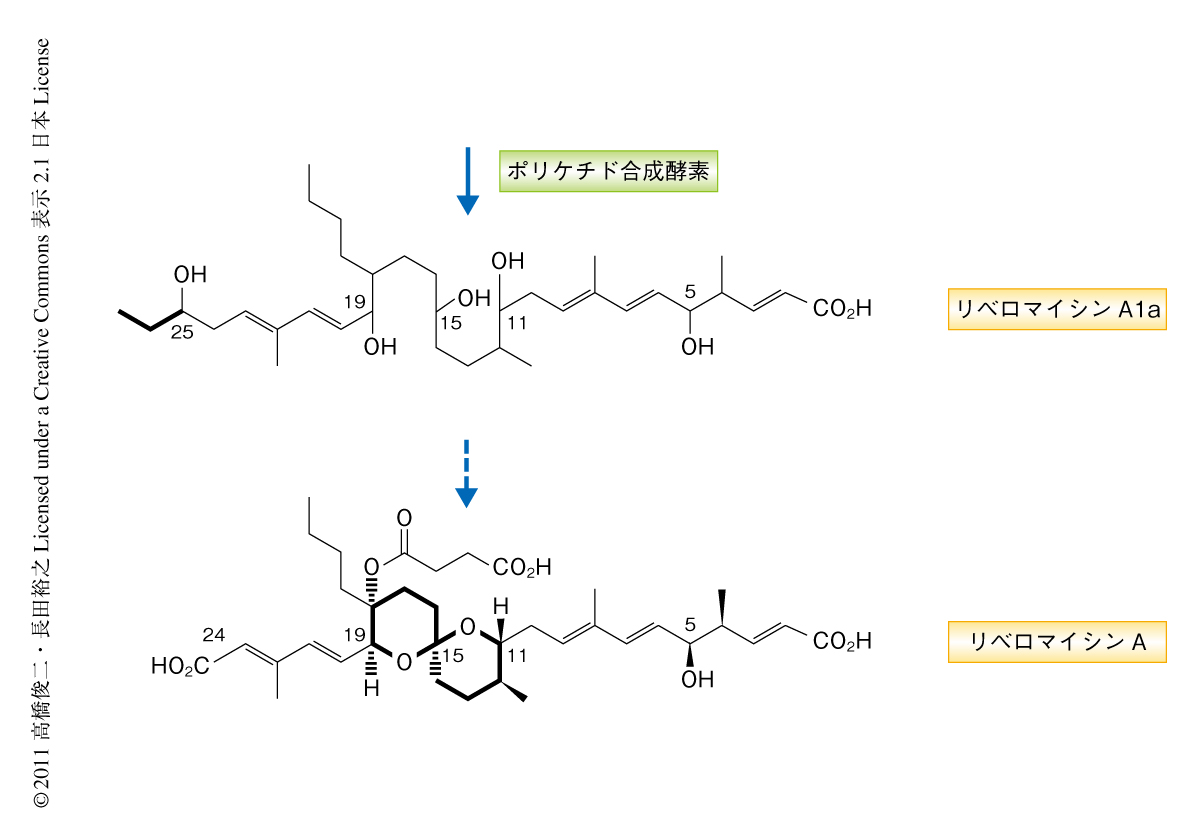
I型ポリケチド合成酵素はケトシンターゼ,アシルトランスフェラーゼ,アシルキャリアータンパク質の基本構造にくわえ,ケトレダクターゼ,デヒドラターゼ,エノイルレダクターゼのそれぞれのドメイン構成にしたがい正確に還元や脱水をうけた炭素鎖が伸長して生合成されることが知られている.RevA,RevB,RevC,RevDの機能ドメインを詳細に解析したところリベロマイシンAの炭素骨格の構成とほぼ一致していたが,最終的な生合成産物の構造には認められない余分なプロピオン酸ユニットの付加したスピロアセタール環化前駆体の存在が予想できた.
ポリケチド合成酵素のドメイン構成から推測されたこの前駆体が生合成の過程において実際に存在するかどうかを検証するため,野生株の生産するリベロマイシン類縁体を系統的に分離および精製したところ,予想された余分なプロピオン酸ユニットを含む環化前駆体リベロマイシンA1aを見い出すことができた(図1).一方,得られた中間体が生合成の真の中間体であるかどうかを検証することは重要であり,ポリケチド骨格を生合成することのできないようポリケチド合成酵素遺伝子を破壊した株を作製し,この破壊株にリベロマイシンA1aを添加するとリベロマイシンAの生産が回復することにより生合成中間体であることを証明した.リベロマイシンA1aはスピロアセタール構造の形成機構を生化学的に解明するための重要な基質として利用した.
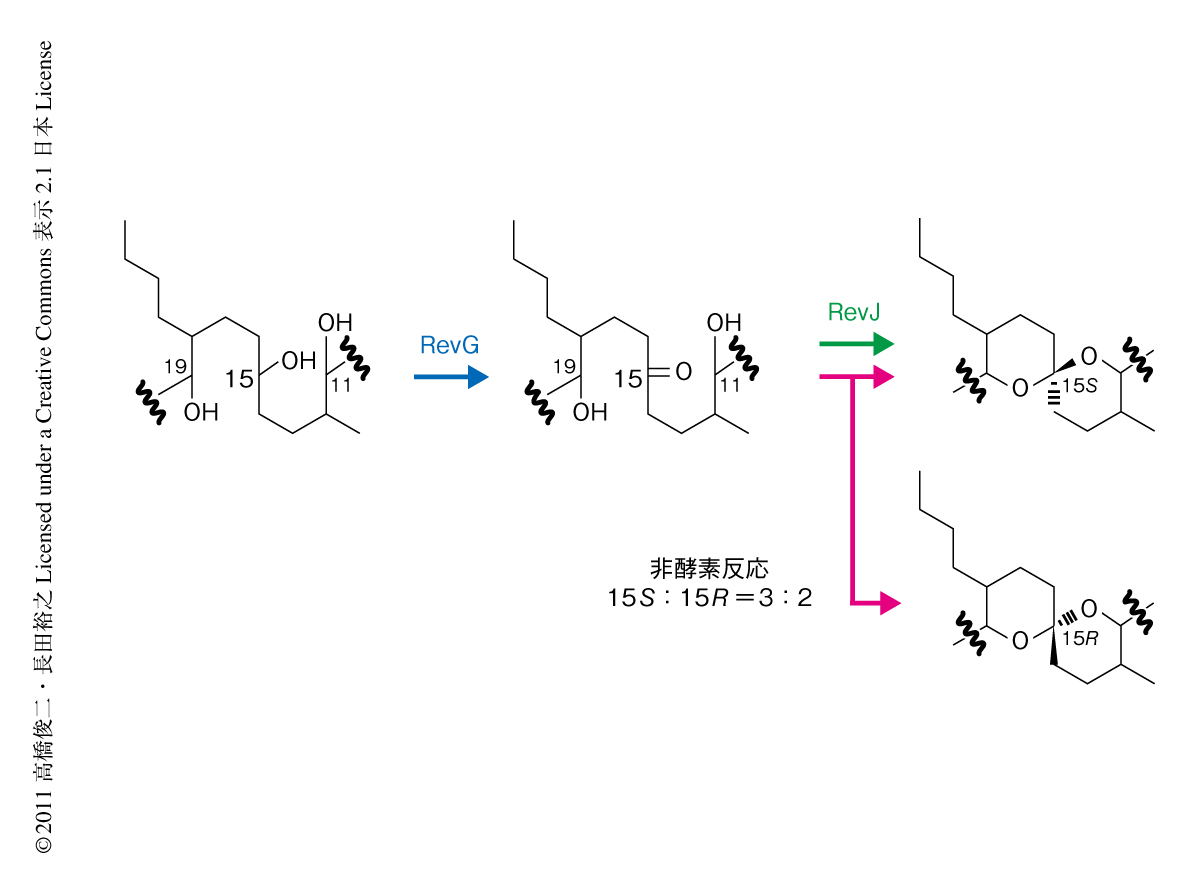
リベロマイシンA1aの同定により,スピロアセタール構造の形成がC11,15,19トリオール構造からの脱水環化反応であることが予想されたが,確証を得るため,[1-13C,18O]酢酸および[1-13C,18O]プロピオン酸の取り込みののちNMR解析を行うことでC-O結合の起源を追跡した.その結果,リベロマイシンの生合成はエポキシド-ケトン経由ではなくジヒドロキシケトンを経由する脱水環化機構であることが証明された.
スピロアセタール環の形成にかかわる遺伝子を同定するためには遺伝子破壊により生合成経路を遮断し蓄積する代謝産物を解析することが重要である.そこで,生合成クラスターにおいて脱水素酵素と機能アノテーションされるRevGの遺伝子破壊を行った.revG破壊株ではポリケチド合成酵素の構造から予測されたとおりリベロマイシンA1aを蓄積したことから,RevGがリベロマイシンA1aを基質として脱水素反応を行うことが強く示唆された.RevGを精製し酵素変換産物を解析したところ,補酵素NAD+の存在下でジヒドロキシケトンを生成することが判明した.このRevG反応産物は弱酸性の培地において非酵素的に環化が進行し15Sおよび15Rと立体構造の異なるスピロアセタール化合物を3:2の割合で生成した(図2).一方,リベロマイシン生産菌は15Sのスピロアセタール構造のみを立体特異的に生産することから,熱力学的に安定な構造に依存して非酵素的に環化が起こっているのではなくスピロアセタール立体制御酵素が存在していることを予想し,生合成遺伝子クラスターに存在する機能不明遺伝子の破壊による解析を進めた.不安定なジヒドロキシケトン中間体を基質とするために立体制御酵素遺伝子の破壊株ではリベロマイシンAの生産量が減少すると予想し,遺伝子破壊による表現型が一致するrevJ遺伝子を見い出した.立体特異的な環化反応を証明するためRevGおよびRevJを精製してリベロマイシンA1aを基質とするカップリング反応を解析した.弱酸性条件での非酵素的な環化を抑えるため反応産物の解析は弱アルカリ条件で行った.その結果,RevJが反応系に存在するときのみスピロアセタール環化反応が効率よく進行し,さらに,反応産物の立体構造はすべて15Sであることを見い出した.
ジヒドロキシケトン合成酵素RevGとスピロアセタール合成酵素RevJの相同性検索の結果,近年,公開されたスピロファンジンA 7) 生産菌のゲノムのドラフト配列から,ともに80%以上の相同性を示す遺伝子が近接して存在する遺伝子クラスターの存在が明らかになった.スピロファンジンAはリベロマイシンAと同様のスピロアセタール構造をもつことから,共通の環化機構の存在が推測された.RevJはTyr-Val-Thr-Asnの4アミノ酸残基が7回くり返す配列を含むが,RevJと45%以上の相同性をもつ配列が古細菌の細胞表面タンパク質に見い出されている8).このタンパク質の生理機能は不明であるが,結晶構造解析からβプロペラドメイン構造をもつことが明らかになっている9).RevJも同様な立体構造をもつと考えられるが,結晶解析によるスピロアセタールの立体構造の制御機構の解明は今後の研究の進展の鍵である.
これまでに,脱水環化反応によるスピロアセタール構造の形成は非酵素的に進行すると考えられてきたが,立体特異的なスピロアセタール構造の形成にかかわる2つの酵素,ジヒドロキシケトン合成酵素とスピロアセタール合成酵素が明らかになった.放線菌は多段階の化学合成により誘導する必要のある化合物を,安定なトリオール体を基質として環化前駆体をつくったのち,立体特異的なスピロアセタール化合物を選択的に生産できる.生物の遺伝子探索から解明された効率的な生合成機構を用いることにより,生命科学および医薬の分野に貢献する新たな生理活性化合物の創製研究の進展が期待される.
略歴:1997年 千葉大学大学院自然科学研究科博士課程 修了,同年 東京大学分子細胞生物学研究所 研究員,1998年 千葉大学大学院医学研究院 助手,2002年 米国Kentucky大学 博士研究員,2005年 理化学研究所長田抗生物質研究室 協力研究員を経て,2009年より理化学研究所基幹研究所 専任研究員.
研究テーマ:リベロマイシンの生合成機構.
関心事:放線菌の生合成遺伝子の活性化による新規2次代謝産物の生産.
長田 裕之(Hiroyuki Osada)
理化学研究所基幹研究所 施設長.
研究室URL:http://cbcore.riken.jp/ja.html
© 2011 高橋俊二・長田裕之 Licensed under CC 表示 2.1 日本
(理化学研究所基幹研究所 ケミカルバイオロジー研究基盤施設)
email:高橋俊二,長田裕之
DOI: 10.7875/first.author.2011.099
Reveromycin A biosynthesis uses RevG and RevJ for stereospecific spiroacetal formation.
Shunji Takahashi, Atsushi Toyoda, Yasuyo Sekiyama, Hiroshi Takagi, Toshihiko Nogawa, Masakazu Uramoto, Ryuichiro Suzuki, Hiroyuki Koshino, Takuto Kumano, Suresh Panthee, Tohru Dairi, Jun Ishikawa, Haruo Ikeda, Yoshiyuki Sakaki, Hiroyuki Osada
Nature Chemical Biology, 7, 461-481 (2011)
要 約
スピロアセタール化合物は自然界に広く存在し,その立体特異的な構造による広範囲の薬理活性が知られている.立体特異的なスピロアセタール構造の生合成機構を解明することは化学合成が困難な化合物の効率的な生産につながる.筆者らは,破骨細胞に選択的にアポトーシスを誘導することで,骨吸収の抑制活性やがんの骨転移の抑制活性をもつスピロアセタール化合物リベロマイシンAの生合成遺伝子クラスターの同定に成功した.遺伝子およびタンパク質の機能を詳細に解析した結果,長いあいだ非酵素的に進行すると考えられてきた脱水環化による立体特異的なスピロアセタール構造の形成には,ジヒドロキシケトン合成酵素とスピロアセタール合成酵素が関与していることを見い出した.
はじめに
微生物は臨床的に重要な薬剤として使われるさまざまなポリケチド化合物(エリスロマイシン,エポチロン,ラパマイシン,ロバスタチンなど)を生産する.ポリケチド化合物の構造の多様化にかかわる酵素反応を解明することは,新しい薬剤の開発に必要な生合成の理論構築につながる.筆者らは,細菌,渦鞭毛藻,海綿動物から広く見い出されているポリケチド化合物のなかで,さまざまな生理活性(イオノホア,プロテインホスファターゼ阻害,駆虫作用,抗菌作用など)をもつことで知られ立体特異的な構造の形成機構に不明な点の多いスピロアセタール化合物に着目した.
1991年,がん細胞の増殖を阻害する物質として放線菌(Streptomyces sp. SN-593)からリベロマイシンAが見い出された1).リベロマイシンAの細胞内での標的はイソロイシルtRNA合成酵素であり2),破骨細胞に選択的にアポトーシスを誘導し骨吸収を阻害する活性をもつ3).破骨細胞に選択的に作用することで肺がんや前立腺がんにより誘導される骨転移を阻害することも明らかになっている4,5).リベロマイシンAのスピロアセタール立体構造の構築は難題であったが有機化学者を魅了し,多段階のステップをへて有機合成が達成された6).一方,生合成に関しては酵素や不安定な生合成中間体の同定が困難であったことから知見がなかった.立体特異的なスピロアセタール構造は生理活性に重要であることから,その遺伝子クラスターを同定し生合成機構を解明することは効率的な化合物の創製手法に道を開くものと考えられている.
もっとも研究の進んでいるポリエーテル化合物であるモネンシンAはスピロアセタール構造をもちエポキシド-ケトン中間体を経由する環化反応が知られている.トウトマイシンおよびエバーメクチンの生合成では脱水によるスピロアセタール構造の環化機構が予想されている.これらの環化反応は生合成遺伝子クラスターが同定されているにもかかわらず環化にかかわる遺伝子が見い出されていないことから,反応は非酵素的に進行すると考えられてきた.一方,スピランジエンAの生合成においては環化反応の過程にP450の関与が予想されているが,詳細な機構は明らかにされていない.また,リベロマイシンAの環化機構については知見がまったく存在しない.このように,広範な解析にもかかわらず脱水環化によるスピロアセタール構造の形成にかかわる生化学的な機構はまったく証明されていなかった.そこで,筆者らは,これらの問題に答えるため,化学的,分子生物学的,生化学的な手法を用いてリベロマイシンAの生合成機構を包括的に解析した.
1.生合成遺伝子クラスターの同定
放線菌2次代謝産物の生合成遺伝子はクラスターを形成している.遺伝子クラスターをクローニングするには,特徴的な化学構造の形成に必須の遺伝子をプローブとしてハイブリダイゼーションにより類似遺伝子を検出する手法が一般的である.リベロマイシンA(図1)の場合,スピロアセタール構造の形成やサクシニル基の転移にかかわる遺伝子が不明であり,関与が容易に予想できるP450およびポリケチド合成酵素は放線菌のゲノムに相同性の高い遺伝子が多数存在するため,特異的なDNAプローブをデザインすることが困難であった.そこで,高生産培地を開発しリベロマイシン生合成遺伝子を選択的に増幅する手法を考えた.
通常,Streptomyces sp. SN-593によるリベロマイシンの生産量は少ないが,長年,検討されてきた培養液にトマトエキスをくわえると生産性が飛躍的に向上する.そこで,生産培地と非生産培地での遺伝子発現の差を利用すれば,リベロマイシンを生産するときに特異的に発現するポリケチド合成酵素遺伝子の同定が可能である.RT-PCR法により特異的に発現が増大する遺伝子を選別し,さらに,これをプローブとしてホスミドライブラリーから遺伝子の再探索を行った.この研究では同時にStreptomyces. sp. SN-593のゲノム配列のドラフト解析も行ったが,ポリケチド合成酵素の遺伝子配列の連結が困難であることから,発現遺伝子の情報とホスミドクローンの情報とを統合することで遺伝子配列を確定する必要があった.以上の結果,リベロマイシン生合成にかかわるポリケチド合成酵素遺伝子群revA,revB,revC,revDと,その近傍に存在する制御遺伝子や修飾酵素遺伝子を含む約91 kbにおよぶ遺伝子クラスターを見い出すことに成功した.
2.遺伝子破壊による生合成中間体の同定
I型ポリケチド合成酵素はケトシンターゼ,アシルトランスフェラーゼ,アシルキャリアータンパク質の基本構造にくわえ,ケトレダクターゼ,デヒドラターゼ,エノイルレダクターゼのそれぞれのドメイン構成にしたがい正確に還元や脱水をうけた炭素鎖が伸長して生合成されることが知られている.RevA,RevB,RevC,RevDの機能ドメインを詳細に解析したところリベロマイシンAの炭素骨格の構成とほぼ一致していたが,最終的な生合成産物の構造には認められない余分なプロピオン酸ユニットの付加したスピロアセタール環化前駆体の存在が予想できた.
ポリケチド合成酵素のドメイン構成から推測されたこの前駆体が生合成の過程において実際に存在するかどうかを検証するため,野生株の生産するリベロマイシン類縁体を系統的に分離および精製したところ,予想された余分なプロピオン酸ユニットを含む環化前駆体リベロマイシンA1aを見い出すことができた(図1).一方,得られた中間体が生合成の真の中間体であるかどうかを検証することは重要であり,ポリケチド骨格を生合成することのできないようポリケチド合成酵素遺伝子を破壊した株を作製し,この破壊株にリベロマイシンA1aを添加するとリベロマイシンAの生産が回復することにより生合成中間体であることを証明した.リベロマイシンA1aはスピロアセタール構造の形成機構を生化学的に解明するための重要な基質として利用した.
3.スピロアセタール環の形成にかかわる酵素の発見
リベロマイシンA1aの同定により,スピロアセタール構造の形成がC11,15,19トリオール構造からの脱水環化反応であることが予想されたが,確証を得るため,[1-13C,18O]酢酸および[1-13C,18O]プロピオン酸の取り込みののちNMR解析を行うことでC-O結合の起源を追跡した.その結果,リベロマイシンの生合成はエポキシド-ケトン経由ではなくジヒドロキシケトンを経由する脱水環化機構であることが証明された.
スピロアセタール環の形成にかかわる遺伝子を同定するためには遺伝子破壊により生合成経路を遮断し蓄積する代謝産物を解析することが重要である.そこで,生合成クラスターにおいて脱水素酵素と機能アノテーションされるRevGの遺伝子破壊を行った.revG破壊株ではポリケチド合成酵素の構造から予測されたとおりリベロマイシンA1aを蓄積したことから,RevGがリベロマイシンA1aを基質として脱水素反応を行うことが強く示唆された.RevGを精製し酵素変換産物を解析したところ,補酵素NAD+の存在下でジヒドロキシケトンを生成することが判明した.このRevG反応産物は弱酸性の培地において非酵素的に環化が進行し15Sおよび15Rと立体構造の異なるスピロアセタール化合物を3:2の割合で生成した(図2).一方,リベロマイシン生産菌は15Sのスピロアセタール構造のみを立体特異的に生産することから,熱力学的に安定な構造に依存して非酵素的に環化が起こっているのではなくスピロアセタール立体制御酵素が存在していることを予想し,生合成遺伝子クラスターに存在する機能不明遺伝子の破壊による解析を進めた.不安定なジヒドロキシケトン中間体を基質とするために立体制御酵素遺伝子の破壊株ではリベロマイシンAの生産量が減少すると予想し,遺伝子破壊による表現型が一致するrevJ遺伝子を見い出した.立体特異的な環化反応を証明するためRevGおよびRevJを精製してリベロマイシンA1aを基質とするカップリング反応を解析した.弱酸性条件での非酵素的な環化を抑えるため反応産物の解析は弱アルカリ条件で行った.その結果,RevJが反応系に存在するときのみスピロアセタール環化反応が効率よく進行し,さらに,反応産物の立体構造はすべて15Sであることを見い出した.
ジヒドロキシケトン合成酵素RevGとスピロアセタール合成酵素RevJの相同性検索の結果,近年,公開されたスピロファンジンA 7) 生産菌のゲノムのドラフト配列から,ともに80%以上の相同性を示す遺伝子が近接して存在する遺伝子クラスターの存在が明らかになった.スピロファンジンAはリベロマイシンAと同様のスピロアセタール構造をもつことから,共通の環化機構の存在が推測された.RevJはTyr-Val-Thr-Asnの4アミノ酸残基が7回くり返す配列を含むが,RevJと45%以上の相同性をもつ配列が古細菌の細胞表面タンパク質に見い出されている8).このタンパク質の生理機能は不明であるが,結晶構造解析からβプロペラドメイン構造をもつことが明らかになっている9).RevJも同様な立体構造をもつと考えられるが,結晶解析によるスピロアセタールの立体構造の制御機構の解明は今後の研究の進展の鍵である.
おわりに
これまでに,脱水環化反応によるスピロアセタール構造の形成は非酵素的に進行すると考えられてきたが,立体特異的なスピロアセタール構造の形成にかかわる2つの酵素,ジヒドロキシケトン合成酵素とスピロアセタール合成酵素が明らかになった.放線菌は多段階の化学合成により誘導する必要のある化合物を,安定なトリオール体を基質として環化前駆体をつくったのち,立体特異的なスピロアセタール化合物を選択的に生産できる.生物の遺伝子探索から解明された効率的な生合成機構を用いることにより,生命科学および医薬の分野に貢献する新たな生理活性化合物の創製研究の進展が期待される.
文 献
- Osada, H., Koshino, H., Isono, K. et al.: Reveromycin A, a new antibiotic which inhibits the mitogenic activity of epidermal growth factor. J. Antibiot., 44, 259-261 (1991)[PubMed]
- Cui, Z., Hirata, D., Tsuchiya, E. et al.: The multidrug resistance-associated protein (MRP) subfamily (Yrs1/Yor1) of Saccharomyces cerevisiae is important for the tolerance to a broad range of organic anions. J. Biol. Chem., 271, 14712-14716 (1996)[PubMed]
- Woo, J. T., Kawatani, M., Kato, M. et al.: Reveromycin A, an agent for osteoporosis, inhibits bone resorption by inducing apoptosis specifically in osteoclasts. Proc. Natl. Acad. Sci. USA, 103, 4729-4734 (2006)[PubMed]
- Muguruma, H., Yano, S., Kakiuchi, S. et al.: Reveromycin A inhibits osteolytic bone metastasis of small-cell lung cancer cells, SBC-5, through an antiosteoclastic activity. Clin. Cancer Res., 11, 8822-8828 (2005)[PubMed]
- Yano, A., Tsutsumi, S., Soga, S. et al.: Inhibition of Hsp90 activates osteoclast c-Src signaling and promotes growth of prostate carcinoma cells in bone. Proc. Natl. Acad. Sci. USA, 105, 15541-15546 (2008)[PubMed]
- Shimizu, T., Masuda, T., Hiramoto, K. et al.: Total synthesis of reveromycin A. Org. Lett., 2, 2153-2156 (2000)[PubMed]
- Holtzel, A., Kempter, C., Metzger, J. W. et al.: Spirofungin, a new antifungal antibiotic from Streptomyces violaceusniger Tu 4113. J. Antibiot., 51, 699-707 (1998)[PubMed]
- Mayerhofer, L.E., Conway de Macario, E., Yao, R. et al.: Structure, organization, and expression of genes coding for envelope components in the archaeon Methanosarcina mazei S-6. Arch. Microbiol., 169, 339-345 (1998)[PubMed]
- Jing, H., Takagi, J., Liu, J. H. et al.: Archaeal surface layer proteins contain βpropeller, PKD, and β helix domains and are related to metazoan cell surface proteins. Structure, 10, 1453-1464 (2002)[PubMed]
著者プロフィール
略歴:1997年 千葉大学大学院自然科学研究科博士課程 修了,同年 東京大学分子細胞生物学研究所 研究員,1998年 千葉大学大学院医学研究院 助手,2002年 米国Kentucky大学 博士研究員,2005年 理化学研究所長田抗生物質研究室 協力研究員を経て,2009年より理化学研究所基幹研究所 専任研究員.
研究テーマ:リベロマイシンの生合成機構.
関心事:放線菌の生合成遺伝子の活性化による新規2次代謝産物の生産.
長田 裕之(Hiroyuki Osada)
理化学研究所基幹研究所 施設長.
研究室URL:http://cbcore.riken.jp/ja.html
© 2011 高橋俊二・長田裕之 Licensed under CC 表示 2.1 日本