マウスRasgrf1遺伝子のインプリンティングとDNAメチル化にはpiRNAおよびその標的となる非コードRNAが必要である
佐々木裕之1・渡部聡朗2
(1九州大学生体防御医学研究所 エピゲノム学分野,2米国Yale大学School of Medicine,Department of Cell Biology)
email:佐々木裕之
DOI: 10.7875/first.author.2011.087
Role for piRNAs and noncoding RNA in de novo DNA methylation of the imprinted mouse Rasgrf1 locus.
Toshiaki Watanabe, Shin-ichi Tomizawa, Kohzoh Mitsuya, Yasushi Totoki, Yasuhiro Yamamoto, Satomi Kuramochi-Miyagawa, Naoko Iida, Yuko Hoki, Patrick J. Murphy, Atsushi Toyoda, Kengo Gotoh, Hitoshi Hiura, Takahiro Arima, Asao Fujiyama, Takashi Sado, Tatsuhiro Shibata, Toru Nakano, Haifan Lin, Kenji Ichiyanagi, Paul D. Soloway, Hiroyuki Sasaki
Science, 332, 848-852 (2011)
インプリンティングは雌雄の生殖細胞で確立されるDNAメチル化の違いにもとづき両親に由来する対立遺伝子のうち一方だけが発現する現象である.インプリンティングは哺乳類の正常な発生に必須で,その異常は奇形症候群や腫瘍を起こす.しかしながら,生殖細胞においてどのような機構により特定の遺伝子が選ばれDNAメチル化されているのかはほとんどわかっていない.筆者らは,レトロトランスポゾンを抑制するpiRNA経路がマウスのインプリント遺伝子Rasgrf1のDNAメチル化に必須であることをみつけた.この遺伝子のインプリンティングには雄の生殖細胞におけるDNAメチル化が必要だが,Rasgrf1遺伝子とは別の染色体に存在するレトロトランスポゾン配列から産生されるpiRNAが,Rasgrf1遺伝子の制御領域から転写される非コードRNAと相互作用していることが明らかになった.筆者らは,piRNAが非コードRNAとの相互作用を介してDNAメチル化酵素を特定の配列へとリクルートするモデルを提唱した.
ゲノムインプリンティング(以下,インプリンティング)をうける遺伝子の近傍にはメチル化可変領域(differentially methylated region:DMR)とよばれる領域があり,この領域のDNAメチル化の状態が対立遺伝子に特異的な発現を制御している.メチル化可変領域における両親に由来するDNAメチル化の違いは雌雄の生殖細胞において確立され,これが受精卵にもち込まれて維持されたものである1,2).しかしながら,雌雄の生殖細胞においてどのような機構で特定の遺伝子が選ばれDNAメチル化されているのかはほとんどわかっていない.
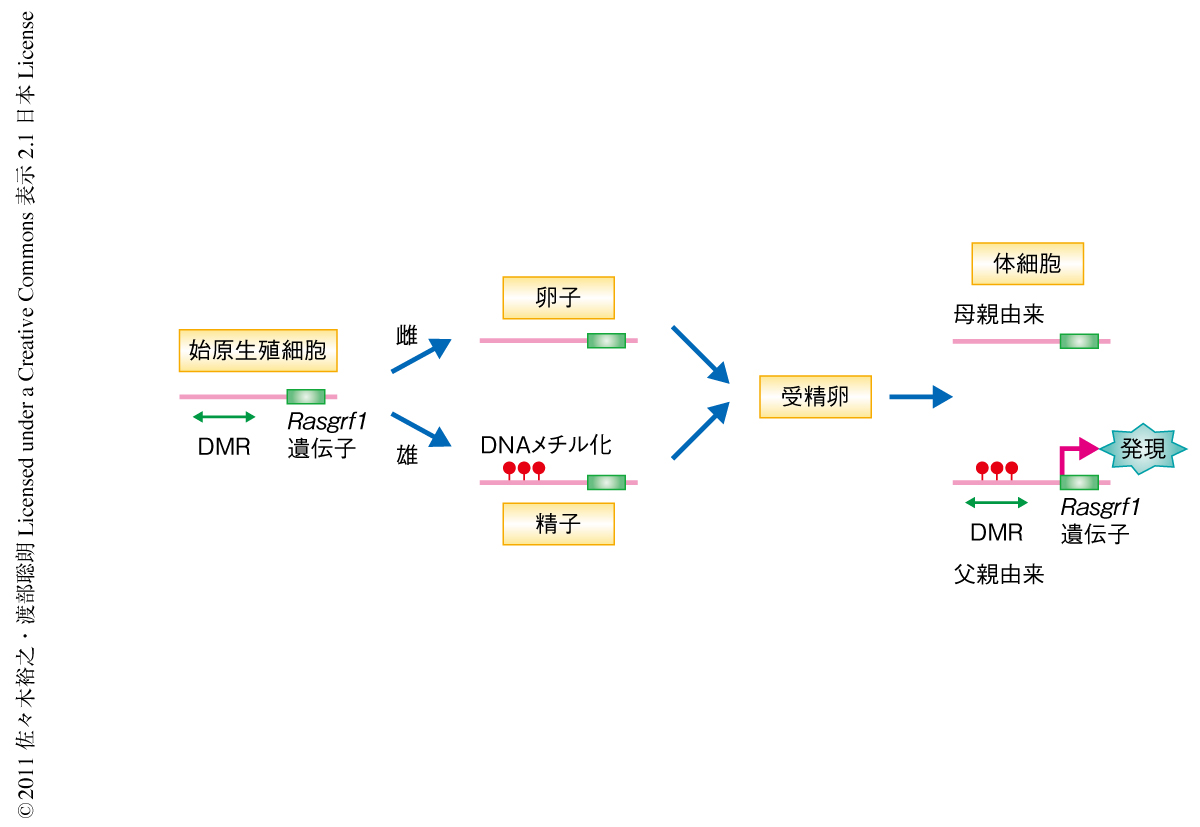
胎生12.5日の始原生殖細胞においてメチル化可変領域はDNA脱メチル化状態であり(インプリントは消去されている),そののち,雄の生殖細胞系列では胎生12.5~18.5日の前精原細胞(または,ゴノサイトとよばれる)において新規のDNAメチル化が起こる(インプリントが確立される).マウスのRasgrf1遺伝子はこのように雄の生殖細胞でDNAメチル化されるインプリント遺伝子である3)(図1).一方,雄の生殖細胞の発生過程においてメチル化可変領域がDNAメチル化される時期は,IAPやLINE-1(または,L1)などのレトロトランスポゾン配列がDNAメチル化される時期と一致する3).これらレトロトランスポゾン配列のDNAメチル化には,DNAメチル化酵素Dnmt3aおよびDnmt3bと,PIWIタンパク質,および,piRNA(PIWI-interacting RNApiRNA)が必要であることがわかっている3-5).piRNAは24~30ヌクレオチド程度の生殖巣に特異的なRNAでPIWIタンパク質と結合する.PIWIタンパク質はpiRNAをガイドとして標的RNA(おもに,レトロトランスポゾン配列に由来)を認識し分解するが,レトロトランスポゾン配列のDNAメチル化にもかかわっている.しかし,その機構は不明である.DNAメチル化酵素Dnmt3aのみでDNAメチル化されるおおかたのインプリント遺伝子とは異なり,Rasgrf1遺伝子のDNAメチル化には別のDNAメチル化酵素Dnmt3bもかかわることがわかっている3,6).そこで,筆者らは,piRNAの生合成および作用経路がRasgrf1遺伝子のDNAメチル化とインプリンティングの特異性の決定にかかわるのではないかとの仮説をたてた.
生殖細胞におけるメチル化可変領域のDNAメチル化にpiRNAがかかわるかどうかを調べるため,Mili遺伝子,Miwi2遺伝子,MitoPLD遺伝子の変異マウスを解析した.Mili遺伝子とMiwi2遺伝子はPIWIファミリーのメンバーを,MitoPLD遺伝子はミトコンドリア膜に存在するホスホリパーゼをコードしており,これらの変異マウスでは精巣のpiRNAが減少し減数分裂期に精子の形成が停止する5,7).変異マウスの精原細胞においてH19遺伝子,Dlk1遺伝子-Gtl2遺伝子,Gpr1遺伝子-Zdbf2遺伝子,Rasgrf1遺伝子のメチル化可変領域(すべて,雄の生殖細胞においてDNAメチル化される)を調べたところ,Rasgrf1遺伝子のメチル化可変領域においてのみDNAメチル化レベルの低下がみられた.影響をうけたのはCpG配列が比較的密に存在する約2 kbの領域で,野生型の精原細胞では77.2~93.4%がDNAメチル化されていたのに対し,Mili変異体で23.3%,Miwi2変異体で58.6%,MitoPLD変異体では16.8%がDNAメチル化されていた.この領域のDNAメチル化がRasgrf1遺伝子のインプリンティングに必須であることはすでに示されており8),今回の結果は,piRNAがRasgrf1遺伝子のインプリンティングに関与することを遺伝学的に証明するものであった.
つぎに,マウス胎仔の精巣から小分子RNAライブラリーを作製し超高速シークエンサーによる網羅的な配列解析を行った.Mili変異体,Miwi2変異体,MitoPLD変異体の3つのpiRNA経路の変異体ではいずれもpiRNAの減少がみられたが,その程度はさきに述べたDNAメチル化の低下とおおまかに相関していた.野生型マウスに由来するpiRNAの配列をくわしく調べたところ,得られた約150万の配列のうち21個がRasgrf1遺伝子のメチル化可変領域にユニークにマップされ,かつ,メチル化可変領域の片方の鎖側の配列と完全に一致していた.この21個の配列はMitoPLD変異体の精巣には存在しなかったことからpiRNAであることがわかった.piRNAは長い1本鎖からなる前駆体RNAが切断をうけ加工されてつくられるので,胎仔の精巣にはこのメチル化可変領域から転写されるRNAがあるものと推測された.
一方,最高2カ所まで配列の違いを許すと,これとは反対の鎖側の狭い領域に数百個のpiRNAがマップされることがわかった.非常に興味深いことに,その領域にはRMER4Bとよばれるレトロトランスポゾン配列が存在していた.RMER4Bはマウスゲノムに数千コピー存在するLTR型レトロトランスポゾンである.この数百個のpiRNAの由来を調べたところ,Rasgrf1遺伝子(第9染色体)とは異なる染色体(第7染色体)に存在するあるひとつのRMER4Bのコピーと配列が完全に一致すること,また,ほかには配列の完全に一致するコピーはないことがわかった.このRMER4Bのコピーを含む第7染色体の領域には多種多様なレトロトランスポゾン配列が存在しており,piRNAの一大生産地となっていた.
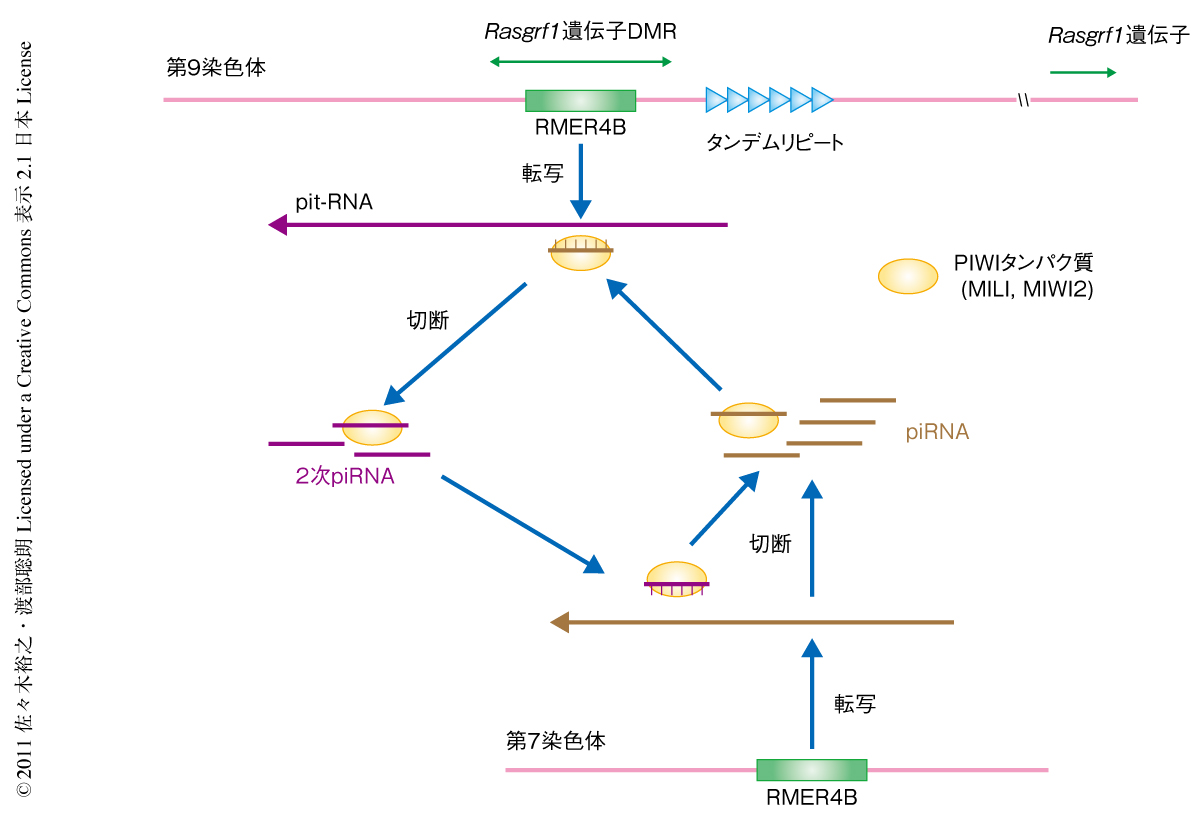
この数百個のpiRNAの配列とRasgrf1遺伝子のメチル化可変領域に由来するpiRNAの配列とを比較すると,多くが10ヌクレオチドの相補性をもっていた.これは,いわゆるピンポンサイクルとよばれるpiRNAの産生サイクルがはたらいていることを示す9,10).すなわち,ここでは第7染色体から産生されたpiRNAがPIWIタンパク質(MILIなど)に取り込まれ,これがRasgrf1遺伝子のメチル化可変領域に由来する標的RNAを認識し切断することで2次piRNAがつくられているものと考えられた(図2).
Rasgrf1遺伝子のメチル化可変領域から転写されるRNAを検出するため,野生型マウスの胎仔の精巣サンプルを用いてRT-PCR法による解析を試みた.その結果,2次piRNAと同一の鎖側に少なくとも2 kbのRNAが存在していることがわかった.このRNAはタンパク質をコードしない非コードRNAであり,胎生12.5日の始原生殖細胞と16.5日の前精原細胞には存在するが胎仔期の精巣の体細胞や生後の精巣には存在せず,卵巣およびさまざまな体細胞組織でも検出されなかった.すなわち,胎仔期の生殖細胞に特異的に存在していた.筆者らは,このRNAをpiRNA-targeted noncoding RNA(pit-RNA)と名づけた.
つぎに,5’RACE法(RACE:rapid amplification of cDNA ends)の変法を用いてpit-RNAに存在する2次piRNA産生のための切断点の検出を試みたところ,主要な切断点のひとつが確認できた.MitoPLD変異体ではこの切断によってできる断片は検出されず,切断をうけていないpit-RNAの量が約5倍に増加していた.これらの結果は,切断がpiRNAに依存的であることを強く支持した.
一方,完全長のpit-RNAを5’RACE法により解析したところ,Rasgrf1遺伝子のメチル化可変領域に隣接するタンデムリピート(直列型の反復配列)に転写開始点のあることがわかった.このタンデムリピートはRasgrf1遺伝子のインプリンティングに必須であり,これを欠失すると雄の生殖細胞におけるメチル化可変領域のDNAメチル化が起こらないことが示されている8).この欠失がpit-RNAの転写を失わせるのではないかと考え,このタンデムリピートを欠損したマウスの前精原細胞を調べたところ確かにpit-RNAが激減していた.このことは,pit-RNAがメチル化可変領域のDNAメチル化に重要なはたらきをしている可能性を示唆した.
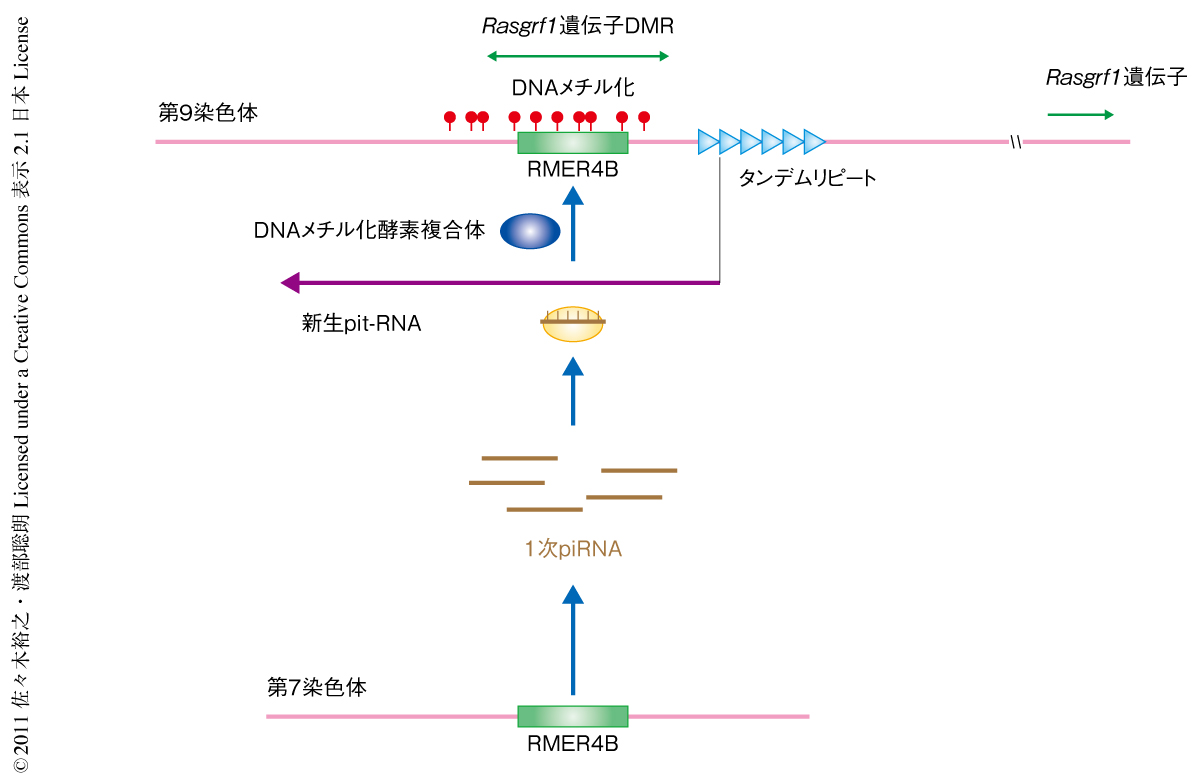
以上より,1)雄の生殖細胞におけるRasgrf1遺伝子のメチル化可変領域のDNAメチル化にはpiRNA経路が必要で,2)pit-RNAも関与しており,3)piRNAとpit-RNAとは少なくとも細胞質において直接に相互作用する(ピンポンサイクル),ことがわかった.筆者らは,3)の知見を核内まで延長し,Rasgrf1遺伝子のメチル化可変領域において転写されつつある新生pit-RNAをpiRNA-PIWI複合体が認識し,これがなんらかのシグナルとなってDNAメチル化酵素が配列に特異的にリクルートされる,というモデルを提唱した(図3).このモデルは,分裂酵母やシロイヌナズナにおいてsiRNA(small interfering RNA)が配列に特異的にエピジェネティックな遺伝子サイレンシングを起こす機構とつうじる.Rasgrf1遺伝子のインプリンティングは哺乳類のなかでマウスとラットにのみみられるが,これらの種の特徴はRasgrf1遺伝子のメチル化可変領域にRMER4Bのコピーとさきに述べたタンデムリピートが存在することである.また,このタンデムリピートの欠失がシスに隣接する領域に影響をあたえること8) は新生pit-RNAの役割と一致した.これらの観察結果は筆者らのモデルを支持していた.
マウスRasgrf1遺伝子のインプリンティングにレトロトランスポゾンの抑制機構であるpiRNA経路が必要であることがわかった.これは,進化の過程で既存の機構を借用し新たな機能を獲得した例なのであろう.Rasgrf1遺伝子は生後の成長を促進する遺伝子なので,その父由来で発現するパターン(新生仔の脳でみられる)はインプリンティングの進化に関するコンフリクト仮説と合致する11).しかしながら,単にレトロトランスポゾン抑制機構に巻き込まれた傍観者(innocent bystander)であるという可能性も否定できない.
piRNA経路はヒトやマウスのゲノムの40%をしめるレトロトランスポゾン配列のDNAメチル化に必須だが,その標的認識機構はわかっていなかった.筆者らが提唱したモデルはこの過程にあてはまるであろう.レトロトランスポゾン配列がインプリンティングをうけないのは,Rasgrf1遺伝子とは異なり,受精ののちの大規模なDNA脱メチル化と新規のDNAメチル化の過程で両親からの由来に関して差がなくなるからであろう.一方,Rasgrf1遺伝子以外のインプリンティングにpiRNA経路は不必要だったことから,DNAメチル化酵素をリクルートする別の機構があるのは明らかである.
今回の発見は不妊,流産,遺伝病の解明に役立つほか,さらに筆者らが提示したDNAメチル化機構のモデルにさらに詳細がくわわれば,疾患治療や生命工学で特定の遺伝子を制御する技術につながる可能性がある.
略歴:1987年 九州大学大学院医学系研究科 修了,国立遺伝学研究所 教授を経て,2010年より九州大学生体防御医学研究所 教授.
研究テーマ:エピゲノムの制御機構.
関心事:自分の時間をつくりだすこと.趣味は歴史と芸術と旅行および桐の花の鑑賞.登山とテニスの再開が目下の関心事.
研究室URL:http://www.bioreg.kyushu-u.ac.jp/labo/epigenome/
渡部 聡朗(Toshiaki Watanabe)
米国Yale大学School of MedicineにてPostdoctoral Fellow.
© 2011 佐々木裕之・渡部聡朗 Licensed under CC 表示 2.1 日本
(1九州大学生体防御医学研究所 エピゲノム学分野,2米国Yale大学School of Medicine,Department of Cell Biology)
email:佐々木裕之
DOI: 10.7875/first.author.2011.087
Role for piRNAs and noncoding RNA in de novo DNA methylation of the imprinted mouse Rasgrf1 locus.
Toshiaki Watanabe, Shin-ichi Tomizawa, Kohzoh Mitsuya, Yasushi Totoki, Yasuhiro Yamamoto, Satomi Kuramochi-Miyagawa, Naoko Iida, Yuko Hoki, Patrick J. Murphy, Atsushi Toyoda, Kengo Gotoh, Hitoshi Hiura, Takahiro Arima, Asao Fujiyama, Takashi Sado, Tatsuhiro Shibata, Toru Nakano, Haifan Lin, Kenji Ichiyanagi, Paul D. Soloway, Hiroyuki Sasaki
Science, 332, 848-852 (2011)
要 約
インプリンティングは雌雄の生殖細胞で確立されるDNAメチル化の違いにもとづき両親に由来する対立遺伝子のうち一方だけが発現する現象である.インプリンティングは哺乳類の正常な発生に必須で,その異常は奇形症候群や腫瘍を起こす.しかしながら,生殖細胞においてどのような機構により特定の遺伝子が選ばれDNAメチル化されているのかはほとんどわかっていない.筆者らは,レトロトランスポゾンを抑制するpiRNA経路がマウスのインプリント遺伝子Rasgrf1のDNAメチル化に必須であることをみつけた.この遺伝子のインプリンティングには雄の生殖細胞におけるDNAメチル化が必要だが,Rasgrf1遺伝子とは別の染色体に存在するレトロトランスポゾン配列から産生されるpiRNAが,Rasgrf1遺伝子の制御領域から転写される非コードRNAと相互作用していることが明らかになった.筆者らは,piRNAが非コードRNAとの相互作用を介してDNAメチル化酵素を特定の配列へとリクルートするモデルを提唱した.
はじめに
ゲノムインプリンティング(以下,インプリンティング)をうける遺伝子の近傍にはメチル化可変領域(differentially methylated region:DMR)とよばれる領域があり,この領域のDNAメチル化の状態が対立遺伝子に特異的な発現を制御している.メチル化可変領域における両親に由来するDNAメチル化の違いは雌雄の生殖細胞において確立され,これが受精卵にもち込まれて維持されたものである1,2).しかしながら,雌雄の生殖細胞においてどのような機構で特定の遺伝子が選ばれDNAメチル化されているのかはほとんどわかっていない.
胎生12.5日の始原生殖細胞においてメチル化可変領域はDNA脱メチル化状態であり(インプリントは消去されている),そののち,雄の生殖細胞系列では胎生12.5~18.5日の前精原細胞(または,ゴノサイトとよばれる)において新規のDNAメチル化が起こる(インプリントが確立される).マウスのRasgrf1遺伝子はこのように雄の生殖細胞でDNAメチル化されるインプリント遺伝子である3)(図1).一方,雄の生殖細胞の発生過程においてメチル化可変領域がDNAメチル化される時期は,IAPやLINE-1(または,L1)などのレトロトランスポゾン配列がDNAメチル化される時期と一致する3).これらレトロトランスポゾン配列のDNAメチル化には,DNAメチル化酵素Dnmt3aおよびDnmt3bと,PIWIタンパク質,および,piRNA(PIWI-interacting RNApiRNA)が必要であることがわかっている3-5).piRNAは24~30ヌクレオチド程度の生殖巣に特異的なRNAでPIWIタンパク質と結合する.PIWIタンパク質はpiRNAをガイドとして標的RNA(おもに,レトロトランスポゾン配列に由来)を認識し分解するが,レトロトランスポゾン配列のDNAメチル化にもかかわっている.しかし,その機構は不明である.DNAメチル化酵素Dnmt3aのみでDNAメチル化されるおおかたのインプリント遺伝子とは異なり,Rasgrf1遺伝子のDNAメチル化には別のDNAメチル化酵素Dnmt3bもかかわることがわかっている3,6).そこで,筆者らは,piRNAの生合成および作用経路がRasgrf1遺伝子のDNAメチル化とインプリンティングの特異性の決定にかかわるのではないかとの仮説をたてた.
1.Rasgrf1遺伝子のDNAメチル化にはpiRNA経路が必要である
生殖細胞におけるメチル化可変領域のDNAメチル化にpiRNAがかかわるかどうかを調べるため,Mili遺伝子,Miwi2遺伝子,MitoPLD遺伝子の変異マウスを解析した.Mili遺伝子とMiwi2遺伝子はPIWIファミリーのメンバーを,MitoPLD遺伝子はミトコンドリア膜に存在するホスホリパーゼをコードしており,これらの変異マウスでは精巣のpiRNAが減少し減数分裂期に精子の形成が停止する5,7).変異マウスの精原細胞においてH19遺伝子,Dlk1遺伝子-Gtl2遺伝子,Gpr1遺伝子-Zdbf2遺伝子,Rasgrf1遺伝子のメチル化可変領域(すべて,雄の生殖細胞においてDNAメチル化される)を調べたところ,Rasgrf1遺伝子のメチル化可変領域においてのみDNAメチル化レベルの低下がみられた.影響をうけたのはCpG配列が比較的密に存在する約2 kbの領域で,野生型の精原細胞では77.2~93.4%がDNAメチル化されていたのに対し,Mili変異体で23.3%,Miwi2変異体で58.6%,MitoPLD変異体では16.8%がDNAメチル化されていた.この領域のDNAメチル化がRasgrf1遺伝子のインプリンティングに必須であることはすでに示されており8),今回の結果は,piRNAがRasgrf1遺伝子のインプリンティングに関与することを遺伝学的に証明するものであった.
2.Rasgrf1遺伝子のメチル化可変領域にマップされるpiRNAの解析
つぎに,マウス胎仔の精巣から小分子RNAライブラリーを作製し超高速シークエンサーによる網羅的な配列解析を行った.Mili変異体,Miwi2変異体,MitoPLD変異体の3つのpiRNA経路の変異体ではいずれもpiRNAの減少がみられたが,その程度はさきに述べたDNAメチル化の低下とおおまかに相関していた.野生型マウスに由来するpiRNAの配列をくわしく調べたところ,得られた約150万の配列のうち21個がRasgrf1遺伝子のメチル化可変領域にユニークにマップされ,かつ,メチル化可変領域の片方の鎖側の配列と完全に一致していた.この21個の配列はMitoPLD変異体の精巣には存在しなかったことからpiRNAであることがわかった.piRNAは長い1本鎖からなる前駆体RNAが切断をうけ加工されてつくられるので,胎仔の精巣にはこのメチル化可変領域から転写されるRNAがあるものと推測された.
一方,最高2カ所まで配列の違いを許すと,これとは反対の鎖側の狭い領域に数百個のpiRNAがマップされることがわかった.非常に興味深いことに,その領域にはRMER4Bとよばれるレトロトランスポゾン配列が存在していた.RMER4Bはマウスゲノムに数千コピー存在するLTR型レトロトランスポゾンである.この数百個のpiRNAの由来を調べたところ,Rasgrf1遺伝子(第9染色体)とは異なる染色体(第7染色体)に存在するあるひとつのRMER4Bのコピーと配列が完全に一致すること,また,ほかには配列の完全に一致するコピーはないことがわかった.このRMER4Bのコピーを含む第7染色体の領域には多種多様なレトロトランスポゾン配列が存在しており,piRNAの一大生産地となっていた.
この数百個のpiRNAの配列とRasgrf1遺伝子のメチル化可変領域に由来するpiRNAの配列とを比較すると,多くが10ヌクレオチドの相補性をもっていた.これは,いわゆるピンポンサイクルとよばれるpiRNAの産生サイクルがはたらいていることを示す9,10).すなわち,ここでは第7染色体から産生されたpiRNAがPIWIタンパク質(MILIなど)に取り込まれ,これがRasgrf1遺伝子のメチル化可変領域に由来する標的RNAを認識し切断することで2次piRNAがつくられているものと考えられた(図2).
3.piRNAの標的となる非コードRNAの役割
Rasgrf1遺伝子のメチル化可変領域から転写されるRNAを検出するため,野生型マウスの胎仔の精巣サンプルを用いてRT-PCR法による解析を試みた.その結果,2次piRNAと同一の鎖側に少なくとも2 kbのRNAが存在していることがわかった.このRNAはタンパク質をコードしない非コードRNAであり,胎生12.5日の始原生殖細胞と16.5日の前精原細胞には存在するが胎仔期の精巣の体細胞や生後の精巣には存在せず,卵巣およびさまざまな体細胞組織でも検出されなかった.すなわち,胎仔期の生殖細胞に特異的に存在していた.筆者らは,このRNAをpiRNA-targeted noncoding RNA(pit-RNA)と名づけた.
つぎに,5’RACE法(RACE:rapid amplification of cDNA ends)の変法を用いてpit-RNAに存在する2次piRNA産生のための切断点の検出を試みたところ,主要な切断点のひとつが確認できた.MitoPLD変異体ではこの切断によってできる断片は検出されず,切断をうけていないpit-RNAの量が約5倍に増加していた.これらの結果は,切断がpiRNAに依存的であることを強く支持した.
一方,完全長のpit-RNAを5’RACE法により解析したところ,Rasgrf1遺伝子のメチル化可変領域に隣接するタンデムリピート(直列型の反復配列)に転写開始点のあることがわかった.このタンデムリピートはRasgrf1遺伝子のインプリンティングに必須であり,これを欠失すると雄の生殖細胞におけるメチル化可変領域のDNAメチル化が起こらないことが示されている8).この欠失がpit-RNAの転写を失わせるのではないかと考え,このタンデムリピートを欠損したマウスの前精原細胞を調べたところ確かにpit-RNAが激減していた.このことは,pit-RNAがメチル化可変領域のDNAメチル化に重要なはたらきをしている可能性を示唆した.
4.想定されるモデル
以上より,1)雄の生殖細胞におけるRasgrf1遺伝子のメチル化可変領域のDNAメチル化にはpiRNA経路が必要で,2)pit-RNAも関与しており,3)piRNAとpit-RNAとは少なくとも細胞質において直接に相互作用する(ピンポンサイクル),ことがわかった.筆者らは,3)の知見を核内まで延長し,Rasgrf1遺伝子のメチル化可変領域において転写されつつある新生pit-RNAをpiRNA-PIWI複合体が認識し,これがなんらかのシグナルとなってDNAメチル化酵素が配列に特異的にリクルートされる,というモデルを提唱した(図3).このモデルは,分裂酵母やシロイヌナズナにおいてsiRNA(small interfering RNA)が配列に特異的にエピジェネティックな遺伝子サイレンシングを起こす機構とつうじる.Rasgrf1遺伝子のインプリンティングは哺乳類のなかでマウスとラットにのみみられるが,これらの種の特徴はRasgrf1遺伝子のメチル化可変領域にRMER4Bのコピーとさきに述べたタンデムリピートが存在することである.また,このタンデムリピートの欠失がシスに隣接する領域に影響をあたえること8) は新生pit-RNAの役割と一致した.これらの観察結果は筆者らのモデルを支持していた.
おわりに
マウスRasgrf1遺伝子のインプリンティングにレトロトランスポゾンの抑制機構であるpiRNA経路が必要であることがわかった.これは,進化の過程で既存の機構を借用し新たな機能を獲得した例なのであろう.Rasgrf1遺伝子は生後の成長を促進する遺伝子なので,その父由来で発現するパターン(新生仔の脳でみられる)はインプリンティングの進化に関するコンフリクト仮説と合致する11).しかしながら,単にレトロトランスポゾン抑制機構に巻き込まれた傍観者(innocent bystander)であるという可能性も否定できない.
piRNA経路はヒトやマウスのゲノムの40%をしめるレトロトランスポゾン配列のDNAメチル化に必須だが,その標的認識機構はわかっていなかった.筆者らが提唱したモデルはこの過程にあてはまるであろう.レトロトランスポゾン配列がインプリンティングをうけないのは,Rasgrf1遺伝子とは異なり,受精ののちの大規模なDNA脱メチル化と新規のDNAメチル化の過程で両親からの由来に関して差がなくなるからであろう.一方,Rasgrf1遺伝子以外のインプリンティングにpiRNA経路は不必要だったことから,DNAメチル化酵素をリクルートする別の機構があるのは明らかである.
今回の発見は不妊,流産,遺伝病の解明に役立つほか,さらに筆者らが提示したDNAメチル化機構のモデルにさらに詳細がくわわれば,疾患治療や生命工学で特定の遺伝子を制御する技術につながる可能性がある.
文 献
- Bartolomei, M. S.: Genomic imprinting: employing and avoiding epigenetic processes. Genes Dev., 23, 2124-2133 (2009)[PubMed]
- Sasaki, H. & Matsui, Y.: Epigenetic events in mammalian germ-cell development: reprogramming and beyond. Nat. Rev. Genet., 9, 129-140 (2008)[PubMed]
- Kato, Y., Kaneda, M., Hata, K. et al.: Role of the Dnmt3 family in de novo methylation of imprinted and repetitive sequences during male germ cell development in the mouse. Hum. Mol. Genet., 16, 2272-2280 (2007)[PubMed]
- Aravin, A. A., Sachidanandam, R., Bourc'his, D. et al.: A piRNA pathway primed by individual transposons is linked to de novo DNA methylation in mice. Mol. Cell, 31, 785-799 (2008)[PubMed]
- Kuramochi-Miyagawa, S., Watanabe, T., Gotoh, K. et al.: DNA methylation of retrotransposon genes is regulated by Piwi family members MILI and MIWI2 in murine fetal testes. Genes Dev., 22, 908-917 (2008)[PubMed]
- Hirasawa, R., Chiba, H., Kaneda, M. et al.: Maternal and zygotic Dnmt1 are necessary and sufficient for the maintenance of DNA methylation imprints during preimplantation development. Genes Dev., 22, 1607-1616 (2008)[PubMed]
- Watanabe, T., Chuma, S., Yamamoto, Y. et al.: MITOPLD is a mitochondrial protein essential for nuage formation and piRNA biogenesis in the mouse germline. Dev. Cell, 20, 364-375 (2011)[PubMed] [新着論文レビュー]
- Yoon, B. J., Herman, H., Sikora, A. et al.: Regulation of DNA methylation of Rasgrf1. Nat. Genet., 30, 92-96 (2002)[PubMed]
- Aravin, A. A., Hannon, G. J. & Brennecke, J.: The Piwi-piRNA pathway provides an adaptive defense in the transposon arms race. Science, 318, 761-764 (2007)[PubMed]
- Brennecke, J., Aravin, A. A., Stark, A. et al.: Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell, 128, 1089-1103 (2007)[PubMed]
- Drake, N. M., Park, Y. J., Shirali, A. S. et al.: Imprint switch mutations at Rasgrf1 support conflict hypothesis of imprinting and define a growth control mechanism upstream of IGF1. Mamm. Genome, 20, 654-663 (2009)[PubMed]
著者プロフィール
略歴:1987年 九州大学大学院医学系研究科 修了,国立遺伝学研究所 教授を経て,2010年より九州大学生体防御医学研究所 教授.
研究テーマ:エピゲノムの制御機構.
関心事:自分の時間をつくりだすこと.趣味は歴史と芸術と旅行および桐の花の鑑賞.登山とテニスの再開が目下の関心事.
研究室URL:http://www.bioreg.kyushu-u.ac.jp/labo/epigenome/
渡部 聡朗(Toshiaki Watanabe)
米国Yale大学School of MedicineにてPostdoctoral Fellow.
© 2011 佐々木裕之・渡部聡朗 Licensed under CC 表示 2.1 日本