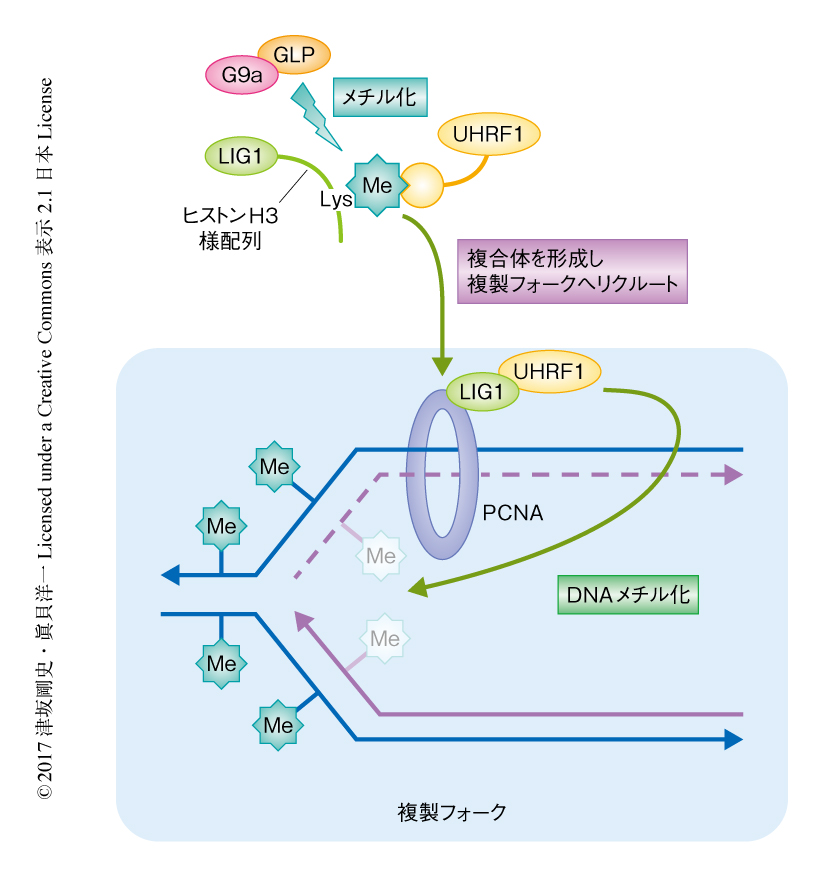
DNA複製タンパク質LIG1のヒストンH3様配列のメチル化はDNA複製とDNAメチル化の継承とをつなぐ
津坂剛史・眞貝洋一
(理化学研究所 眞貝細胞記憶研究室)
email:津坂剛史,眞貝洋一
DOI: 10.7875/first.author.2017.092
Methylation of DNA ligase 1 by G9a/GLP recruits UHRF1 to replicating DNA and regulates DNA methylation.
Laure Ferry, Alexandra Fournier, Takeshi Tsusaka, Guillaume Adelmant, Tadahiro Shimazu, Shohei Matano, Olivier Kirsh, Rachel Amouroux, Naoshi Dohmae, Takehiro Suzuki, Guillaume J. Filion, Wen Deng, Maud de Dieuleveult, Lauriane Fritsch, Srikanth Kudithipudi, Albert Jeltsch, Heinrich Leonhardt, Petra Hajkova, Jarrod A. Marto, Kyohei Arita, Yoichi Shinkai, Pierre-Antoine Defossez
Molecular Cell, 67, 550-565.e5 (2017)
ヒストンのメチル化やDNAのメチル化はDNAの塩基配列によらないエピジェネティックな遺伝子発現の制御の中枢を担うと考えられている.ヒストン以外のタンパク質もメチル化されることが明らかにされつつあるが,それらの機能の多くはいまだベールにつつまれている.この研究において,筆者らは,DNA複製タンパク質であるLIG1がヒストンH3様の配列をもち,G9a-GLP複合体によりメチル化されることを見い出した.さらに,メチル化したLIG1はUHRF1により認識されることがわかった.UHRF1はDNAメチル化酵素DNMT1を介しDNA複製にともない新生鎖のDNAメチル化をひき起こし,DNAメチル化のパターンを継承させる役割をもつ.このUHRF1の機能におけるメチル化したLIG1の関与について調べたところ,LIG1はそのメチル化に依存してUHRF1を複製フォークへとリクルートすることによりDNAメチル化を制御することが明らかにされた.これらの結果から,LIG1のヒストンH3様配列のメチル化はDNA複製とそれにともなうDNAメチル化の継承とをつなぐ機構であることが示唆された.
多くの細胞は基本的にゲノムにコードされた単一の遺伝情報からなりたち,エピジェネティックな制御機構により,それぞれに固有の遺伝子発現のパターンを形成する.ヒストンのメチル化やDNAのメチル化は細胞に固有のパターンを示し,遺伝子の発現を規定すると考えられている.ヒストンのメチル化およびDNAメチル化のパターンは細胞分裂をへても安定的に維持されるが,その継承の機構については多くの謎が残されている.なかでも,DNA複製の際にどのように“古い”情報を読み取り“新しい”媒体,すなわち,ヒストンおよびDNAへと移行するのかは肝要かつ難解な疑問である.
ヒストンのメチル化に比べるとDNAメチル化のパターンの継承については理解が進んでおり,中心的な役割を担うタンパク質としてUHRF1およびDNMT1が見い出されている1-3).DNA複製の直後において,鋳型DNA鎖がメチル化されていたとしても新生鎖DNAはメチル化されておらず,2本鎖DNAはヘミメチル化状態にあると考えられている.UHRF1はこのヘミメチル化状態を認識し,DNAメチル化酵素DNMT1をリクルートすることにより新生鎖のDNAメチル化をひき起こす2,3).しかしながら,UHRF1がどのようにDNA複製の場にリクルートされるのかなど,依然として不明な点も多い4).
筆者らの研究グループは,ヒストンメチル化酵素であるG9a-GLP複合体のヒストン以外の新規の基質を探索するため,メチル化プローブとプロテオミクスとを組み合わせた網羅的な解析をしたところ5),岡崎フラグメントを連結するDNA複製タンパク質であるLIG1が同定された.また,UHRF1の機能を詳細に知るため,質量分析法によりその結合タンパク質を探索したところ,同じくLIG1が同定された.これらの結果から,ひとつのストーリーがうかびあがった.
G9a-GLP複合体はヒストンH3のLys9をメチル化するヒストンメチル化酵素として同定されたが,いくつかのヒストン以外のタンパク質もメチル化する6).G9a-GLP複合体の新規の基質を探索したところLIG1が同定された.LIG1はC末端領域に酵素活性ドメインをもち,N末端にDNA複製に中心的な役割をもつPCNAとの結合ドメインをもつ7).G9a-GLP複合体はヒストンH3のLys9をメチル化することからその周辺のアミノ酸配列を精査したところ,LIG1のN末端側の領域にヒストンH3とよく似た配列が見い出された.このヒストンH3様配列はマウス,ラット,アフリカツメガエルなどにおいて保存されていたが,ショウジョウバエには保存されていなかった.
LIG1のもつヒストンH3様配列がG9a-GLP複合体によりメチル化されるかどうかin vitroにおいて調べたところ,G9aあるいはGLPのどちらの存在のもとでもLIG1へのメチル基の取り込みがみられた.LIG1が細胞において実際にメチル化されているかどうか調べるため,ヒトのHEK293細胞あるいはマウスのES細胞からLIG1を免疫沈降し質量分析法により解析した結果,LIG1の多くはメチル化されていた.また,LIG1のメチル化は,G9a欠損細胞においては予想に反して亢進し,GLP欠損細胞においては減少,G9a GLP二重欠損細胞においては完全に消失した.一方,ヒストンH3のLys9のメチル化はこれらいずれの欠損細胞においても同じ程度に大きく減少しており,LIG1のメチル化の状態とは一致しなかった.これらの結果から,LIG1のメチル化におけるG9あるいはGLPの寄与率はヒストンH3に対するものとは異なり,GLPはG9aよりLIG1に対する酵素活性の強いことが示唆された.
UHRF1はTTDドメインを介してメチル化したヒストンH3のLys9と結合する8,9).UHRF1の結合タンパク質を質量分析法により網羅的に探索したところLIG1が同定された.変異体を用いた解析により,UHRF1はTTDドメインを介してLIG1と結合することが明らかにされた.また,ヒストンH3様配列のLysをArgに置換したLIG1変異体はUHRF1と結合せず,さらに,G9a GLP二重欠損細胞においてLIG1とUHRF1との結合はみられなかった.以上の結果から,UHRF1とLIG1との結合にはG9a-GLP複合体によるLIG1のヒストンH3様配列のLysのメチル化およびUHRF1のTTDドメインが重要であることが強く示唆された.精製したタンパク質を用いたゲルシフトアッセイにより,UHRF1のTTDドメインとLIG1のヒストンH3様配列との結合が直接的,かつ,LIG1のメチル化に依存的であることが確認された.さらに,等温滴定型カロリメトリーおよび蛍光偏光法により,UHRF1はメチル化したヒストンH3のLys9よりもメチル化したLIG1と強く結合することが示唆された.以上により,UHRF1はTTDドメインを介し直接的にメチル化したLIG1と結合することが明らかにされた.
LIG1はPCNAと結合することにより複製フォークに局在することから7),LIG1がUHRF1と結合しこれを複製フォークへとリクルートすることが考えられた.この仮説を証明するため,チミジンのアナログであるEdUを用いて複製フォークを可視化し10),免疫染色と組み合わせてマウスのES細胞におけるLIG1およびUHRF1の局在をみたところ,どちらも複製フォークとの共局在が観察された.一方で,G9a欠損細胞においてはUHRF1の複製フォークへの局在はわずかに減少し,GLP欠損細胞ではさらに大きく減少,G9a GLP二重欠損細胞においてはほとんど共局在がみられなかった.また,これらいずれの欠損細胞においてもLIG1の局在に変化はなかった.UHRF1はDNAメチル化酵素であるDNMT1を複製フォークにリクルートすることにより新生鎖のDNAメチル化をひき起こす2,3).そこで,G9a欠損細胞,GLP欠損細胞,G9a GLP二重欠損細胞においてDNMT1の局在を調べたところ,UHRF1と同様の結果が得られた.以上より,G9a-GLP複合体はUHRF1およびそれにつづくDNMT1の複製フォークへのリクルートを制御することが明らかにされた.
G9a-GLP複合体によるUHRF1の局在の制御にはヒストンH3のLys9のメチル化,あるいは,LIG1のメチル化が関与すると考えられた.CRISPR-Cas9を用いたゲノム編集法により,LIG1のヒストンH3様配列を欠損させた細胞を樹立した.この細胞は酵素活性ドメインを保持するがヒストンH3様配列を欠失したLIG1を発現し,このLIG1変異体は正常に複製フォークに局在したことから,LIG1の酵素活性に影響をおよぼすことなくヒストンH3様配列の重要性のみを調べることができると考えられた.このLIG1のヒストンH3様配列を欠損させた細胞においてUHRF1およびDNMT1の局在を調べたところ,複製フォークへのリクルートが大きく低下していた.さらに,ヒストンH3様配列のLysの重要性を調べるため,このLIG1のヒストンH3様配列を欠損させた細胞に野生型のLIG1あるいはヒストンH3様配列のLysをArgに置換したLIG1変異体を発現させたところ,野生型LIG1の発現によりUHRF1およびDNMT1の複製フォークへの局在が部分的に回復した一方,LIG1変異体の発現により回復はみられなかった.以上の結果から,G9a-GLP複合体によるLIG1のヒストンH3様配列のLysのメチル化がUHRF1の複製フォークへの局在に重要であることが強く示唆された.
以上の結果から,LIG1のヒストンH3様配列のメチル化がDNA複製にともなうUHRF1に依存的なDNAメチル化の継承に関与することが示唆された.そこで,LIG1のヒストンH3様配列を欠損させた細胞においてDNAメチル化の量を質量分析法およびLUMA(luminometric methylation assay)法により解析したところ,野生型の細胞に比べ低下していた.さらに,ゲノムのどのような部位においてDNAメチル化が低下しているのかを調べるため,RRBS(reduced representation of bisulfite sequence)法によりLIG1のヒストンH3様配列を欠損させた細胞およびUHRF1欠損細胞を解析したところ,類似した領域においてDNAメチル化が低下した状態にあり,共通する部位が多く存在した.このゲノムの全体にわたるDNAメチルの低下は,複製にともなう新生鎖のDNAメチル化が効率的に起こらないために生じたと考えられた.
ヒストンH3様配列のLysの重要性について調べるため,LIG1のヒストンH3様配列を欠損させた細胞に野生型のLIG1あるいはヒストンH3様配列のLysをArgに置換したLIG1変異体を発現させたところ,野生型LIG1の発現によりDNAメチル化は部分的に回復したが,LIG1変異体の発現により回復はみられなかった.さらに,G9a欠損細胞,GLP欠損細胞,G9a GLP二重欠損細胞においてDNAメチル化の量をLUMA法により調べたところ,野生型の細胞に比べ,G9a欠損細胞,GLP欠損細胞,二重欠損細胞の順にDNAメチル化が低下していた.以上の結果から,G9a-GLP複合体によるLIG1のヒストンH3様配列のメチル化はUHRF1の複製フォークへのリクルートを介してDNAメチル化を制御すると考えられた(図1).
LIG1を欠損したES細胞を樹立し解析したところ,UHRF1およびDNMT1の複製フォークへの局在の低下を示した一方,DNAメチル化の低下は示さなかった.これは,LIG1が欠損したことによりなんらかのバックアップ機構がはたらき,DNAメチル化が維持あるいは回復されたためと考えられた.LIG1が機能できなくなるとLIG3がその機能を部分的に補完することから11),LIG1欠損細胞においてLIG3がDNAメチル化の維持あるいは回復を担うのかもしれない.この場合,LIG3はヒストンH3様配列をもたないことから,LIG1とは異なった結合様式によりUHRF1と結合する,あるいは,UHRF1に依存しないDNAメチル化の維持あるいは回復の起こる可能性が考えられた.LIG1欠損細胞にヒストンH3様配列のLysをArgに置換したLIG1変異体を発現させたところDNAメチル化の低下が観察され,一方で,野生型のLIG1を発現させてもDNAメチル化の状態は変化しなかった.この結果からも,LIG1のヒストンH3様配列のメチル化がDNAメチル化を制御するというモデルが支持された.
LIG1はラギング鎖において岡崎フラグメントを結合する酵素として見い出されたが,この研究により,DNA複製にともなうDNAメチル化の継承においてはたらくという新たな一面が明らかにされた.では,はたしてLIG1はラギング鎖においてのみDNAメチル化に貢献するのであろうか? あるいは,リーディング鎖とラギング鎖のどちらにおいてもDNAメチル化に貢献するのだろうか? 現在のところ,この疑問に対する答えはもちあわせないが,リーディング鎖とラギング鎖とを区別して解析することにより結論が得られるのではないかと思っている.
また,G9a-GLP複合体によるLIG1のメチル化のような,ヒストン以外のタンパク質のメチル化については徐々に明らかにされつつあるが,ここで疑問が生じる.“ヒストンメチル化酵素”として同定されたタンパク質は,はたしてヒストンのメチル化を介して機能するのか? あるいは,G9aがKDM1Aのメチル化を介して転写を制御するように12),ヒストン以外のタンパク質のメチル化を介して機能するのだろうか? この研究においては,G9a欠損細胞およびGLP欠損細胞においてLIG1のメチル化に対する影響とヒストンのメチル化に対する影響とが異なったため両者は区別された.また,LIG1のヒストンH3様配列を欠損させた細胞が樹立されたことからLIG1のメチル化を介すると結論づけられた.CRISPR-Cas9系の応用によりゲノム編集のみならずヒストンの修飾を操作することが可能になりつつある昨今,因果関係をきちんと証明することは意義深いと思える.
略歴:京都大学大学院医学研究科博士後期課程 在学中.
研究テーマ:タンパク質メチル化酵素の新規の基質の同定およびその生物学的な重要性.
抱負:これはおもしろい! と自負できる研究をする.
眞貝 洋一(Yoichi Shinkai)
理化学研究所 主任研究員.
研究室URL:http://shinkai.riken.jp/index.html
© 2017 津坂剛史・眞貝洋一 Licensed under CC 表示 2.1 日本
(理化学研究所 眞貝細胞記憶研究室)
email:津坂剛史,眞貝洋一
DOI: 10.7875/first.author.2017.092
Methylation of DNA ligase 1 by G9a/GLP recruits UHRF1 to replicating DNA and regulates DNA methylation.
Laure Ferry, Alexandra Fournier, Takeshi Tsusaka, Guillaume Adelmant, Tadahiro Shimazu, Shohei Matano, Olivier Kirsh, Rachel Amouroux, Naoshi Dohmae, Takehiro Suzuki, Guillaume J. Filion, Wen Deng, Maud de Dieuleveult, Lauriane Fritsch, Srikanth Kudithipudi, Albert Jeltsch, Heinrich Leonhardt, Petra Hajkova, Jarrod A. Marto, Kyohei Arita, Yoichi Shinkai, Pierre-Antoine Defossez
Molecular Cell, 67, 550-565.e5 (2017)
要 約
ヒストンのメチル化やDNAのメチル化はDNAの塩基配列によらないエピジェネティックな遺伝子発現の制御の中枢を担うと考えられている.ヒストン以外のタンパク質もメチル化されることが明らかにされつつあるが,それらの機能の多くはいまだベールにつつまれている.この研究において,筆者らは,DNA複製タンパク質であるLIG1がヒストンH3様の配列をもち,G9a-GLP複合体によりメチル化されることを見い出した.さらに,メチル化したLIG1はUHRF1により認識されることがわかった.UHRF1はDNAメチル化酵素DNMT1を介しDNA複製にともない新生鎖のDNAメチル化をひき起こし,DNAメチル化のパターンを継承させる役割をもつ.このUHRF1の機能におけるメチル化したLIG1の関与について調べたところ,LIG1はそのメチル化に依存してUHRF1を複製フォークへとリクルートすることによりDNAメチル化を制御することが明らかにされた.これらの結果から,LIG1のヒストンH3様配列のメチル化はDNA複製とそれにともなうDNAメチル化の継承とをつなぐ機構であることが示唆された.
はじめに
多くの細胞は基本的にゲノムにコードされた単一の遺伝情報からなりたち,エピジェネティックな制御機構により,それぞれに固有の遺伝子発現のパターンを形成する.ヒストンのメチル化やDNAのメチル化は細胞に固有のパターンを示し,遺伝子の発現を規定すると考えられている.ヒストンのメチル化およびDNAメチル化のパターンは細胞分裂をへても安定的に維持されるが,その継承の機構については多くの謎が残されている.なかでも,DNA複製の際にどのように“古い”情報を読み取り“新しい”媒体,すなわち,ヒストンおよびDNAへと移行するのかは肝要かつ難解な疑問である.
ヒストンのメチル化に比べるとDNAメチル化のパターンの継承については理解が進んでおり,中心的な役割を担うタンパク質としてUHRF1およびDNMT1が見い出されている1-3).DNA複製の直後において,鋳型DNA鎖がメチル化されていたとしても新生鎖DNAはメチル化されておらず,2本鎖DNAはヘミメチル化状態にあると考えられている.UHRF1はこのヘミメチル化状態を認識し,DNAメチル化酵素DNMT1をリクルートすることにより新生鎖のDNAメチル化をひき起こす2,3).しかしながら,UHRF1がどのようにDNA複製の場にリクルートされるのかなど,依然として不明な点も多い4).
筆者らの研究グループは,ヒストンメチル化酵素であるG9a-GLP複合体のヒストン以外の新規の基質を探索するため,メチル化プローブとプロテオミクスとを組み合わせた網羅的な解析をしたところ5),岡崎フラグメントを連結するDNA複製タンパク質であるLIG1が同定された.また,UHRF1の機能を詳細に知るため,質量分析法によりその結合タンパク質を探索したところ,同じくLIG1が同定された.これらの結果から,ひとつのストーリーがうかびあがった.
1.LIG1はヒストンH3様配列をもちG9a-GLP複合体によりメチル化される
G9a-GLP複合体はヒストンH3のLys9をメチル化するヒストンメチル化酵素として同定されたが,いくつかのヒストン以外のタンパク質もメチル化する6).G9a-GLP複合体の新規の基質を探索したところLIG1が同定された.LIG1はC末端領域に酵素活性ドメインをもち,N末端にDNA複製に中心的な役割をもつPCNAとの結合ドメインをもつ7).G9a-GLP複合体はヒストンH3のLys9をメチル化することからその周辺のアミノ酸配列を精査したところ,LIG1のN末端側の領域にヒストンH3とよく似た配列が見い出された.このヒストンH3様配列はマウス,ラット,アフリカツメガエルなどにおいて保存されていたが,ショウジョウバエには保存されていなかった.
LIG1のもつヒストンH3様配列がG9a-GLP複合体によりメチル化されるかどうかin vitroにおいて調べたところ,G9aあるいはGLPのどちらの存在のもとでもLIG1へのメチル基の取り込みがみられた.LIG1が細胞において実際にメチル化されているかどうか調べるため,ヒトのHEK293細胞あるいはマウスのES細胞からLIG1を免疫沈降し質量分析法により解析した結果,LIG1の多くはメチル化されていた.また,LIG1のメチル化は,G9a欠損細胞においては予想に反して亢進し,GLP欠損細胞においては減少,G9a GLP二重欠損細胞においては完全に消失した.一方,ヒストンH3のLys9のメチル化はこれらいずれの欠損細胞においても同じ程度に大きく減少しており,LIG1のメチル化の状態とは一致しなかった.これらの結果から,LIG1のメチル化におけるG9あるいはGLPの寄与率はヒストンH3に対するものとは異なり,GLPはG9aよりLIG1に対する酵素活性の強いことが示唆された.
2.LIG1はそのメチル化に依存してUHRF1と複合体を形成する
UHRF1はTTDドメインを介してメチル化したヒストンH3のLys9と結合する8,9).UHRF1の結合タンパク質を質量分析法により網羅的に探索したところLIG1が同定された.変異体を用いた解析により,UHRF1はTTDドメインを介してLIG1と結合することが明らかにされた.また,ヒストンH3様配列のLysをArgに置換したLIG1変異体はUHRF1と結合せず,さらに,G9a GLP二重欠損細胞においてLIG1とUHRF1との結合はみられなかった.以上の結果から,UHRF1とLIG1との結合にはG9a-GLP複合体によるLIG1のヒストンH3様配列のLysのメチル化およびUHRF1のTTDドメインが重要であることが強く示唆された.精製したタンパク質を用いたゲルシフトアッセイにより,UHRF1のTTDドメインとLIG1のヒストンH3様配列との結合が直接的,かつ,LIG1のメチル化に依存的であることが確認された.さらに,等温滴定型カロリメトリーおよび蛍光偏光法により,UHRF1はメチル化したヒストンH3のLys9よりもメチル化したLIG1と強く結合することが示唆された.以上により,UHRF1はTTDドメインを介し直接的にメチル化したLIG1と結合することが明らかにされた.
3.G9a-GLP複合体によるLIG1のメチル化はUHRF1の複製フォークへの局在に寄与する
LIG1はPCNAと結合することにより複製フォークに局在することから7),LIG1がUHRF1と結合しこれを複製フォークへとリクルートすることが考えられた.この仮説を証明するため,チミジンのアナログであるEdUを用いて複製フォークを可視化し10),免疫染色と組み合わせてマウスのES細胞におけるLIG1およびUHRF1の局在をみたところ,どちらも複製フォークとの共局在が観察された.一方で,G9a欠損細胞においてはUHRF1の複製フォークへの局在はわずかに減少し,GLP欠損細胞ではさらに大きく減少,G9a GLP二重欠損細胞においてはほとんど共局在がみられなかった.また,これらいずれの欠損細胞においてもLIG1の局在に変化はなかった.UHRF1はDNAメチル化酵素であるDNMT1を複製フォークにリクルートすることにより新生鎖のDNAメチル化をひき起こす2,3).そこで,G9a欠損細胞,GLP欠損細胞,G9a GLP二重欠損細胞においてDNMT1の局在を調べたところ,UHRF1と同様の結果が得られた.以上より,G9a-GLP複合体はUHRF1およびそれにつづくDNMT1の複製フォークへのリクルートを制御することが明らかにされた.
G9a-GLP複合体によるUHRF1の局在の制御にはヒストンH3のLys9のメチル化,あるいは,LIG1のメチル化が関与すると考えられた.CRISPR-Cas9を用いたゲノム編集法により,LIG1のヒストンH3様配列を欠損させた細胞を樹立した.この細胞は酵素活性ドメインを保持するがヒストンH3様配列を欠失したLIG1を発現し,このLIG1変異体は正常に複製フォークに局在したことから,LIG1の酵素活性に影響をおよぼすことなくヒストンH3様配列の重要性のみを調べることができると考えられた.このLIG1のヒストンH3様配列を欠損させた細胞においてUHRF1およびDNMT1の局在を調べたところ,複製フォークへのリクルートが大きく低下していた.さらに,ヒストンH3様配列のLysの重要性を調べるため,このLIG1のヒストンH3様配列を欠損させた細胞に野生型のLIG1あるいはヒストンH3様配列のLysをArgに置換したLIG1変異体を発現させたところ,野生型LIG1の発現によりUHRF1およびDNMT1の複製フォークへの局在が部分的に回復した一方,LIG1変異体の発現により回復はみられなかった.以上の結果から,G9a-GLP複合体によるLIG1のヒストンH3様配列のLysのメチル化がUHRF1の複製フォークへの局在に重要であることが強く示唆された.
4.G9a-GLP複合体によるLIG1のメチル化はDNAメチル化を制御する
以上の結果から,LIG1のヒストンH3様配列のメチル化がDNA複製にともなうUHRF1に依存的なDNAメチル化の継承に関与することが示唆された.そこで,LIG1のヒストンH3様配列を欠損させた細胞においてDNAメチル化の量を質量分析法およびLUMA(luminometric methylation assay)法により解析したところ,野生型の細胞に比べ低下していた.さらに,ゲノムのどのような部位においてDNAメチル化が低下しているのかを調べるため,RRBS(reduced representation of bisulfite sequence)法によりLIG1のヒストンH3様配列を欠損させた細胞およびUHRF1欠損細胞を解析したところ,類似した領域においてDNAメチル化が低下した状態にあり,共通する部位が多く存在した.このゲノムの全体にわたるDNAメチルの低下は,複製にともなう新生鎖のDNAメチル化が効率的に起こらないために生じたと考えられた.
ヒストンH3様配列のLysの重要性について調べるため,LIG1のヒストンH3様配列を欠損させた細胞に野生型のLIG1あるいはヒストンH3様配列のLysをArgに置換したLIG1変異体を発現させたところ,野生型LIG1の発現によりDNAメチル化は部分的に回復したが,LIG1変異体の発現により回復はみられなかった.さらに,G9a欠損細胞,GLP欠損細胞,G9a GLP二重欠損細胞においてDNAメチル化の量をLUMA法により調べたところ,野生型の細胞に比べ,G9a欠損細胞,GLP欠損細胞,二重欠損細胞の順にDNAメチル化が低下していた.以上の結果から,G9a-GLP複合体によるLIG1のヒストンH3様配列のメチル化はUHRF1の複製フォークへのリクルートを介してDNAメチル化を制御すると考えられた(図1).
LIG1を欠損したES細胞を樹立し解析したところ,UHRF1およびDNMT1の複製フォークへの局在の低下を示した一方,DNAメチル化の低下は示さなかった.これは,LIG1が欠損したことによりなんらかのバックアップ機構がはたらき,DNAメチル化が維持あるいは回復されたためと考えられた.LIG1が機能できなくなるとLIG3がその機能を部分的に補完することから11),LIG1欠損細胞においてLIG3がDNAメチル化の維持あるいは回復を担うのかもしれない.この場合,LIG3はヒストンH3様配列をもたないことから,LIG1とは異なった結合様式によりUHRF1と結合する,あるいは,UHRF1に依存しないDNAメチル化の維持あるいは回復の起こる可能性が考えられた.LIG1欠損細胞にヒストンH3様配列のLysをArgに置換したLIG1変異体を発現させたところDNAメチル化の低下が観察され,一方で,野生型のLIG1を発現させてもDNAメチル化の状態は変化しなかった.この結果からも,LIG1のヒストンH3様配列のメチル化がDNAメチル化を制御するというモデルが支持された.
おわりに
LIG1はラギング鎖において岡崎フラグメントを結合する酵素として見い出されたが,この研究により,DNA複製にともなうDNAメチル化の継承においてはたらくという新たな一面が明らかにされた.では,はたしてLIG1はラギング鎖においてのみDNAメチル化に貢献するのであろうか? あるいは,リーディング鎖とラギング鎖のどちらにおいてもDNAメチル化に貢献するのだろうか? 現在のところ,この疑問に対する答えはもちあわせないが,リーディング鎖とラギング鎖とを区別して解析することにより結論が得られるのではないかと思っている.
また,G9a-GLP複合体によるLIG1のメチル化のような,ヒストン以外のタンパク質のメチル化については徐々に明らかにされつつあるが,ここで疑問が生じる.“ヒストンメチル化酵素”として同定されたタンパク質は,はたしてヒストンのメチル化を介して機能するのか? あるいは,G9aがKDM1Aのメチル化を介して転写を制御するように12),ヒストン以外のタンパク質のメチル化を介して機能するのだろうか? この研究においては,G9a欠損細胞およびGLP欠損細胞においてLIG1のメチル化に対する影響とヒストンのメチル化に対する影響とが異なったため両者は区別された.また,LIG1のヒストンH3様配列を欠損させた細胞が樹立されたことからLIG1のメチル化を介すると結論づけられた.CRISPR-Cas9系の応用によりゲノム編集のみならずヒストンの修飾を操作することが可能になりつつある昨今,因果関係をきちんと証明することは意義深いと思える.
文 献
- Foll, M. G. & Bestor, T. H.: Eukaryotic cytosine methyltransferases. Annu. Rev. Biochem., 74, 481-514 (2005)[PubMed]
- Sharif, J., Muto, M., Takebayashi, S. et al.: The SRA protein Np95 mediates epigenetic inheritance by recruiting Dnmt1 to methylated DNA. Nature, 450, 908-912 (2007)[PubMed]
- Bostick, M., Kim, J. K., Esteve, P. O. et al.: UHRF1 plays a role in maintaining DNA methylation in mammalian cells. Science, 317, 1760-1764 (2007)[PubMed]
- Jeltsch, A. & Jurkowska, R. Z.: New concepts in DNA methylation. Trends Biochem. Sci., 39, 310-318 (2014)[PubMed]
- Shimazu, T., Barjau, J., Sohtome, Y. et al.: Selenium-based S-adenosylmethionine analog reveals the mammalian seven-beta-strand methyltransferase METTL10 to be an EF1A1 lysine methyltransferase. PLoS One, 9, e105394 (2014)[PubMed]
- Shinkai, Y. & Tachibana, M.: H3K9 methyltransferase G9a and the related molecule GLP. Genes Dev., 25, 781-788 (2011)[PubMed]
- Howes, T. R. & Tomkinson, A. E.: DNA ligase I, the replicative DNA ligase. Subcell. Biochem., 62, 327-341 (2012)[PubMed]
- Rottach, A., Frauer, C., Pichler, G. et al.: The multi-domain protein Np95 connects DNA methylation and histone modification. Nucleic Acids Res., 38, 1796-1804 (2010)[PubMed]
- Nady, N., Lemak, A., Walker, J. R. et al.: Recognition of multivalent histone states associated with heterochromatin by UHRF1 protein. J. Biol. Chem., 286, 24300-24311 (2011)[PubMed]
- Salic, A. & Mitchison, T. J.: A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc. Natl. Acad. Sci. USA, 105, 2415-2420 (2008)[PubMed]
- Le Chalony, C., Hoffschir, F., Gauthier, L. R. et al.: Partial complementation of a DNA ligase I deficiency by DNA ligase III and its impact on cell survival and telomere stability in mammalian cells. Cell. Mol. Life Sci., 69, 2933-2949 (2012)[PubMed]
- Metzger, E., Willmann, D., McMillan, J. et al.: Assembly of methylated KDM1A and CHD1 drives androgen receptor-dependent transcription and translocation. Nat. Struct. Mol. Biol., 23, 132-139 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:京都大学大学院医学研究科博士後期課程 在学中.
研究テーマ:タンパク質メチル化酵素の新規の基質の同定およびその生物学的な重要性.
抱負:これはおもしろい! と自負できる研究をする.
眞貝 洋一(Yoichi Shinkai)
理化学研究所 主任研究員.
研究室URL:http://shinkai.riken.jp/index.html
© 2017 津坂剛史・眞貝洋一 Licensed under CC 表示 2.1 日本