変則暗号の成立に寄与する新規のtRNA修飾の発見
長尾翌手可・鈴木 勉
(東京大学大学院工学系研究科 化学生命工学専攻)
email:長尾翌手可,鈴木 勉
DOI: 10.7875/first.author.2017.085
Hydroxylation of a conserved tRNA modification establishes non-universal genetic code in echinoderm mitochondria.
Asuteka Nagao, Mitsuhiro Ohara, Kenjyo Miyauchi, Shin-ichi Yokobori, Akihiko Yamagishi, Kimitsuna Watanabe, Tsutomu Suzuki
Nature Structural & Molecular Biology, 24, 778-782 (2017)
動物のミトコンドリアの翻訳系においては,しばしば普遍暗号から逸脱した変則暗号が用いられる.進化の過程において変則暗号が成立する際にはtRNAにおける修飾塩基の獲得が重要な役割を担う.ウニやヒトデなど棘皮動物のミトコンドリアにおいては,通常はLysを指定するAAAコドンがAsnを指定するよう暗号変化している.過去の研究において,tRNAAsnはアンチコドンの2字目にシュードウリジンを獲得することによりAAAコドンを解読するようになることが明らかにされているが,tRNALysがどのような機構によりAAAコドンの誤認識をふせいでいるかについてはわかっていない.この研究において,筆者らは,キタムラサキウニのミトコンドリアからtRNALysを単離精製し高感度な質量分析法により解析したところ,アンチコドンの3’側の隣接部位に新規の修飾塩基であるヒドロキシ-N6-スレオニルカルバモイルアデノシンを見い出した.リボソームのAサイトにおけるtRNAのコドン認識能を評価したところ,この修飾塩基はAAAコドンへの結合能を抑制する役割のあることが判明した.以上の結果から,棘皮動物のミトコンドリアにおいてtRNALysが新たな修飾塩基を獲得することによりAAAコドンの暗号変化に寄与したと考えられた.
1960年代に遺伝暗号表が決定されて以来,すべての生物は共通の遺伝暗号,すなわち,普遍暗号を用いてDNAからタンパク質へ遺伝情報を変換すると信じられていた.このことは生物一元説の大きな根拠にもなっている.しかし,1979年にヒトのミトコンドリアのDNA塩基配列が決定され1),AUAコドンがMet,UGAコドンがTrp,AGAコドンおよびAGGコドンが終止を指定することが判明し,ミトコンドリアの翻訳系において普遍暗号から逸脱した変則暗号の用いられていることが明らかにされた.それ以降,さまざまな動物のミトコンドリアにおいてあいついで変則暗号が発見され2),核のゲノムにおいてもマイコプラズマ,Candida属の酵母,原生動物などにおいて変則暗号がみつかり,遺伝暗号は普遍ではなく変化しうるものであると認識されるようになった3).
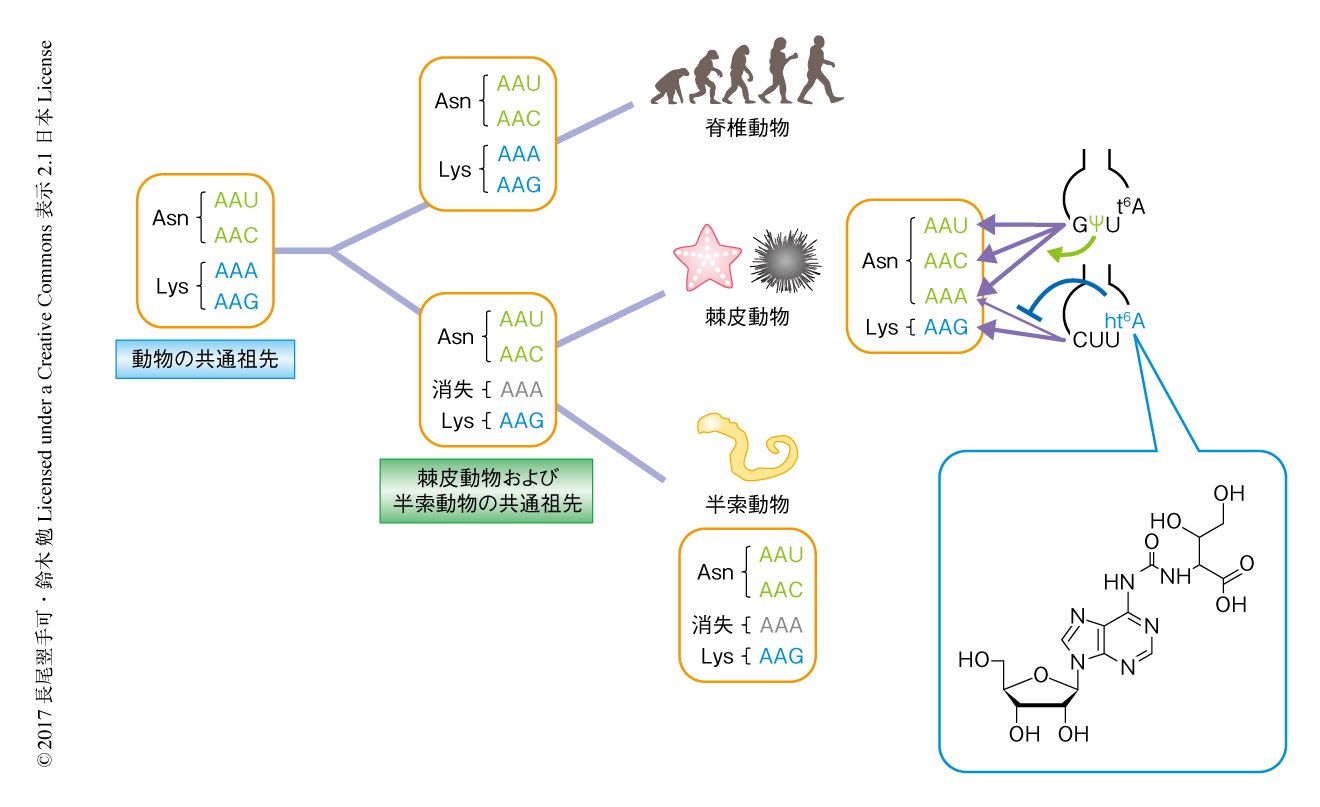
遺伝暗号の解読にはtRNAがかかわり,とくにとなりあったコドンを読み分けるためにはtRNAのアンチコドンに存在する修飾塩基が重要な役割を担う.とくに,tRNAが新たな修飾塩基を獲得することが変則暗号の成立にかかわることが示唆されている2,4).ウニやヒトデなどの棘皮動物のミトコンドリアにおいては,本来はLysを指定するAAAコドンがAsnを指定するよう変化している.1999年,ヒトデのミトコンドリアのtRNAAsnはアンチコドンの2字目にシュードウリジンをもつことが見い出された5).実際に,試験管内翻訳系を用いた実験により,このシュードウリジンをもつtRNAはAsnの本来のコドンであるAAUコドンおよびAACコドンにくわえAAAコドンを読む活性のあることが示された.一方で,棘皮動物のミトコンドリアのtRNALysはアンチコドンとしてCUUをもつため,AAGコドンのみを解読しAAAコドンとは対合しないと考えられてきた.しかし,アンチコドン1字目のCはしばしばコドン3字目のAと対合することが知られており6-8),とくに,アンチコドンの3’側の隣接部位である37位のN6-スレオニルカルバモイルアデノシン(t6A)がこの変則的な対合を可能にすることが示唆されていた9).そこで,筆者らは,棘皮動物のミトコンドリアのtRNALysにはAAAコドンを誤翻訳しないしくみが備わっているのではないかと考え,キタムラサキウニのミトコンドリアからtRNAを精製しその修飾塩基を詳細に解析した.
tRNAに含まれる修飾塩基を調べるためには,その生物から目的のtRNAを単離し精製する必要がある.とくにミトコンドリアのtRNAは存在量がわずかであり,一連の解析に足る量を確保するためには,古典的な生化学的な手法にくわえ,ここでは書ききれないような労力およびノウハウを要する.キタムラサキウニの卵巣からRNA画分を大量に取得し,陰イオンカラムなどによりtRNA画分を濃縮したのち,目的とするtRNAに相補的なプローブを設計し,筆者らの所属する研究室において開発された往復循環クロマトグラフィー法10) により,ミトコンドリアのtRNAAsnおよびtRNALysを単離精製することに成功した.高感度な質量分析法であるRNA-MS法11) を用い単離されたtRNAに含まれる修飾塩基を解析したところ,tRNAAsnのアンチコドンの2字目には,ヒトデと同様にシュードウリジンが検出され,棘皮動物のミトコンドリアにおいてAAAコドンは共通の機構により解読されることが確認された.tRNALysについては,アンチコドンには修飾塩基は見い出されなかったが,アンチコドンの3’側の隣接部位である37位に428 Daの新規の修飾体の存在することが判明した.また,同じ位置からt6Aが微量に検出されたことから,この新規の修飾体はt6Aの誘導体である可能性が考えられた.精密質量の測定からこの新規の修飾体とt6Aとの質量差は15.99522 Daであり,これはちょうど酸素原子1つ(15.99491 Da)に相当したことから,この新規の修飾体はt6Aが水酸化された誘導体であると予想された.さらに,衝突活性化解離法(collision-induced dissociation:CID)による解析により,新規の修飾体はt6Aのスレオニン側鎖のメチル基が水酸化された構造であると推定された.
この新規の修飾体の構造を決定するためには,候補となる化合物を合成し,天然物と比較する必要がある.t6Aの側鎖がスレオニンから4-ヒドロキシスレオニンに置きかわった構造が予想され,t6Aは大腸菌に由来するt6A合成酵素YrdC,YgjD,YeaZ,YjeEの組換え体により試験管内において再構成できるため,基質としてスレオニンの代わりに4-ヒドロキシスレオニンを用いて再構成するという戦略をとった.4-ヒドロキシスレオニンは大腸菌のLtaEを用いグリコールアルデヒドとグリシンを縮合させることにより合成した.試験管内において転写合成された大腸菌tRNALys変異体(アンチコドンCUU)に,4-ヒドロキシスレオニン,ATP,HCO3-の存在下においてt6A合成酵素を作用させたところ,ほぼ100%の効率で37位にヒドロキシ-N6-スレオニルカルバモイルアデノシン(ht6A)を導入することに成功した.この再構成したht6A,および,ミトコンドリアのtRNALysに由来する新規の修飾体を,逆相および順相の異なるカラムを用いた液体クロマトグラフィー-質量分析計にてコインジェクション解析をしたところ,いずれの条件においても溶出時間は完全に一致した.また,衝突活性化解離法による解析においてプロダクトイオンのパターンも完全に一致したことから,この新規の修飾体はht6Aであることが判明した(図1).
ht6Aの機能を解析するため,試験管内において転写合成したtRNALys(アンチコドンCUU)の37位にt6Aあるいはht6Aを酵素的に導入した.大腸菌のリボソームを用い,37位にA37,t6A37,ht6A37の異なる修飾塩基をもつ3種類のtRNALysについてAサイトにおけるAAGコドンおよびAAAコドンに対する結合能について評価した.その結果,3種類のtRNAはいずれも効率よくAAGコドンと結合したが,AAAコドンについては,t6A37はA37より高い結合能を示した.この結果より,t6A37がアンチコドン1字目のCとコドン3字目のAとの対合を安定化するというこれまでの予想がはじめて実験的に証明された.一方,ht6A37はt6A37と比較してAAAコドンに対する結合能が有意に低下した.以上の結果から,ht6AにはアンチコドンとしてCUUをもつtRNAがAAAコドンを誤認識するのをふせぐ役割のあることが示された(図1).
t6Aはすべての生物に保存されているtRNAの修飾塩基であり,細菌においては生育に必須であることが知られている.ht6Aを含めこれまでに5種類のt6A誘導体が知られているが,水酸化されたものはht6Aがはじめてである.一般に,t6Aおよびその誘導体はリボソームにおいてコドンの認識能を高める役割のあることが知られていたが,今回,ht6Aへの水酸基の付加はt6Aの機能をキャンセルする役割を担うことが見い出された.ht6Aの水酸基がどのようにしてAAAコドンとの結合を抑制するのかについては構造生物学的な解析が必要であるが,ひとつの官能基の付加が変則暗号の成立に関与するという今回の知見は,生化学的にも進化学的にも興味深い.
AAAコドンの暗号変化を動物におけるミトコンドリアの進化の過程において考えると,棘皮動物の祖先においてはAAAコドンがミトコンドリアのゲノムから一時的に消失したと考えられている3).実際に,棘皮動物と近縁の半索動物であるギボシムシの一種Balanoglossus carnosusのミトコンドリアのゲノムにおいてAAAコドンは使われていないことからも12),棘皮動物と半索動物の共通祖先においてミトコンドリアのゲノムからAAAコドンが一時的に消失した可能性が強く示唆される.棘皮動物へと進化する過程においてミトコンドリアのtRNAAsnがアンチコドンの2字目にシュードウリジンを獲得すると,ふたたびAAAコドンが出現した際にtRNAAsnに解読されるようになり暗号変化が生じる.この時点で,tRNALysがt6A37をもつと,一部のAAAコドンがLysに誤翻訳され,ミトコンドリアのゲノムにおいてAAAコドンが一時的に2つのアミノ酸を指定する状態になる可能性があった.この状況を回避するため,tRNALysのt6A37を特異的に水酸化する酵素が出現し,ht6A37を獲得することによりAAAコドンがLysからAsnへと完全に暗号変化したと考えられる.
今後は,ht6Aへの変換をつかさどる水酸化酵素の探索も含め,動物のミトコンドリアにおける変則暗号の成立とtRNAにおける修飾塩基との関係を探究していきたい.
略歴:2009年 東京大学大学院工学系研究科博士後期課程 修了,同年 バイオ産業情報化コンソーシアム 特別研究員,2010年 東京大学大学院工学系研究科 研究員を経て,同 助教.
研究テーマ:遺伝暗号の解読における固さと柔らかさにせまる.
関心事:コドン表.パスタの乳化.
鈴木 勉(Tsutomu Suzuki)
東京大学大学院工学系研究科 教授.
研究室URL:http://rna.chem.t.u-tokyo.ac.jp/
© 2017 長尾翌手可・鈴木 勉 Licensed under CC 表示 2.1 日本
(東京大学大学院工学系研究科 化学生命工学専攻)
email:長尾翌手可,鈴木 勉
DOI: 10.7875/first.author.2017.085
Hydroxylation of a conserved tRNA modification establishes non-universal genetic code in echinoderm mitochondria.
Asuteka Nagao, Mitsuhiro Ohara, Kenjyo Miyauchi, Shin-ichi Yokobori, Akihiko Yamagishi, Kimitsuna Watanabe, Tsutomu Suzuki
Nature Structural & Molecular Biology, 24, 778-782 (2017)
要 約
動物のミトコンドリアの翻訳系においては,しばしば普遍暗号から逸脱した変則暗号が用いられる.進化の過程において変則暗号が成立する際にはtRNAにおける修飾塩基の獲得が重要な役割を担う.ウニやヒトデなど棘皮動物のミトコンドリアにおいては,通常はLysを指定するAAAコドンがAsnを指定するよう暗号変化している.過去の研究において,tRNAAsnはアンチコドンの2字目にシュードウリジンを獲得することによりAAAコドンを解読するようになることが明らかにされているが,tRNALysがどのような機構によりAAAコドンの誤認識をふせいでいるかについてはわかっていない.この研究において,筆者らは,キタムラサキウニのミトコンドリアからtRNALysを単離精製し高感度な質量分析法により解析したところ,アンチコドンの3’側の隣接部位に新規の修飾塩基であるヒドロキシ-N6-スレオニルカルバモイルアデノシンを見い出した.リボソームのAサイトにおけるtRNAのコドン認識能を評価したところ,この修飾塩基はAAAコドンへの結合能を抑制する役割のあることが判明した.以上の結果から,棘皮動物のミトコンドリアにおいてtRNALysが新たな修飾塩基を獲得することによりAAAコドンの暗号変化に寄与したと考えられた.
はじめに
1960年代に遺伝暗号表が決定されて以来,すべての生物は共通の遺伝暗号,すなわち,普遍暗号を用いてDNAからタンパク質へ遺伝情報を変換すると信じられていた.このことは生物一元説の大きな根拠にもなっている.しかし,1979年にヒトのミトコンドリアのDNA塩基配列が決定され1),AUAコドンがMet,UGAコドンがTrp,AGAコドンおよびAGGコドンが終止を指定することが判明し,ミトコンドリアの翻訳系において普遍暗号から逸脱した変則暗号の用いられていることが明らかにされた.それ以降,さまざまな動物のミトコンドリアにおいてあいついで変則暗号が発見され2),核のゲノムにおいてもマイコプラズマ,Candida属の酵母,原生動物などにおいて変則暗号がみつかり,遺伝暗号は普遍ではなく変化しうるものであると認識されるようになった3).
遺伝暗号の解読にはtRNAがかかわり,とくにとなりあったコドンを読み分けるためにはtRNAのアンチコドンに存在する修飾塩基が重要な役割を担う.とくに,tRNAが新たな修飾塩基を獲得することが変則暗号の成立にかかわることが示唆されている2,4).ウニやヒトデなどの棘皮動物のミトコンドリアにおいては,本来はLysを指定するAAAコドンがAsnを指定するよう変化している.1999年,ヒトデのミトコンドリアのtRNAAsnはアンチコドンの2字目にシュードウリジンをもつことが見い出された5).実際に,試験管内翻訳系を用いた実験により,このシュードウリジンをもつtRNAはAsnの本来のコドンであるAAUコドンおよびAACコドンにくわえAAAコドンを読む活性のあることが示された.一方で,棘皮動物のミトコンドリアのtRNALysはアンチコドンとしてCUUをもつため,AAGコドンのみを解読しAAAコドンとは対合しないと考えられてきた.しかし,アンチコドン1字目のCはしばしばコドン3字目のAと対合することが知られており6-8),とくに,アンチコドンの3’側の隣接部位である37位のN6-スレオニルカルバモイルアデノシン(t6A)がこの変則的な対合を可能にすることが示唆されていた9).そこで,筆者らは,棘皮動物のミトコンドリアのtRNALysにはAAAコドンを誤翻訳しないしくみが備わっているのではないかと考え,キタムラサキウニのミトコンドリアからtRNAを精製しその修飾塩基を詳細に解析した.
1.キタムラサキウニのミトコンドリアのtRNAの単離精製および新規の修飾体の発見
tRNAに含まれる修飾塩基を調べるためには,その生物から目的のtRNAを単離し精製する必要がある.とくにミトコンドリアのtRNAは存在量がわずかであり,一連の解析に足る量を確保するためには,古典的な生化学的な手法にくわえ,ここでは書ききれないような労力およびノウハウを要する.キタムラサキウニの卵巣からRNA画分を大量に取得し,陰イオンカラムなどによりtRNA画分を濃縮したのち,目的とするtRNAに相補的なプローブを設計し,筆者らの所属する研究室において開発された往復循環クロマトグラフィー法10) により,ミトコンドリアのtRNAAsnおよびtRNALysを単離精製することに成功した.高感度な質量分析法であるRNA-MS法11) を用い単離されたtRNAに含まれる修飾塩基を解析したところ,tRNAAsnのアンチコドンの2字目には,ヒトデと同様にシュードウリジンが検出され,棘皮動物のミトコンドリアにおいてAAAコドンは共通の機構により解読されることが確認された.tRNALysについては,アンチコドンには修飾塩基は見い出されなかったが,アンチコドンの3’側の隣接部位である37位に428 Daの新規の修飾体の存在することが判明した.また,同じ位置からt6Aが微量に検出されたことから,この新規の修飾体はt6Aの誘導体である可能性が考えられた.精密質量の測定からこの新規の修飾体とt6Aとの質量差は15.99522 Daであり,これはちょうど酸素原子1つ(15.99491 Da)に相当したことから,この新規の修飾体はt6Aが水酸化された誘導体であると予想された.さらに,衝突活性化解離法(collision-induced dissociation:CID)による解析により,新規の修飾体はt6Aのスレオニン側鎖のメチル基が水酸化された構造であると推定された.
2.新規の修飾塩基ht6Aの発見
この新規の修飾体の構造を決定するためには,候補となる化合物を合成し,天然物と比較する必要がある.t6Aの側鎖がスレオニンから4-ヒドロキシスレオニンに置きかわった構造が予想され,t6Aは大腸菌に由来するt6A合成酵素YrdC,YgjD,YeaZ,YjeEの組換え体により試験管内において再構成できるため,基質としてスレオニンの代わりに4-ヒドロキシスレオニンを用いて再構成するという戦略をとった.4-ヒドロキシスレオニンは大腸菌のLtaEを用いグリコールアルデヒドとグリシンを縮合させることにより合成した.試験管内において転写合成された大腸菌tRNALys変異体(アンチコドンCUU)に,4-ヒドロキシスレオニン,ATP,HCO3-の存在下においてt6A合成酵素を作用させたところ,ほぼ100%の効率で37位にヒドロキシ-N6-スレオニルカルバモイルアデノシン(ht6A)を導入することに成功した.この再構成したht6A,および,ミトコンドリアのtRNALysに由来する新規の修飾体を,逆相および順相の異なるカラムを用いた液体クロマトグラフィー-質量分析計にてコインジェクション解析をしたところ,いずれの条件においても溶出時間は完全に一致した.また,衝突活性化解離法による解析においてプロダクトイオンのパターンも完全に一致したことから,この新規の修飾体はht6Aであることが判明した(図1).
3.新規の修飾塩基ht6AはAAAコドンの認識を抑制する
ht6Aの機能を解析するため,試験管内において転写合成したtRNALys(アンチコドンCUU)の37位にt6Aあるいはht6Aを酵素的に導入した.大腸菌のリボソームを用い,37位にA37,t6A37,ht6A37の異なる修飾塩基をもつ3種類のtRNALysについてAサイトにおけるAAGコドンおよびAAAコドンに対する結合能について評価した.その結果,3種類のtRNAはいずれも効率よくAAGコドンと結合したが,AAAコドンについては,t6A37はA37より高い結合能を示した.この結果より,t6A37がアンチコドン1字目のCとコドン3字目のAとの対合を安定化するというこれまでの予想がはじめて実験的に証明された.一方,ht6A37はt6A37と比較してAAAコドンに対する結合能が有意に低下した.以上の結果から,ht6AにはアンチコドンとしてCUUをもつtRNAがAAAコドンを誤認識するのをふせぐ役割のあることが示された(図1).
おわりに
t6Aはすべての生物に保存されているtRNAの修飾塩基であり,細菌においては生育に必須であることが知られている.ht6Aを含めこれまでに5種類のt6A誘導体が知られているが,水酸化されたものはht6Aがはじめてである.一般に,t6Aおよびその誘導体はリボソームにおいてコドンの認識能を高める役割のあることが知られていたが,今回,ht6Aへの水酸基の付加はt6Aの機能をキャンセルする役割を担うことが見い出された.ht6Aの水酸基がどのようにしてAAAコドンとの結合を抑制するのかについては構造生物学的な解析が必要であるが,ひとつの官能基の付加が変則暗号の成立に関与するという今回の知見は,生化学的にも進化学的にも興味深い.
AAAコドンの暗号変化を動物におけるミトコンドリアの進化の過程において考えると,棘皮動物の祖先においてはAAAコドンがミトコンドリアのゲノムから一時的に消失したと考えられている3).実際に,棘皮動物と近縁の半索動物であるギボシムシの一種Balanoglossus carnosusのミトコンドリアのゲノムにおいてAAAコドンは使われていないことからも12),棘皮動物と半索動物の共通祖先においてミトコンドリアのゲノムからAAAコドンが一時的に消失した可能性が強く示唆される.棘皮動物へと進化する過程においてミトコンドリアのtRNAAsnがアンチコドンの2字目にシュードウリジンを獲得すると,ふたたびAAAコドンが出現した際にtRNAAsnに解読されるようになり暗号変化が生じる.この時点で,tRNALysがt6A37をもつと,一部のAAAコドンがLysに誤翻訳され,ミトコンドリアのゲノムにおいてAAAコドンが一時的に2つのアミノ酸を指定する状態になる可能性があった.この状況を回避するため,tRNALysのt6A37を特異的に水酸化する酵素が出現し,ht6A37を獲得することによりAAAコドンがLysからAsnへと完全に暗号変化したと考えられる.
今後は,ht6Aへの変換をつかさどる水酸化酵素の探索も含め,動物のミトコンドリアにおける変則暗号の成立とtRNAにおける修飾塩基との関係を探究していきたい.
文 献
- Barrell, B. G., Bankier, A. T. & Drouin, J.: A different genetic code in human mitochondria. Nature, 282, 189-194 (1979)[PubMed]
- Watanabe, K. & Yokobori, S.: tRNA modification and genetic code variations in animal mitochondria. J. Nucleic Acids, 2011, 623095 (2011)[PubMed]
- Osawa, S.: Evolution of the Genetic Code. pp. 205, Oxford University Press, Oxford (1995)
- Suzuki, T., Nagao, A. & Suzuki, T.: Human mitochondrial tRNAs: biogenesis, function, structural aspects, and diseases. Annu. Rev. Genet., 45, 299-329 (2011)[PubMed]
- Tomita, K., Ueda, T. & Watanabe, K.: The presence of pseudouridine in the anticodon alters the genetic code: a possible mechanism for assignment of the AAA lysine codon as asparagine in echinoderm mitochondria. Nucleic Acids Res., 27, 1683-1639 (1999)[PubMed]
- Hirsh, D.: Tryptophan tRNA of Escherichia coli. Nature, 228, 57 (1970)[PubMed]
- Stern, L. & Schulman, L. H.: The role of the minor base N4-acetylcytidine in the function of the Escherichia coli noninitiator methionine transfer RNA. J. Biol. Chem., 253, 6132-6139 (1978)[PubMed]
- Schmeing, T. M., Voorhees, R. M., Kelley, A. C. et al.: How mutations in tRNA distant from the anticodon affect the fidelity of decoding. Nat. Struct. Mol. Biol., 18, 432-436 (2011)[PubMed]
- Yokobori, S., Suzuki, T. & Watanabe, K.: Genetic code variations in mitochondria: tRNA as a major determinant of genetic code plasticity. J. Mol. Evol., 53, 314-326 (2001)[PubMed]
- Miyauchi, K., Ohara, T. & Suzuki, T.: Automated parallel isolation of multiple species of non-coding RNAs by the reciprocal circulating chromatography method. Nucleic Acids Res., 35, e24 (2007)[PubMed]
- Suzuki, T., Ikeuchi, Y., Noma, A. et al.: Mass spectrometric identification and characterization of RNA-modifying enzymes. Methods Enzymol., 425, 211-229 (2007)[PubMed]
- Castresana, J., Feldmaier-Fuchs, G. & Paabo, S.: Codon reassignment and amino acid composition in hemichordate mitochondria. Proc. Natl. Acad. Sci. USA, 95, 3703-3707 (1998)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2009年 東京大学大学院工学系研究科博士後期課程 修了,同年 バイオ産業情報化コンソーシアム 特別研究員,2010年 東京大学大学院工学系研究科 研究員を経て,同 助教.
研究テーマ:遺伝暗号の解読における固さと柔らかさにせまる.
関心事:コドン表.パスタの乳化.
鈴木 勉(Tsutomu Suzuki)
東京大学大学院工学系研究科 教授.
研究室URL:http://rna.chem.t.u-tokyo.ac.jp/
© 2017 長尾翌手可・鈴木 勉 Licensed under CC 表示 2.1 日本