Tudor-SNに依存的なmiRNAの分解はG1期/S期移行を促進する
三好 啓太
(米国Rochester大学School of Medicine and Dentistry,Department of Biochemistry and Biophysics)
email:三好啓太
DOI: 10.7875/first.author.2017.058
Tudor-SN-mediated endonucleolytic decay of human cell microRNAs promotes G1/S phase transition.
Reyad A. Elbarbary, Keita Miyoshi, Jason R. Myers, Peicheng Du, John M. Ashton, Bin Tian, Lynne E. Maquat
Science, 356, 859-862 (2017)
miRNAは遺伝子の発現を制御する非コード小分子RNAのひとつである.miRNAはRISCとよばれるRNA-タンパク質複合体と結合し,配列に依存して標的となるmRNAの翻訳の抑制あるいは不安定化をみちびきその発現を抑制する.このことは,miRNAそれ自体の発現の制御が非常に重要であることを意味する.この研究において,筆者らは,ヒトの細胞における成熟型miRNAの分解の機構に注目し,新たなmiRNAの発現制御機構について明らかにした.RISCの構成タンパク質のひとつとして知られるTudor-SNをノックダウンした細胞におけるmiRNAの発現の解析および生化学的な解析により,Tudor-SNのエンドヌクレアーゼ活性により特異的な成熟型miRNAが分解されることが明らかにされた.さらに,Tudor-SNを欠損した細胞の解析により,Tudor-SNはG1期/S期移行において特異的なmiRNAの分解をひき起こし,G1期/S期移行に必須な遺伝子の発現を促進することが明らかにされた.
miRNA(microRNA)は22~24塩基からなる非コード小分子RNAであり,標的となるmRNAと配列に特異的に対合することによりその発現を時空間的に制御する.miRNA遺伝子の転写,核におけるプロセシング,細胞質への輸送,細胞質におけるDicerに依存的なプロセシング,RISC(RNA-induced silencing complex)とよばれるRNA-タンパク質複合体の形成をへて,22~24塩基の1本鎖RNAである成熟型miRNAが生合成される1).miRNAはRISCの形成により機能をはたす.具体的には,miRNAがRISCの中核となるタンパク質であるArgonauteと直接に結合し,標的となるmRNAを配列に依存して認識するガイドとして機能する.そののち,標的となるmRNAの翻訳の抑制および/あるいは不安定化がひき起こされる.
Tudor-SNは種間で保存されたRNA分解酵素であり,5つのSNドメインおよびTudorドメインからなる.細菌のTudor-SNのホモログを用いたin vitroにおける解析により,Tudor-SNは標的となるRNAをエンドヌクレアーゼ活性により切断したのち,3’-5’エキソヌクレアーゼ活性により分解することが明らかにされた2).Tudor-SNはショウジョウバエや線虫のRISCにおける最初のエンドヌクレアーゼとして同定され,RISC活性の中核となるタンパク質であると予測されたが3),のちに,RISC活性の中核となるタンパク質はTudor-SNではなくArgonauteであることが証明された4).哺乳類におけるTudor-SNの機能は多岐にわたり,転写因子としての機能や,ADARによるアデノシンからイノシンへのRNA編集をうけたRNAの分解などにも関与する5).また,哺乳類の細胞においてpri-miR-142はADARによるRNA編集をうけたのちTudor-SNにより分解される6).
ヒトのHEK293T細胞を用いてRNAi法によりTudor-SNをノックダウンし,miRNAの生合成におけるTudor-SNの関与についてRT-定量PCR法およびノーザンブロット法により解析した.いくつかのmiRNAを選び,それらのpri-miRNA,pre-miRNA,成熟型miRNAの発現量を解析した結果,miR-31-5p,miR-29b-3p,miR-125aにおいて成熟型miRNAの蓄積が観察された.一方,それらのpri-miRNAおよびpre-miRNAの発現量に変化はなかった.また,miR-3648およびmiR-128-3pにおいて成熟型miRNAの蓄積は観察されなかった.これらの結果より,Tudor-SNが特異的な成熟型miRNAの発現に関与することが示唆された.また,HeLa細胞を用いた実験においても同様の結果が得られた.
Tudor-SNにより発現の制御されるmiRNAは細胞において機能的であるどうか調べた.miR-31-5pおよびmiR-29b-3pはITGA5およびLOXの発現を抑制することが知られている.Tudor-SNをノックダウンしたHEK293T細胞において,ITGA5およびLOXの発現は抑制された.一方,Tudor-SNをノックダウンしたHEK293T細胞にmiR-31-5pあるいはmiR-29b-3pに対する阻害剤を添加したところ,ITGA5あるいはLOXの発現は上昇した.これらの結果から,Tudor-SNは機能的なmiRNAの発現制御に関与することが示された.
Tudor-SNをノックダウンしたHEK293T細胞においてmiRNA前駆体の蓄積が観察されなかったことから,Tudor-SNによるmiRNAの発現制御は,RISCの形成においてmiRNAがArgonauteにローディングされる段階,あるいは,RISCが形成されたのちと考えられた.Tudor-SNをノックダウンしたHEK293T細胞の抽出液を用いた生化学的な手法により,Tudor-SNはmiRNAのArgonaute2へのローディングには関与しないことが明らかにされた.DicerとTudor-SNをともにノックダウンしたHEK293T細胞においてTudor-SNの標的となるmiRNAの半減期を解析することにより,RISCが形成されたのちのTudor-SNによるmiRNAの発現制御について調べた.その結果,Tudor-SNの標的となるmiRNAの半減期は延長された.さらに,人工のmiRNAの擬様体をTudor-SNをノックダウンしたHEK293T細胞に導入したところ,その半減期は延長された.これらの結果から,Tudor-SNによるmiRNAの発現制御は,miRNA生合成における制御ではなく,RISCが形成されたのちの成熟型miRNAを標的とすることが示唆された.
この機構におけるTudor-SNのエンドヌクレアーゼ活性の関与について調べるため,SNドメインを欠損したTudor-SN変異体を解析したところ,SN1ドメインおよびSN4ドメインの関与が明らかにされた.また,スタフィロコッカスリボヌクレアーゼの阻害剤であるpdTpをHEK293T細胞の培養液に添加したところ,Tudor-SNの標的となるmiRNAの蓄積が観察された.これらの結果から,この機構においてはTudor-SNのSNドメインによるmiRNAの分解活性が必須であることが明らかにされ,このmiRNAの分解の機構はTumiD(Tudor-SN-mediated miRNA decay)と名づけられた.
Tudor-SNをノックダウンしたHEK293T細胞を用いたmiRNAシークエンス解析において,35の成熟型miRNAについて蓄積が観察された.この結果はRT-定量PCR法により確認し,Tudor-SNをノックダウンしたHEK293T細胞においてmiRNA前駆体に変化がみられず成熟型miRNAの蓄積が観察されたmiRNAをTumiD標的miRNAとした.
ゲノムにおいてmiRNAクラスターとよばれる複数のmiRNAを発現する転写ユニットが存在する.いくつかのmiRNAクラスターに由来するmiRNAの発現につきTumiD機構の関与について調べた結果,同じmiRNAクラスターに由来してもそれぞれのmiRNAによりTumiD機構による影響は異なっていた.このことから,TumiD機構はそれぞれの成熟型miRNAを特異的に標的とすることが支持された.
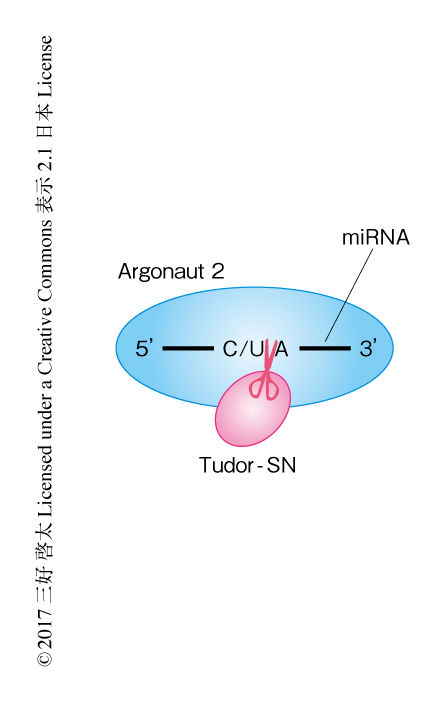
大腸菌から精製した組換えTudor-SNを用いてmiRNAのin vitroにおける切断アッセイを行った.TumiD機構の標的であるmiR-31-5p,miR-29b-3p,miR-126-3p,また,TumiD機構の標的ではないmiR-3648のTudor-SNによる切断の様式を解析した結果,Tudor-SNはCA配列およびUA配列をCa2+に依存して切断した(図1).Tudor-SNにより切断されないmiR-3648はCA配列あるいはUA配列を含まなかったことから,miR-31-5pのCA配列を含む領域をmiR-3648の配列と置換したキメラmiRNAについて解析したところTudor-SNにより切断されなくなった.また,CA間あるいはUA間のホスホジスルフィド結合をホスホロチオエート結合に置換したところTudor-SN切断活性に対し耐性を示した.さらに,miR-3648にCA配列を導入してTudor-SNによる切断アッセイを行ったところ,Tudor-SNはCA配列あるいはUA配列が両方の末端から5塩基より内側に存在するmiRNAを効率的に切断することが明らかにされた.この結果とmiRNAシークエンス解析により同定された35のTumiD標的miRNAとを照らし合わせると,33のmiRNAがこの定義にあてはまった.また,これらのmiRNAのマウスにおけるホモログについて調べたところ,31のmiRNAが保存されていた.
Tudor-SNはArgonaute2と結合したmiRNAを切断できるのかどうか,HEK293T細胞の抽出液を用いて合成miRNAをArgonaute2にローディングさせ,免疫沈降法によりmiRNA-Argonaute2複合体を得てここに組換えTudor-SNを添加したところ,miRNAは切断された.この結果から,Tudor-SNが細胞においてArgonaute2と結合したmiRNAの切断することは可能であると示唆された.
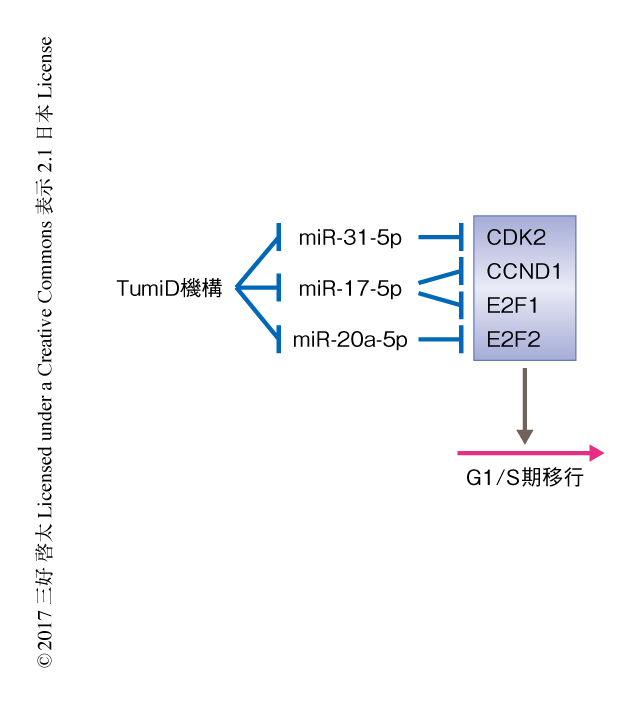
マウスの胎仔線維芽細胞を用いた研究により,Tudor-SNは細胞周期におけるG1期からS期への移行に関与することが明らかにされていた7).そこで,Tudor-SNがHEK293T細胞においても同様の機能をもつかどうか検証するため,CRISPR-Cas9系を用いた遺伝子編集技術によりTudor-SNを欠損したHEK293T細胞を作製した.Tudor-SNを欠損したHEK293T細胞においてはG1期/S期移行に遅れが観察され,G1期からS期への移行においてmiR-31-5p,miR-17-5p,miR-20a-5の蓄積が観察された.これらのmiRNAはG1期/S期移行に必要な遺伝子の発現抑制に関与し,すなわち,miR-31-5pはCDK2を,miR-17-5pはCCND1およびE2F1を,miR-20a-5pはE2F2の発現を抑制する.Tudor-SNを欠損したHEK293T細胞においてはこれらのmiRNAが蓄積し,標的となる遺伝子の発現が抑制された(図2).一方で,これらのmiRNAの阻害剤をTudor-SNを欠損したHEK293T細胞に導入すると,標的となる遺伝子の発現抑制は阻害された.さらに,Tudor-SNを欠損したHEK293T細胞に野生型のTudor-SNを発現させると,これらのmiRNAの発現が抑制され,それらの標的となる遺伝子の発現が促進された.これらの結果より,時期に特異的なTumiD機構を介したmiRNAの発現制御により,細胞周期に関連する遺伝子の発現が制御されることが明らかにされた.
この研究において,Tudor-SNのエンドヌクレアーゼ活性を介した特異的な成熟型miRNAの分解機構が明らかにされた.これまで,植物,線虫,ヒトにおいてエキソヌクレアーゼによるmiRNAの分解が報告されているが8),エンドヌクレアーゼによるmiRNA分解機構の報告ははじめてである.成熟型miRNAの分解機構についての知見は少ない.その理由のひとつとして,miRNAの半減期が平均して5日以上9) と長く解析が困難であることがあげられる.この研究においては,Tudor-SNのsiRNAを細胞にトランスフェクションしてから4日後に解析したことから,TumiD機構の標的となるmiRNAの数は同定された数よりも実際にはさらに多いと考えられる.
TumiD機構に関しては多くの疑問が残されている.Tudor-SNはRISCにおいて標的となるmRNAと対合したmiRNAを分解できるのだろうか? あるいは,TumiD機構のためにRISCからmiRNAを解離させるタンパク質が必要なのか? また,おのおののmiRNAはTumiD機構により分解される時期や場所は異なると考えられるが,その特異性はどのように生じるのか? トリガーとなるタンパク質が必要なのか? TumiD機構の標的となるmiRNAに対し機能的な分類は可能なのか?
ヒトのさまざまながん細胞においてTudor-SNの過剰発現が観察されており,その発現量とがん患者の予後には関連がみられる.Tudor-SNは発がん経路に関連するタンパク質と相互作用することにより発がんを促進すると考えられている5).これにくわえ,Tudor-SNの過剰発現によりひき起こされるmiRNAの発現制御の異常もがん化の促進の一因と考えられるのではないだろうか.
略歴:2004年 広島大学大学院生物圏科学研究科 修了,同年 徳島大学ゲノム機能研究センター 博士研究員,2008年 慶應義塾大学医学部 助教,2012年 米国Rochester大学School of Medicine and Dentistry博士研究員を経て,2015年より同Staff Scientist.
研究テーマ:小分子RNAの生合成の機構およびその制御.
© 2017 三好 啓太 Licensed under CC 表示 2.1 日本
(米国Rochester大学School of Medicine and Dentistry,Department of Biochemistry and Biophysics)
email:三好啓太
DOI: 10.7875/first.author.2017.058
Tudor-SN-mediated endonucleolytic decay of human cell microRNAs promotes G1/S phase transition.
Reyad A. Elbarbary, Keita Miyoshi, Jason R. Myers, Peicheng Du, John M. Ashton, Bin Tian, Lynne E. Maquat
Science, 356, 859-862 (2017)
要 約
miRNAは遺伝子の発現を制御する非コード小分子RNAのひとつである.miRNAはRISCとよばれるRNA-タンパク質複合体と結合し,配列に依存して標的となるmRNAの翻訳の抑制あるいは不安定化をみちびきその発現を抑制する.このことは,miRNAそれ自体の発現の制御が非常に重要であることを意味する.この研究において,筆者らは,ヒトの細胞における成熟型miRNAの分解の機構に注目し,新たなmiRNAの発現制御機構について明らかにした.RISCの構成タンパク質のひとつとして知られるTudor-SNをノックダウンした細胞におけるmiRNAの発現の解析および生化学的な解析により,Tudor-SNのエンドヌクレアーゼ活性により特異的な成熟型miRNAが分解されることが明らかにされた.さらに,Tudor-SNを欠損した細胞の解析により,Tudor-SNはG1期/S期移行において特異的なmiRNAの分解をひき起こし,G1期/S期移行に必須な遺伝子の発現を促進することが明らかにされた.
はじめに
miRNA(microRNA)は22~24塩基からなる非コード小分子RNAであり,標的となるmRNAと配列に特異的に対合することによりその発現を時空間的に制御する.miRNA遺伝子の転写,核におけるプロセシング,細胞質への輸送,細胞質におけるDicerに依存的なプロセシング,RISC(RNA-induced silencing complex)とよばれるRNA-タンパク質複合体の形成をへて,22~24塩基の1本鎖RNAである成熟型miRNAが生合成される1).miRNAはRISCの形成により機能をはたす.具体的には,miRNAがRISCの中核となるタンパク質であるArgonauteと直接に結合し,標的となるmRNAを配列に依存して認識するガイドとして機能する.そののち,標的となるmRNAの翻訳の抑制および/あるいは不安定化がひき起こされる.
Tudor-SNは種間で保存されたRNA分解酵素であり,5つのSNドメインおよびTudorドメインからなる.細菌のTudor-SNのホモログを用いたin vitroにおける解析により,Tudor-SNは標的となるRNAをエンドヌクレアーゼ活性により切断したのち,3’-5’エキソヌクレアーゼ活性により分解することが明らかにされた2).Tudor-SNはショウジョウバエや線虫のRISCにおける最初のエンドヌクレアーゼとして同定され,RISC活性の中核となるタンパク質であると予測されたが3),のちに,RISC活性の中核となるタンパク質はTudor-SNではなくArgonauteであることが証明された4).哺乳類におけるTudor-SNの機能は多岐にわたり,転写因子としての機能や,ADARによるアデノシンからイノシンへのRNA編集をうけたRNAの分解などにも関与する5).また,哺乳類の細胞においてpri-miR-142はADARによるRNA編集をうけたのちTudor-SNにより分解される6).
1.Tudor-SNは特異的な成熟型miRNAの発現に関与する
ヒトのHEK293T細胞を用いてRNAi法によりTudor-SNをノックダウンし,miRNAの生合成におけるTudor-SNの関与についてRT-定量PCR法およびノーザンブロット法により解析した.いくつかのmiRNAを選び,それらのpri-miRNA,pre-miRNA,成熟型miRNAの発現量を解析した結果,miR-31-5p,miR-29b-3p,miR-125aにおいて成熟型miRNAの蓄積が観察された.一方,それらのpri-miRNAおよびpre-miRNAの発現量に変化はなかった.また,miR-3648およびmiR-128-3pにおいて成熟型miRNAの蓄積は観察されなかった.これらの結果より,Tudor-SNが特異的な成熟型miRNAの発現に関与することが示唆された.また,HeLa細胞を用いた実験においても同様の結果が得られた.
Tudor-SNにより発現の制御されるmiRNAは細胞において機能的であるどうか調べた.miR-31-5pおよびmiR-29b-3pはITGA5およびLOXの発現を抑制することが知られている.Tudor-SNをノックダウンしたHEK293T細胞において,ITGA5およびLOXの発現は抑制された.一方,Tudor-SNをノックダウンしたHEK293T細胞にmiR-31-5pあるいはmiR-29b-3pに対する阻害剤を添加したところ,ITGA5あるいはLOXの発現は上昇した.これらの結果から,Tudor-SNは機能的なmiRNAの発現制御に関与することが示された.
2.Tudor-SNはRISCが形成されたのちにmiRNAを分解する
Tudor-SNをノックダウンしたHEK293T細胞においてmiRNA前駆体の蓄積が観察されなかったことから,Tudor-SNによるmiRNAの発現制御は,RISCの形成においてmiRNAがArgonauteにローディングされる段階,あるいは,RISCが形成されたのちと考えられた.Tudor-SNをノックダウンしたHEK293T細胞の抽出液を用いた生化学的な手法により,Tudor-SNはmiRNAのArgonaute2へのローディングには関与しないことが明らかにされた.DicerとTudor-SNをともにノックダウンしたHEK293T細胞においてTudor-SNの標的となるmiRNAの半減期を解析することにより,RISCが形成されたのちのTudor-SNによるmiRNAの発現制御について調べた.その結果,Tudor-SNの標的となるmiRNAの半減期は延長された.さらに,人工のmiRNAの擬様体をTudor-SNをノックダウンしたHEK293T細胞に導入したところ,その半減期は延長された.これらの結果から,Tudor-SNによるmiRNAの発現制御は,miRNA生合成における制御ではなく,RISCが形成されたのちの成熟型miRNAを標的とすることが示唆された.
この機構におけるTudor-SNのエンドヌクレアーゼ活性の関与について調べるため,SNドメインを欠損したTudor-SN変異体を解析したところ,SN1ドメインおよびSN4ドメインの関与が明らかにされた.また,スタフィロコッカスリボヌクレアーゼの阻害剤であるpdTpをHEK293T細胞の培養液に添加したところ,Tudor-SNの標的となるmiRNAの蓄積が観察された.これらの結果から,この機構においてはTudor-SNのSNドメインによるmiRNAの分解活性が必須であることが明らかにされ,このmiRNAの分解の機構はTumiD(Tudor-SN-mediated miRNA decay)と名づけられた.
3.TumiD機構の標的となるmiRNAの同定
Tudor-SNをノックダウンしたHEK293T細胞を用いたmiRNAシークエンス解析において,35の成熟型miRNAについて蓄積が観察された.この結果はRT-定量PCR法により確認し,Tudor-SNをノックダウンしたHEK293T細胞においてmiRNA前駆体に変化がみられず成熟型miRNAの蓄積が観察されたmiRNAをTumiD標的miRNAとした.
ゲノムにおいてmiRNAクラスターとよばれる複数のmiRNAを発現する転写ユニットが存在する.いくつかのmiRNAクラスターに由来するmiRNAの発現につきTumiD機構の関与について調べた結果,同じmiRNAクラスターに由来してもそれぞれのmiRNAによりTumiD機構による影響は異なっていた.このことから,TumiD機構はそれぞれの成熟型miRNAを特異的に標的とすることが支持された.
4.Tudor-SNはArgonaute2と結合したmiRNAのCA配列あるいはUA配列を切断する
大腸菌から精製した組換えTudor-SNを用いてmiRNAのin vitroにおける切断アッセイを行った.TumiD機構の標的であるmiR-31-5p,miR-29b-3p,miR-126-3p,また,TumiD機構の標的ではないmiR-3648のTudor-SNによる切断の様式を解析した結果,Tudor-SNはCA配列およびUA配列をCa2+に依存して切断した(図1).Tudor-SNにより切断されないmiR-3648はCA配列あるいはUA配列を含まなかったことから,miR-31-5pのCA配列を含む領域をmiR-3648の配列と置換したキメラmiRNAについて解析したところTudor-SNにより切断されなくなった.また,CA間あるいはUA間のホスホジスルフィド結合をホスホロチオエート結合に置換したところTudor-SN切断活性に対し耐性を示した.さらに,miR-3648にCA配列を導入してTudor-SNによる切断アッセイを行ったところ,Tudor-SNはCA配列あるいはUA配列が両方の末端から5塩基より内側に存在するmiRNAを効率的に切断することが明らかにされた.この結果とmiRNAシークエンス解析により同定された35のTumiD標的miRNAとを照らし合わせると,33のmiRNAがこの定義にあてはまった.また,これらのmiRNAのマウスにおけるホモログについて調べたところ,31のmiRNAが保存されていた.
Tudor-SNはArgonaute2と結合したmiRNAを切断できるのかどうか,HEK293T細胞の抽出液を用いて合成miRNAをArgonaute2にローディングさせ,免疫沈降法によりmiRNA-Argonaute2複合体を得てここに組換えTudor-SNを添加したところ,miRNAは切断された.この結果から,Tudor-SNが細胞においてArgonaute2と結合したmiRNAの切断することは可能であると示唆された.
5.TumiD機構はG1期/S期移行を促進する
マウスの胎仔線維芽細胞を用いた研究により,Tudor-SNは細胞周期におけるG1期からS期への移行に関与することが明らかにされていた7).そこで,Tudor-SNがHEK293T細胞においても同様の機能をもつかどうか検証するため,CRISPR-Cas9系を用いた遺伝子編集技術によりTudor-SNを欠損したHEK293T細胞を作製した.Tudor-SNを欠損したHEK293T細胞においてはG1期/S期移行に遅れが観察され,G1期からS期への移行においてmiR-31-5p,miR-17-5p,miR-20a-5の蓄積が観察された.これらのmiRNAはG1期/S期移行に必要な遺伝子の発現抑制に関与し,すなわち,miR-31-5pはCDK2を,miR-17-5pはCCND1およびE2F1を,miR-20a-5pはE2F2の発現を抑制する.Tudor-SNを欠損したHEK293T細胞においてはこれらのmiRNAが蓄積し,標的となる遺伝子の発現が抑制された(図2).一方で,これらのmiRNAの阻害剤をTudor-SNを欠損したHEK293T細胞に導入すると,標的となる遺伝子の発現抑制は阻害された.さらに,Tudor-SNを欠損したHEK293T細胞に野生型のTudor-SNを発現させると,これらのmiRNAの発現が抑制され,それらの標的となる遺伝子の発現が促進された.これらの結果より,時期に特異的なTumiD機構を介したmiRNAの発現制御により,細胞周期に関連する遺伝子の発現が制御されることが明らかにされた.
おわりに
この研究において,Tudor-SNのエンドヌクレアーゼ活性を介した特異的な成熟型miRNAの分解機構が明らかにされた.これまで,植物,線虫,ヒトにおいてエキソヌクレアーゼによるmiRNAの分解が報告されているが8),エンドヌクレアーゼによるmiRNA分解機構の報告ははじめてである.成熟型miRNAの分解機構についての知見は少ない.その理由のひとつとして,miRNAの半減期が平均して5日以上9) と長く解析が困難であることがあげられる.この研究においては,Tudor-SNのsiRNAを細胞にトランスフェクションしてから4日後に解析したことから,TumiD機構の標的となるmiRNAの数は同定された数よりも実際にはさらに多いと考えられる.
TumiD機構に関しては多くの疑問が残されている.Tudor-SNはRISCにおいて標的となるmRNAと対合したmiRNAを分解できるのだろうか? あるいは,TumiD機構のためにRISCからmiRNAを解離させるタンパク質が必要なのか? また,おのおののmiRNAはTumiD機構により分解される時期や場所は異なると考えられるが,その特異性はどのように生じるのか? トリガーとなるタンパク質が必要なのか? TumiD機構の標的となるmiRNAに対し機能的な分類は可能なのか?
ヒトのさまざまながん細胞においてTudor-SNの過剰発現が観察されており,その発現量とがん患者の予後には関連がみられる.Tudor-SNは発がん経路に関連するタンパク質と相互作用することにより発がんを促進すると考えられている5).これにくわえ,Tudor-SNの過剰発現によりひき起こされるmiRNAの発現制御の異常もがん化の促進の一因と考えられるのではないだろうか.
文 献
- Ha, M. & Kim, V. N.: Regulation of microRNA biogenesis. Nat. Rev. Mol. Cell. Biol., 15, 509-524 (2014)[PubMed]
- Reddi, K. K.: Mode of action of micrococcal phosphodiesterase. Nature, 187, 74-75 (1960)[PubMed]
- Caudy, A. A., Ketting, R. F., Hammond, S. M. et al.: A micrococcal nuclease homologue in RNAi effector complexes. Nature, 425, 411-414 (2003)[PubMed]
- Song, J. J., Smith, S. K., Hannon, G. J. et al.: Crystal structure of Argonaute and its implications for RISC slicer activity. Science, 305, 1434-1437 (2004)[PubMed]
- Gutierrez-Beltran, E., Denisenko, T. V., Zhivotovsky, B. et al.: Tudor staphylococcal nuclease: biochemistry and functions. Cell Death Differ., 23, 1739-1748 (2016)[PubMed]
- Yang, W., Chendrimada, T. P., Wang, Q. et al.: Modulation of microRNA processing and expression through RNA editing by ADAR deaminases. Nat. Struct. Mol. Biol., 13, 13-21 (2006)[PubMed]
- Su, C., Zhang, C., Tecle, A. et al.: Tudor staphylococcal nuclease (Tudor-SN), a novel regulator facilitating G1/S phase transition, acting as a co-activator of E2F-1 in cell cycle regulation. J. Biol. Chem., 290, 7208-7220 (2015)[PubMed]
- Ruegger, S. & Grosshans, H.: MicroRNA turnover: when, how, and why. Trends Biochem. Sci., 37, 436-446 (2012)[PubMed]
- Gantier, M. P., McCoy, C. E., Rusinova, I. et al.: Analysis of microRNA turnover in mammalian cells following Dicer1 ablation. Nucleic Acids Res., 39, 5692-5703 (2011)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2004年 広島大学大学院生物圏科学研究科 修了,同年 徳島大学ゲノム機能研究センター 博士研究員,2008年 慶應義塾大学医学部 助教,2012年 米国Rochester大学School of Medicine and Dentistry博士研究員を経て,2015年より同Staff Scientist.
研究テーマ:小分子RNAの生合成の機構およびその制御.
© 2017 三好 啓太 Licensed under CC 表示 2.1 日本