オーバーラッピングジヌクレオソームのX線結晶構造解析
加藤大貴・越阪部晃永・胡桃坂仁志
(早稲田大学大学院先進理工学研究科 電気・情報生命専攻構造生物学研究室)
email:胡桃坂仁志
DOI: 10.7875/first.author.2017.033
Crystal structure of the overlapping dinucleosome composed of hexasome and octasome.
Daiki Kato, Akihisa Osakabe, Yasuhiro Arimura, Yuka Mizukami, Naoki Horikoshi, Kazumi Saikusa, Satoko Akashi, Yoshifumi Nishimura, Sam-Yong Park, Jumpei Nogami, Kazumitsu Maehara, Yasuyuki Ohkawa, Atsushi Matsumoto, Hidetoshi Kono, Rintaro Inoue, Masaaki Sugiyama, Hitoshi Kurumizaka
Science, 356, 205-208 (2017)
真核生物においてゲノムDNAはクロマチンを形成することにより核の内部に収納される.クロマチンはヌクレオソームを基本単位として構成されるが,ヌクレオソームはゲノムDNAの機能の発現に対しおおむね阻害的にはたらく.それゆえ,ゲノムDNAが転写,複製,修復,組換えなどの機能を発現する際にはクロマチンリモデリングタンパク質によりヌクレオソームの再配置がひき起こされることが知られている.SWI/SNFファミリーに属するクロマチンリモデリングタンパク質によりヌクレオソームが再配置される際に,再配置されたヌクレオソームが隣接するヌクレオソームと衝突し2つのヌクレオソームが重なり合ったオーバーラッピングジヌクレオソームが形成されることが報告されている.しかし,その構造や実態は明らかにされていなかった.この研究においては,オーバーラッピングジヌクレオソームの立体構造をX線結晶構造解析により原子分解能で明らかにした.オーバーラッピングジヌクレオソームにおいてはヒストン六量体にDNAが巻きついたヘキサソームユニットとヒストン八量体にDNAが巻きついたオクタソームユニットが緊密に重なり合った構造を形成していた.さらに,単体のヌクレオソームの立体構造においてはみられなかったヘキサソームユニットとオクタソームユニットとのあいだの新規の相互作用が見い出された.さらに,ゲノム解析によりヒトの細胞においてオーバーラッピングジヌクレオソームが転写開始点の直下に形成されることが示唆された.また,生化学的な解析によりオーバーラッピングジヌクレオソームにはリンカーヒストンであるヒストンH1が1分子のみ結合するという結果が得られた.これらの結果から,ヌクレオソームの再配置により形成される新規のクロマチン構造ユニットであるオーバーラッピングジヌクレオソームの立体構造が明らかになり,クロマチンリモデリング機構の一端が明らかにされた.
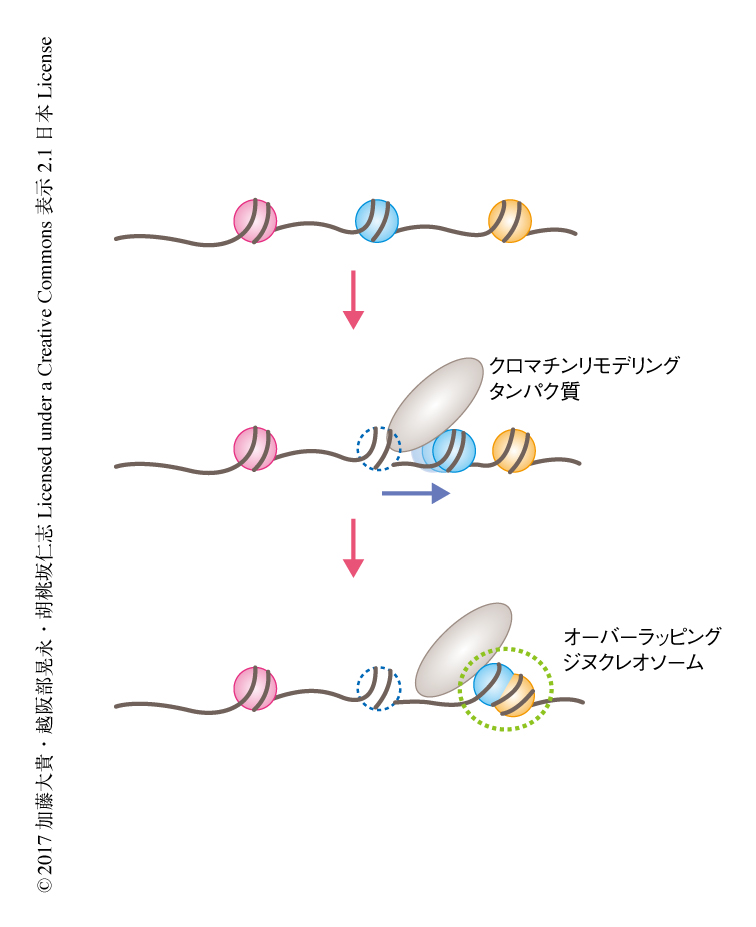
遺伝情報の担体であるゲノムDNAはクロマチンを形成することによりコンパクトに折りたたまれて核の内部に収納される.クロマチンはヌクレオソームを基本単位とし,それらが連なることにより構成される.ヌクレオソームは4種類のヒストン,ヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4のおのおの2分子ずつからなるヒストン八量体のまわりに,約150塩基対のDNAが左巻きに1.65回巻きついた円盤状の構造体である1).クロマチンはダイナミックに構造変換することが知られており,近年,このクロマチンの動的な構造変換がDNAの機能の発現を制御することが明らかにされつつある.クロマチンの構造変換のひとつとしてクロマチンリモデリングが知られている.クロマチンリモデリングの際にはヌクレオソームがゲノムDNAにおいて再配置するが2,3),その際には,隣接するヌクレオソームと再配置されたヌクレオソームとが衝突し2つのヌクレオソームが重なり合ったオーバーラッピングジヌクレオソームが形成されると指摘されていた4)(図1).また,先行研究により,DNA配列に依存的にオーバーラッピングジヌクレオソームが形成される可能性も示唆されていた4-6).しかし,オーバーラッピングジヌクレオソームの立体構造は明らかにされておらず,その形成の機構や機能などについての知見はほとんど得られていなかった.この研究においては,試験管内におけるオーバーラッピングジヌクレオソームの再構成に成功し,X線結晶構造解析によりその立体構造を原子分解能で明らかにした.
ヒトに由来するヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4を組換えタンパク質として精製し,ヒストン八量体を再構成した.先行研究により,強力なポジショニング配列である601配列を改変しタンデムにならべた250塩基対のDNAを用いることにより試験管内においてオーバーラッピングジヌクレオソームが再構成されることが示されていた4,7).そこで,この先行研究において示された250塩基対のDNAおよびヒストン八量体を用いて,塩透析法により試験管内においてオーバーラッピングジヌクレオソームを再構成した.再構成されたオーバーラッピングジヌクレオソームを電気泳動により高純度に精製し,ネイティブエレクトロスプレーイオン化法を用いた質量分析法により解析した.その結果,オーバーラッピングジヌクレオソームには3分子のヒストンH2A,3分子のヒストンH2B,4分子のヒストンH3,4分子のヒストンH4が含まれることが明らかにされた.これは,先行研究により示唆されていたオーバーラッピングジヌクレオソームの構成とよく一致した.さまざまな長さのDNAを用いて再構成し精製した試料を用いて同様に解析したところ,266塩基対のDNAを用いた場合にはオーバーラッピングジヌクレオソームは形成されず,通常のヌクレオソームが2つ連なったジヌクレオソームが形成された.
精製したオーバーラッピングジヌクレオソームを用いて,シッティングドロップ蒸気拡散法により結晶化することによりオーバーラッピングジヌクレオソームの単結晶が得られた.得られた単結晶を用いて,大型放射光施設SPring-8においてX線回折実験を実施し,3.14Åの分解能のX線回折データを取得した.ヒトのヌクレオソームの立体構造をサーチモデルとした分子置換法により位相を決定し8),オーバーラッピングジヌクレオソームの立体構造を決定することに成功した(PDB ID:5GSE,図2).
立体構造から,オーバーラッピングジヌクレオソームはヒストン六量体にDNAが巻きついたヘキサソームユニットとヒストン八量体にDNAが巻きついたオクタソームユニットが緊密に重なり合うことにより,250塩基対のDNAがヒストン十四量体のまわりに約3回巻きついた特殊な構造を形成することが明らかにされた(図2).また,ヘキサソームユニットではオクタソームユニットと接触する面においてそれぞれ1分子のヒストンH2AとヒストンH2B(ヒストンH2A-ヒストンH2B二量体)が欠落していた.さらに,ヒストンH2A-ヒストンH2B二量体の欠落によりヌクレオソームのディスク様の表面に存在する酸性パッチとよばれる領域が1カ所失われ,さらに,オクタソームユニットの片側の酸性パッチが隣接するヘキサソームユニットによりおおいかくされていた.これまでの報告により,核内タンパク質がヌクレオソームの酸性パッチと特異的に相互作用することが示されている9).このことから,オーバーラッピングジヌクレオソームが形成されることにより,核内タンパク質と酸性パッチとの結合が通常のヌクレオソームに比べ低下することが考えられた.実際に,酸性パッチと特異的に結合する核内タンパク質RCC1の結合が,対象として使用した通常のジヌクレオソームに比べ,オーバーラッピングジヌクレオソームにおいてはいちじるしく低下した.さらに,オーバーラッピングジヌクレオソームの内部の構造の特徴として,オクタソームユニットとの接触面に位置するヘキサソームユニットのヒストンH3のαNヘリックスが顕著に短くなっていた.
オーバーラッピングジヌクレオソームにおいては1組のヒストンH2A-ヒストンH2B二量体が欠落することによりヘキサソームユニットとオクタソームユニットとが緊密に接触する.オーバーラッピングジヌクレオソームの構造を詳細に解析した結果,オクタソームユニットに巻きついたDNAとヘキサソームユニットのヒストンH3のLys56およびヒストンH3のThr80,また,ヘキサソームユニットに巻きついたDNAとオクタソームユニットのヒストンH2AのAsn68,ヒストンH2AのArg71,ヒストンH2BのLys108,ヒストンH2BのSer112,ヒストンH2BのLys116が近接することが明らかにされた.したがって,これら7種類のアミノ酸残基がヘキサソームユニットとオクタソームユニットとの結合に関与することが考えられた.そこで,これら7種類すべてのアミノ酸残基に変異を導入したヒストン変異体を作製し,これらを含む変異型のオーバーラッピングジヌクレオソームを再構成し,ヒストンの含量を質量分析法により解析した.その結果,変異型のオーバーラッピングジヌクレオソームは野生型のオーバーラッピングジヌクレオソームと同様のヒストンの構成をもつことが確認された.X線小角散乱法により溶液におけるオーバーラッピングジヌクレオソームの構造を解析したところ,変異型のオーバーラッピングジヌクレオソームの慣性半径は野生型のオーバーラッピングジヌクレオソームに比べ大きくなっていた.このことから,変異型のオーバーラッピングジヌクレオソームにおいてはヘキサソームユニットとオクタソームユニットとの相互作用が野生型のオーバーラッピングジヌクレオソームよりも減弱されたため,より開いた構造を形成すると考えられた.変異体をさらに詳細に解析することにより,ヒストンH3のLys56およびヒストンH3のThr80がヘキサソームユニットとオクタソームユニットとのあいだの緊密な相互作用において重要であることが明らかにされた.
リンカーヒストンであるヒストンH1はヌクレオソームのDNAおよびヌクレオソームのあいだのリンカーDNAの両方と結合することによりクロマチンをコンパクトに凝縮するはたらきをもつと考えられている10).X線結晶構造解析によりオーバーラッピングジヌクレオソームにおいては2つのヌクレオソームのあいだのリンカーDNAがなくなっていることが明らかにされた.そこで,オーバーラッピングジヌクレオソームに対するヒストンH1の結合能をゲルシフトアッセイにより評価した.その結果,オクタソームユニットが2つ連なった通常のジヌクレオソームでは2段階のバンドシフトが検出されたが,オーバーラッピングジヌクレオソームでは1段階のバンドシフトのみが検出された.また,ヒストンH1とオーバーラッピングジヌクレオソームとの複合体,および,ヒストンH1と通常のジヌクレオソームとの複合体をスクロース密度勾配を用いた分析超遠心法により精製し,SDS-ポリアクリルアミドゲル電気泳動法によりおのおののヒストンの含量を検討した.その結果,オーバーラッピングジヌクレオソームには通常のジヌクレオソームと比べ半分の量のヒストンH1が結合したことから,オーバーラッピングジヌクレオソームにはヒストンH1が1分子のみ結合することが示唆された.さらに,ヒストンH1がオーバーラッピングジヌクレオソームのどの領域に結合するかを明らかにするためフットプリンティング法により解析したところ,オクタソームユニットのDNAの中心付近に結合することがわかった.
再構成したオーバーラッピングジヌクレオソームは,リンカーDNAを特異的に切断するDNA分解酵素ミクロコッカスヌクレアーゼに対し高い耐性を示した.この性質を利用して,ヒトの細胞におけるオーバーラッピングジヌクレオソームの形成について調べた.HeLa細胞から核を調製し,ミクロコッカスヌクレアーゼによりクロマチンをモノヌクレオソームのレベルまで消化した.得られたDNA断片を電気泳動法により精製し,およそ150塩基対および250塩基対のDNA断片を次世代シークエンサーにより解析しゲノムワイドにマッピングした.その結果,転写開始点の直下の領域に250塩基対のDNA断片が有意にマッピングされた.このことから,実際にオーバーラッピングジヌクレオソームはヒトのゲノムにおいて転写開始点の直下に形成されることが示唆された.
クロマチンリモデリングにともなうクロマチンの構造変換の機構についてはいまだ不明な点が多い.この研究においては,クロマチンリモデリングにより生じるオーバーラッピングジヌクレオソームの立体構造の解明に成功した.オーバーラッピングジヌクレオソームはヒストンH2A-ヒストンH2B二量体の欠落によりヘキサソームユニットとオクタソームユニットとが緊密に重なり合った特殊な高次構造を形成することが明らかにされた.さらに,HeLa細胞において転写開始点の直下のヌクレオソームにオーバーラッピングジヌクレオソームが形成されることを示唆するデータが得られた.今後,オーバーラッピングジヌクレオソームとDNAの機能の発現との詳細な関係性が明らかにされると期待される.
1997年にはじめてヌクレオソームの立体構造が明らかにされてから20年が経ち,これまで100種以上のヌクレオソームの立体構造が解析された.しかし,いずれもヒストン八量体にDNAが巻きついた通常のヌクレオソームの構造と類似したものであった.また,近年の全ゲノム解析から遺伝子の発現とクロマチンの動的な構造変換の機構との関連が明らかにされつつある.この研究において明らかにされた,ヒストン十四量体にDNAが約3回巻きついた新規のクロマチン構造ユニットであるオーバーラッピングジヌクレオソームの立体構造の知見をもとに,クロマチンの動的な構造変換の機構の理解が進展することが期待される.
略歴:早稲田大学大学院先進理工学研究科博士後期課程 在学中.
研究テーマ:クロマチンの構造解析.
抱負:遺伝子発現の制御機構をクロマチンダイナミクスの視点から明らかにしたい.
越阪部 晃永(Akihisa Osakabe)
オーストリアGregor Mendel Institute博士研究員.
胡桃坂 仁志(Hitoshi Kurumizaka)
早稲田大学大学院先進理工学研究科 教授.
研究室URL:http://www.eb.waseda.ac.jp/kurumizaka/
© 2017 加藤大貴・越阪部晃永・胡桃坂仁志 Licensed under CC 表示 2.1 日本
(早稲田大学大学院先進理工学研究科 電気・情報生命専攻構造生物学研究室)
email:胡桃坂仁志
DOI: 10.7875/first.author.2017.033
Crystal structure of the overlapping dinucleosome composed of hexasome and octasome.
Daiki Kato, Akihisa Osakabe, Yasuhiro Arimura, Yuka Mizukami, Naoki Horikoshi, Kazumi Saikusa, Satoko Akashi, Yoshifumi Nishimura, Sam-Yong Park, Jumpei Nogami, Kazumitsu Maehara, Yasuyuki Ohkawa, Atsushi Matsumoto, Hidetoshi Kono, Rintaro Inoue, Masaaki Sugiyama, Hitoshi Kurumizaka
Science, 356, 205-208 (2017)
要 約
真核生物においてゲノムDNAはクロマチンを形成することにより核の内部に収納される.クロマチンはヌクレオソームを基本単位として構成されるが,ヌクレオソームはゲノムDNAの機能の発現に対しおおむね阻害的にはたらく.それゆえ,ゲノムDNAが転写,複製,修復,組換えなどの機能を発現する際にはクロマチンリモデリングタンパク質によりヌクレオソームの再配置がひき起こされることが知られている.SWI/SNFファミリーに属するクロマチンリモデリングタンパク質によりヌクレオソームが再配置される際に,再配置されたヌクレオソームが隣接するヌクレオソームと衝突し2つのヌクレオソームが重なり合ったオーバーラッピングジヌクレオソームが形成されることが報告されている.しかし,その構造や実態は明らかにされていなかった.この研究においては,オーバーラッピングジヌクレオソームの立体構造をX線結晶構造解析により原子分解能で明らかにした.オーバーラッピングジヌクレオソームにおいてはヒストン六量体にDNAが巻きついたヘキサソームユニットとヒストン八量体にDNAが巻きついたオクタソームユニットが緊密に重なり合った構造を形成していた.さらに,単体のヌクレオソームの立体構造においてはみられなかったヘキサソームユニットとオクタソームユニットとのあいだの新規の相互作用が見い出された.さらに,ゲノム解析によりヒトの細胞においてオーバーラッピングジヌクレオソームが転写開始点の直下に形成されることが示唆された.また,生化学的な解析によりオーバーラッピングジヌクレオソームにはリンカーヒストンであるヒストンH1が1分子のみ結合するという結果が得られた.これらの結果から,ヌクレオソームの再配置により形成される新規のクロマチン構造ユニットであるオーバーラッピングジヌクレオソームの立体構造が明らかになり,クロマチンリモデリング機構の一端が明らかにされた.
はじめに
遺伝情報の担体であるゲノムDNAはクロマチンを形成することによりコンパクトに折りたたまれて核の内部に収納される.クロマチンはヌクレオソームを基本単位とし,それらが連なることにより構成される.ヌクレオソームは4種類のヒストン,ヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4のおのおの2分子ずつからなるヒストン八量体のまわりに,約150塩基対のDNAが左巻きに1.65回巻きついた円盤状の構造体である1).クロマチンはダイナミックに構造変換することが知られており,近年,このクロマチンの動的な構造変換がDNAの機能の発現を制御することが明らかにされつつある.クロマチンの構造変換のひとつとしてクロマチンリモデリングが知られている.クロマチンリモデリングの際にはヌクレオソームがゲノムDNAにおいて再配置するが2,3),その際には,隣接するヌクレオソームと再配置されたヌクレオソームとが衝突し2つのヌクレオソームが重なり合ったオーバーラッピングジヌクレオソームが形成されると指摘されていた4)(図1).また,先行研究により,DNA配列に依存的にオーバーラッピングジヌクレオソームが形成される可能性も示唆されていた4-6).しかし,オーバーラッピングジヌクレオソームの立体構造は明らかにされておらず,その形成の機構や機能などについての知見はほとんど得られていなかった.この研究においては,試験管内におけるオーバーラッピングジヌクレオソームの再構成に成功し,X線結晶構造解析によりその立体構造を原子分解能で明らかにした.
1.オーバーラッピングジヌクレオソームの立体構造の決定
ヒトに由来するヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4を組換えタンパク質として精製し,ヒストン八量体を再構成した.先行研究により,強力なポジショニング配列である601配列を改変しタンデムにならべた250塩基対のDNAを用いることにより試験管内においてオーバーラッピングジヌクレオソームが再構成されることが示されていた4,7).そこで,この先行研究において示された250塩基対のDNAおよびヒストン八量体を用いて,塩透析法により試験管内においてオーバーラッピングジヌクレオソームを再構成した.再構成されたオーバーラッピングジヌクレオソームを電気泳動により高純度に精製し,ネイティブエレクトロスプレーイオン化法を用いた質量分析法により解析した.その結果,オーバーラッピングジヌクレオソームには3分子のヒストンH2A,3分子のヒストンH2B,4分子のヒストンH3,4分子のヒストンH4が含まれることが明らかにされた.これは,先行研究により示唆されていたオーバーラッピングジヌクレオソームの構成とよく一致した.さまざまな長さのDNAを用いて再構成し精製した試料を用いて同様に解析したところ,266塩基対のDNAを用いた場合にはオーバーラッピングジヌクレオソームは形成されず,通常のヌクレオソームが2つ連なったジヌクレオソームが形成された.
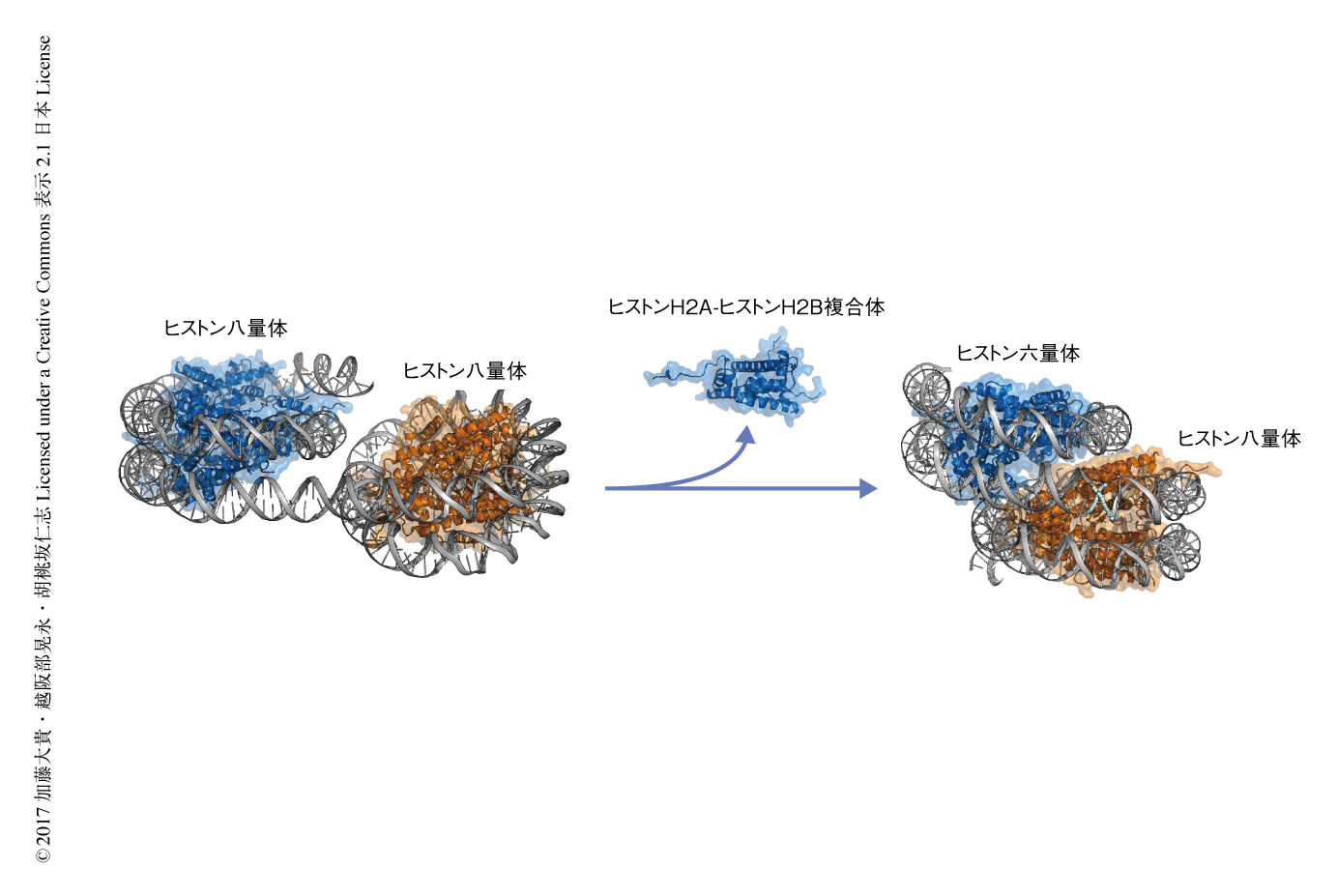
精製したオーバーラッピングジヌクレオソームを用いて,シッティングドロップ蒸気拡散法により結晶化することによりオーバーラッピングジヌクレオソームの単結晶が得られた.得られた単結晶を用いて,大型放射光施設SPring-8においてX線回折実験を実施し,3.14Åの分解能のX線回折データを取得した.ヒトのヌクレオソームの立体構造をサーチモデルとした分子置換法により位相を決定し8),オーバーラッピングジヌクレオソームの立体構造を決定することに成功した(PDB ID:5GSE,図2).
2.オーバーラッピングジヌクレオソームの立体構造の特徴
立体構造から,オーバーラッピングジヌクレオソームはヒストン六量体にDNAが巻きついたヘキサソームユニットとヒストン八量体にDNAが巻きついたオクタソームユニットが緊密に重なり合うことにより,250塩基対のDNAがヒストン十四量体のまわりに約3回巻きついた特殊な構造を形成することが明らかにされた(図2).また,ヘキサソームユニットではオクタソームユニットと接触する面においてそれぞれ1分子のヒストンH2AとヒストンH2B(ヒストンH2A-ヒストンH2B二量体)が欠落していた.さらに,ヒストンH2A-ヒストンH2B二量体の欠落によりヌクレオソームのディスク様の表面に存在する酸性パッチとよばれる領域が1カ所失われ,さらに,オクタソームユニットの片側の酸性パッチが隣接するヘキサソームユニットによりおおいかくされていた.これまでの報告により,核内タンパク質がヌクレオソームの酸性パッチと特異的に相互作用することが示されている9).このことから,オーバーラッピングジヌクレオソームが形成されることにより,核内タンパク質と酸性パッチとの結合が通常のヌクレオソームに比べ低下することが考えられた.実際に,酸性パッチと特異的に結合する核内タンパク質RCC1の結合が,対象として使用した通常のジヌクレオソームに比べ,オーバーラッピングジヌクレオソームにおいてはいちじるしく低下した.さらに,オーバーラッピングジヌクレオソームの内部の構造の特徴として,オクタソームユニットとの接触面に位置するヘキサソームユニットのヒストンH3のαNヘリックスが顕著に短くなっていた.
3.ヘキサソームユニットとオクタソームユニットとの相互作用
オーバーラッピングジヌクレオソームにおいては1組のヒストンH2A-ヒストンH2B二量体が欠落することによりヘキサソームユニットとオクタソームユニットとが緊密に接触する.オーバーラッピングジヌクレオソームの構造を詳細に解析した結果,オクタソームユニットに巻きついたDNAとヘキサソームユニットのヒストンH3のLys56およびヒストンH3のThr80,また,ヘキサソームユニットに巻きついたDNAとオクタソームユニットのヒストンH2AのAsn68,ヒストンH2AのArg71,ヒストンH2BのLys108,ヒストンH2BのSer112,ヒストンH2BのLys116が近接することが明らかにされた.したがって,これら7種類のアミノ酸残基がヘキサソームユニットとオクタソームユニットとの結合に関与することが考えられた.そこで,これら7種類すべてのアミノ酸残基に変異を導入したヒストン変異体を作製し,これらを含む変異型のオーバーラッピングジヌクレオソームを再構成し,ヒストンの含量を質量分析法により解析した.その結果,変異型のオーバーラッピングジヌクレオソームは野生型のオーバーラッピングジヌクレオソームと同様のヒストンの構成をもつことが確認された.X線小角散乱法により溶液におけるオーバーラッピングジヌクレオソームの構造を解析したところ,変異型のオーバーラッピングジヌクレオソームの慣性半径は野生型のオーバーラッピングジヌクレオソームに比べ大きくなっていた.このことから,変異型のオーバーラッピングジヌクレオソームにおいてはヘキサソームユニットとオクタソームユニットとの相互作用が野生型のオーバーラッピングジヌクレオソームよりも減弱されたため,より開いた構造を形成すると考えられた.変異体をさらに詳細に解析することにより,ヒストンH3のLys56およびヒストンH3のThr80がヘキサソームユニットとオクタソームユニットとのあいだの緊密な相互作用において重要であることが明らかにされた.
4.オーバーラッピングジヌクレオソームに対するヒストンH1の結合
リンカーヒストンであるヒストンH1はヌクレオソームのDNAおよびヌクレオソームのあいだのリンカーDNAの両方と結合することによりクロマチンをコンパクトに凝縮するはたらきをもつと考えられている10).X線結晶構造解析によりオーバーラッピングジヌクレオソームにおいては2つのヌクレオソームのあいだのリンカーDNAがなくなっていることが明らかにされた.そこで,オーバーラッピングジヌクレオソームに対するヒストンH1の結合能をゲルシフトアッセイにより評価した.その結果,オクタソームユニットが2つ連なった通常のジヌクレオソームでは2段階のバンドシフトが検出されたが,オーバーラッピングジヌクレオソームでは1段階のバンドシフトのみが検出された.また,ヒストンH1とオーバーラッピングジヌクレオソームとの複合体,および,ヒストンH1と通常のジヌクレオソームとの複合体をスクロース密度勾配を用いた分析超遠心法により精製し,SDS-ポリアクリルアミドゲル電気泳動法によりおのおののヒストンの含量を検討した.その結果,オーバーラッピングジヌクレオソームには通常のジヌクレオソームと比べ半分の量のヒストンH1が結合したことから,オーバーラッピングジヌクレオソームにはヒストンH1が1分子のみ結合することが示唆された.さらに,ヒストンH1がオーバーラッピングジヌクレオソームのどの領域に結合するかを明らかにするためフットプリンティング法により解析したところ,オクタソームユニットのDNAの中心付近に結合することがわかった.
5.ヒトの細胞におけるオーバーラッピングジヌクレオソームの形成
再構成したオーバーラッピングジヌクレオソームは,リンカーDNAを特異的に切断するDNA分解酵素ミクロコッカスヌクレアーゼに対し高い耐性を示した.この性質を利用して,ヒトの細胞におけるオーバーラッピングジヌクレオソームの形成について調べた.HeLa細胞から核を調製し,ミクロコッカスヌクレアーゼによりクロマチンをモノヌクレオソームのレベルまで消化した.得られたDNA断片を電気泳動法により精製し,およそ150塩基対および250塩基対のDNA断片を次世代シークエンサーにより解析しゲノムワイドにマッピングした.その結果,転写開始点の直下の領域に250塩基対のDNA断片が有意にマッピングされた.このことから,実際にオーバーラッピングジヌクレオソームはヒトのゲノムにおいて転写開始点の直下に形成されることが示唆された.
おわりに
クロマチンリモデリングにともなうクロマチンの構造変換の機構についてはいまだ不明な点が多い.この研究においては,クロマチンリモデリングにより生じるオーバーラッピングジヌクレオソームの立体構造の解明に成功した.オーバーラッピングジヌクレオソームはヒストンH2A-ヒストンH2B二量体の欠落によりヘキサソームユニットとオクタソームユニットとが緊密に重なり合った特殊な高次構造を形成することが明らかにされた.さらに,HeLa細胞において転写開始点の直下のヌクレオソームにオーバーラッピングジヌクレオソームが形成されることを示唆するデータが得られた.今後,オーバーラッピングジヌクレオソームとDNAの機能の発現との詳細な関係性が明らかにされると期待される.
1997年にはじめてヌクレオソームの立体構造が明らかにされてから20年が経ち,これまで100種以上のヌクレオソームの立体構造が解析された.しかし,いずれもヒストン八量体にDNAが巻きついた通常のヌクレオソームの構造と類似したものであった.また,近年の全ゲノム解析から遺伝子の発現とクロマチンの動的な構造変換の機構との関連が明らかにされつつある.この研究において明らかにされた,ヒストン十四量体にDNAが約3回巻きついた新規のクロマチン構造ユニットであるオーバーラッピングジヌクレオソームの立体構造の知見をもとに,クロマチンの動的な構造変換の機構の理解が進展することが期待される.
文 献
- Luger, K., Mader, A. W., Richmond, R. K. et al.: Crystal structure of the nucleosome core particle at 2.8Å resolution. Nature, 389, 251-260 (1997)[PubMed]
- Mueller-Planitz, F., Klinker, H. & Becker, P. B.: Nucleosome sliding mechanisms: new twists in a looped history. Nat. Struct. Mol. Biol., 20, 1026-1032 (2013)[PubMed]
- Clapier, C. R. & Cairns, B. R.: The biology of chromatin remodeling complexes. Annu. Rev. Biochem., 78, 273-304 (2009)[PubMed]
- Engeholm, M., de Jager, M., Flaus, A. et al.: Nucleosomes can invade DNA territories occupied by their neighbors. Nat. Struct. Mol. Biol., 16, 151-158 (2009)[PubMed]
- Brogaard, K. R., Xi, L., Wang, J. P. et al.: A map of nucleosome positions in yeast at base-pair resolution. Nature, 486, 496-501 (2012)[PubMed]
- Struhl, K. & Segal, E.: Determinants of nucleosome positioning. Nat. Struct. Mol. Biol., 20, 267-273 (2013)[PubMed]
- Lowary, P. T. & Widom, J.: New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol., 276, 19-42 (1998)[PubMed]
- Tachiwana, H., Kagawa, W., Osakabe, A. et al.: Structural basis of instability of the nucleosome containing a testis-specific histone variant, human H3T. Proc. Natl. Acad. Sci. USA, 107, 10454-10459 (2010)[PubMed]
- Makde, R. D., England, J. R., Yennawar, H. P. et al.: Structure of RCC1 chromatin factor bound to the nucleosome core particle. Nature, 467, 562-566 (2010)[PubMed]
- Simpson, R. T.: Structure of the chromatosome, a chromatin particle containing 160 base pairs of DNA and all the histones. Biochemistry, 17, 5524-5531 (1978)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:早稲田大学大学院先進理工学研究科博士後期課程 在学中.
研究テーマ:クロマチンの構造解析.
抱負:遺伝子発現の制御機構をクロマチンダイナミクスの視点から明らかにしたい.
越阪部 晃永(Akihisa Osakabe)
オーストリアGregor Mendel Institute博士研究員.
胡桃坂 仁志(Hitoshi Kurumizaka)
早稲田大学大学院先進理工学研究科 教授.
研究室URL:http://www.eb.waseda.ac.jp/kurumizaka/
© 2017 加藤大貴・越阪部晃永・胡桃坂仁志 Licensed under CC 表示 2.1 日本