tRNA前駆体は5’キャップ構造により安定化されている
大平高之・鈴木 勉
(東京大学大学院工学系研究科 化学生命工学専攻)
email:大平高之,鈴木 勉
DOI: 10.7875/first.author.2016.080
Precursors of tRNAs are stabilized by methylguanosine cap structures.
Takayuki Ohira, Tsutomu Suzuki
Nature Chemical Biology, 12, 648-655 (2016)
遺伝暗号の解読をつかさどるtRNAは転写ののち多様な修飾をうけることにより成熟し本来の機能を発揮する.しかしながら,tRNA前駆体がプロセシングされる過程においてどのタイミングで修飾されるのか,また,その順序については不明な点が多く残されている.このような背景から,出芽酵母からさまざまなtRNA前駆体を単離しRNA高感度質量分析法を駆使してtRNAの修飾について詳細に解析したところ,5’リーダー配列をもつtRNA前駆体に5’キャップ構造が形成されていることが見い出された.一般に,5’キャップ構造はRNAポリメラーゼIIの転写と共役して形成されることが知られているが,tRNAはRNAポリメラーゼIIIの転写産物であり,この発見はこれまでの常識をくつがえす知見となった.遺伝学的な解析により,この5’キャップ構造はtRNA前駆体を5’エキソヌクレアーゼによる分解から保護する役割のあることが明らかにされた.
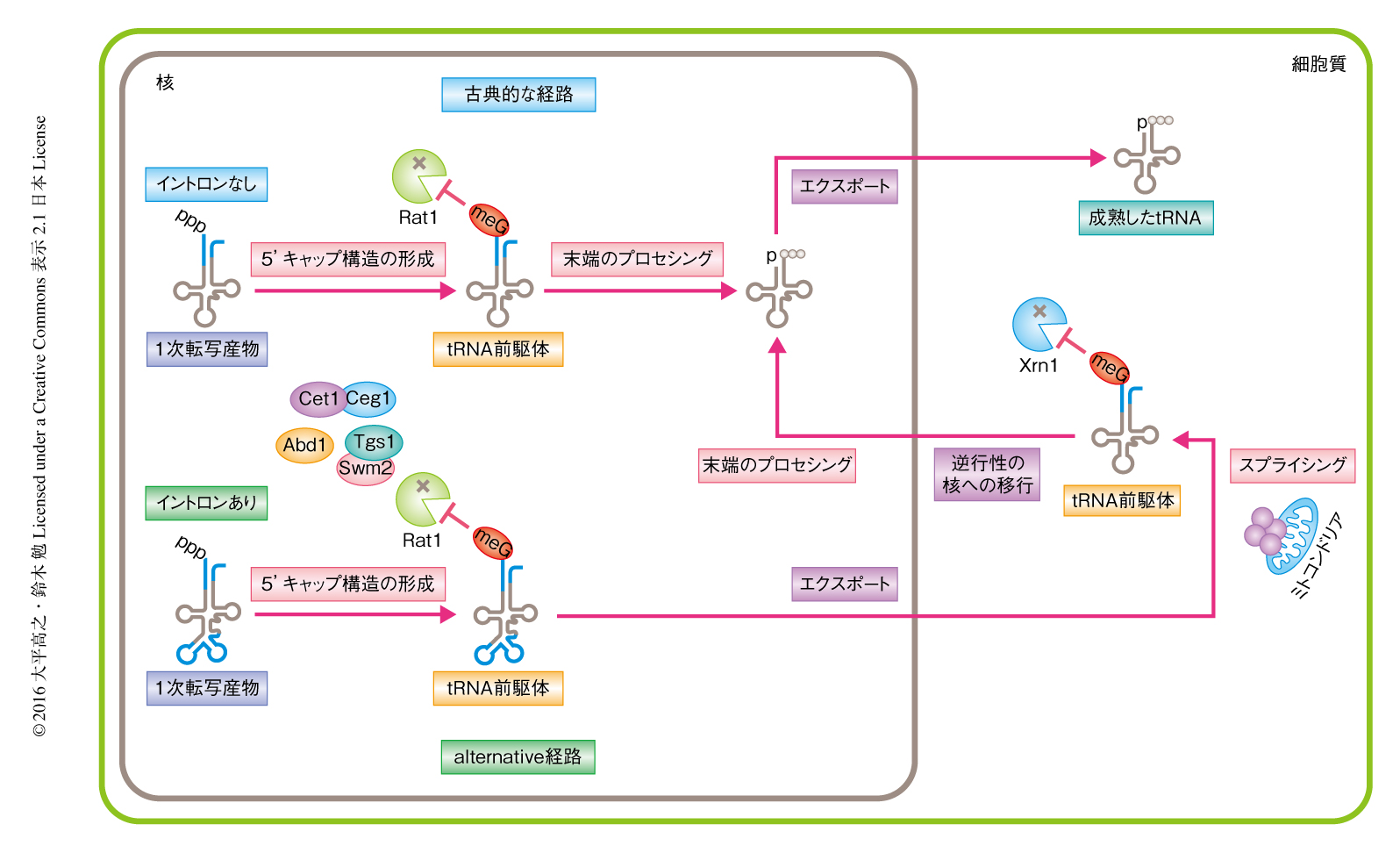
tRNAはタンパク質の合成の過程においてmRNAのもつ遺伝情報をアミノ酸の配列へと変換するアダプター分子である.活発に増殖する細胞においてはタンパク質の高い合成能を維持するため大量のtRNAを合成する必要がある.真核生物における細胞質でのtRNAの合成に関する研究は出芽酵母を用いて詳細に行われている1).出芽酵母は43種類のtRNAをもち,これらは274個のtRNA遺伝子にコードされている.このうち,59の遺伝子がイントロンを含む2).tRNA遺伝子は核においてRNAポリメラーゼIIIにより転写され1次転写産物が生じる.そののち,RNase Pにより5’リーダー配列が切除され,Trz1pあるいは3’エキソヌクレアーゼにより3’トレーラー配列が取り除かれる.つづいて,CCA付加酵素により3’末端にCCA配列が付加される.5’末端および3’末端がプロセシングされたtRNA前駆体はExportin-T(Los1p)およびRanGTPase(Gsp1p)の作用により細胞質へと輸送される.tRNA前駆体がイントロンをもつ場合には,細胞質へと輸送されたのちミトコンドリア外膜に局在するtRNAスプライシング装置によりイントロンが除去される3).このようなtRNAの成熟化の経路を古典的な(classical)経路とよぶ.ところが,先行研究において,5’末端および3’末端のプロセシングよりさきにイントロンが取り除かれたtRNA前駆体の存在が知られていた4)(図1).tRNAスプライシング装置は細胞質にあることから,プロセシングされるまえのtRNA前駆体が細胞質へと輸送されイントロンが除去されることにより,この特徴的なtRNA前駆体が生じる.このtRNA前駆体は細胞質から核へと逆行的に輸送されることが示されていることから5),このtRNA前駆体は核へと移行したのち,核において5’末端および3’末端がプロセシングされ,ふたたび細胞質へと輸送されるという,もうひとつのtRNAの成熟化の経路であるalternative経路の存在が示唆される.
出芽酵母のtRNAには25種類の修飾が含まれており,tRNAの全残基のおよそ15%が修飾されている.tRNAの修飾はtRNAの安定性やコドンの認識などtRNAの構造や機能において重要な役割をはたしている.tRNAの修飾はtRNA前駆体がプロセシングされる過程において,核や細胞質に局在するtRNA修飾酵素により形成される.以前に,筆者らは,フェニルアラニンtRNAの37位にあるワイブトシン修飾が形成される過程において,tRNA前駆体は細胞質においてスプライシングされたのち核へと逆行的に輸送され,その第1段階の修飾である1-メチルグアノシン修飾をうけたのち,ふたたび細胞質へと輸送されることが必要であることを報告した6).このように,tRNAの成熟の過程において,tRNA前駆体が細胞内をダイナミックに移動しながらプロセシングや修飾をうけていることがしだいに明らかにされつつある.筆者らは,このような背景から,tRNAの成熟の過程をより詳細に理解することを目的として,出芽酵母からさまざまなtRNA前駆体を単離し修飾の状態や末端の構造について解析してきた.
tRNA前駆体の修飾の状態や末端の構造を調べるため,5’リーダー配列を切除するRNase Pの発現を制御できる出芽酵母の株を用いた.このtetO7-POP4株はドキシサイクリンの添加によりRNase Pの構成成分であるPop4pの発現を転写の段階において抑制することができる.この株においてRNase Pを抑制して5’リーダー配列をもつtRNA前駆体を蓄積させ,筆者らの研究室において開発された往復循環クロマトグラフィー7) およびゲルの切り出しにより単離し,RNA高感度質量分析法8) によりその構造を解析した.その結果,5’リーダー配列の5’末端にm7Gキャップ構造,ジメチルGキャップ構造,トリメチルGキャップ構造というメチルグアノシンキャップ構造をもつtRNA前駆体が見い出された.この結果を検証するため,tetO7-POP4株においてRNase Pを抑制し,抗キャップ抗体を用いて全RNA画分から5’キャップ構造をもつRNAを免疫沈降により濃縮したのち,数種類のtRNAをノーザンブロット法により解析した.その結果,さまざまなtRNA前駆体が免疫沈降され,5’リーダー配列をもつtRNA前駆体は5’キャップ構造をもつことが明らかにされ,この現象を“tRNA前駆体のキャップ化”(pre-tRNA capping)と命名した.
出芽酵母の野生株からtRNA前駆体を単離したところ,やはり5’キャップ構造が検出された.野生株において5’キャップ構造をもつ率は5~25%であり,RNase Pを抑制したtetO7-POP4株における率と比較すると低かったが,この解析は活発に増殖する細胞におけるtRNA前駆体のスナップショットをみており,実際には,さらに多くのtRNA前駆体が5’キャップ構造をもつ可能性があると考えられた.
さまざまなtRNA前駆体の末端の構造の解析により,5’キャップ構造をもつ率はtRNA前駆体の種ごとに大きく異なることがわかった.5’リーダー配列が3’トレーラー配列と対合しやすいと5’キャップ構造をもつ率は低くなり,逆に,5’末端が1本鎖構造をとりやすいtRNA前駆体ほど5’キャップ構造をもつ率が高くなることが示唆された.5’キャップ構造の形成はtRNA前駆体の末端の構造に大きく影響されることが判明した.
ヒトのHeLa細胞を用いて同様に解析したところ,5’キャップ構造をもつtRNA前駆体はヒトの細胞においても観察された.この結果から,tRNA前駆体における5’キャップ構造の形成は真核生物の種をこえて広く保存されている可能性が示唆された.
通常,5’キャップ構造はmRNAやsnRNAなどRNAポリメラーゼIIの転写産物において形成されることが知られている.それは,転写の開始初期の段階においてRNAポリメラーゼIIにキャップ形成酵素が結合することにより,新生したRNA鎖の5’末端に5’キャップ構造が形成されるからである.tRNAは通常はRNAポリメラーゼIIIにより転写されるが,一部がRNAポリメラーゼIIにより転写されることによりtRNA前駆体において5’キャップ構造が形成される可能性が考えられた.そこで,RNAポリメラーゼIIIの温度感受性株を非許容温度において培養してRNAポリメラーゼIIIによる転写を阻害した際に,5’キャップ構造をもつtRNA前駆体の量に変化がみられるかどうかを調べた.その結果,抗キャップ抗体により免疫沈降させた画分においてtRNA前駆体のノーザンブロッティング法におけるシグナルが顕著に減少したことから,5’キャップ構造をもつtRNA前駆体はRNAポリメラーゼIIIの転写産物であることが判明した.
tRNA前駆体における5’キャップ構造の形成に必要な酵素を同定した.出芽酵母における5’キャップ構造の付加酵素であるCeg1pは必須遺伝子にコードされているため,tetO7-POP4株を背景としてCeg1pの温度感受性変異株および過剰発現株を構築した.それぞれの株を非許容温度にて培養することによりRNase Pを抑制して5’リーダー配列をもつtRNA前駆体を蓄積させたのち,全RNAを抽出しそこからtRNAIleUAUを単離した.RNA高感度質量分析法により解析したところ,Ceg1pの温度感受性変異株では5’キャップ構造の形成が阻害され,Ceg1pの過剰発現株では5’キャップ構造をもつ率が上昇した.したがって,Ceg1pがtRNA前駆体における5’キャップ構造の形成に必須であることが判明した(図1).さらに,5’キャップ構造のメチル化酵素であるTgs1pがジメチルGキャップ構造およびトリメチルGキャップ構造の形成にかかわること,Tgs1pのパートナータンパク質であるSwm2pがTgs1pのはたらきをサポートすることも示された(図1).5’末端に二リン酸を形成する脱リン酸化酵素Cet1p,および,m7Gキャップ構造の形成に必要なメチル化酵素Abd1pの関与については未解析であるが,これらもtRNA前駆体における5’キャップ構造の形成に必要であると推察される.
一般に,5’キャップ構造にはRNAを5’エキソヌクレアーゼから保護する役割が知られている.そこで,tRNA前駆体も同様に5’キャップ構造により分解から保護されているかどうかを検証するため,tetO7-POP4株を背景としたCeg1pの温度感受性変異株および過剰発現株を用い,これらの株を非許容温度にて培養することによりRNase Pを抑制し,蓄積するtRNA前駆体の量をノーザンブロット法により調べた.その結果,Ceg1pの過剰発現株においては蓄積するtRNA前駆体の量が増加したのに対し,Ceg1pの温度感受性変異株においては顕著に減少した.そこで,tRNA前駆体が5’エキソヌクレアーゼにより分解されているかどうかを調べるため,tetO7-POP4株を背景としてMET22遺伝子の欠損株を構築した.MET22遺伝子を欠損させると5’エキソヌクレアーゼ(Rat1pおよびXrn1p)の活性を強力に阻害する5’,3’-ビスホスホアデノシンが細胞に蓄積する.この株においてRNase Pを抑制したところ,一部の5’リーダー配列をもつtRNA前駆体が顕著に蓄積した.また,細胞質に局在する5’エキソヌクレアーゼXrn1pを欠損させたところtRNA前駆体が蓄積した.これらの結果から,5’リーダー配列をもつtRNA前駆体は通常は5’エキソヌクレアーゼの標的になっており,tRNA前駆体における5’キャップ構造の形成は5’エキソヌクレアーゼによる分解からtRNA前駆体を保護する役割のあることが明らかにされた(図1).
この研究により,tRNA前駆体が5’キャップ構造をもつことがはじめて示された.また,5’キャップ構造はtRNA前駆体を5’エキソヌクレアーゼによる分解から保護する役割のあることが明らかにされた.tRNAの成熟の過程においてtRNA前駆体は細胞内をダイナミックに移動しながらプロセシングされることから,5’キャップ構造によるtRNA前駆体の保護はtRNAの成熟において重要な役割を担うことが推察された.とくに,alternative経路においては5’リーダー配列を保持したtRNA前駆体が細胞内を長距離かつ長時間にわたり移動するため5’キャップ構造が必要であると考えられた(図1).
tRNA前駆体において5’キャップ構造の役割はほかにもあるだろう.5’キャップ構造はtRNA前駆体とプロセシング酵素や輸送タンパク質との相互作用に影響をおよぼす可能性がある.また,5’キャップ構造がキャップ結合タンパク質との相互作用を介してtRNAの成熟を制御する可能性についても,今後,検証する予定である.また,5’キャップ構造をもつtRNA前駆体がキャップ結合タンパク質と相互作用すれば,5’キャップ構造をもつmRNAやほかのRNAポリメラーゼIIの転写産物と競合することになり,遺伝子発現になんらかの影響をおよぼす可能性が考えられる.今後のさらなる解析により,tRNA前駆体における5’キャップ構造の形成が関与する新しい遺伝子発現の制御機構を見い出したいと考えている.
略歴:2010年 東京大学大学院工学系研究科博士課程 修了,同年 同 学術支援専門職員,2013年 同 特任研究員を経て,同年より同 助教.
研究テーマ:tRNAの成熟化の機構.
抱負:急行に善歩なし.
鈴木 勉(Tsutomu Suzuki)
東京大学大学院工学系研究科 教授.
研究室URL:http://rna.chem.t.u-tokyo.ac.jp/
© 2016 大平高之・鈴木 勉 Licensed under CC 表示 2.1 日本
(東京大学大学院工学系研究科 化学生命工学専攻)
email:大平高之,鈴木 勉
DOI: 10.7875/first.author.2016.080
Precursors of tRNAs are stabilized by methylguanosine cap structures.
Takayuki Ohira, Tsutomu Suzuki
Nature Chemical Biology, 12, 648-655 (2016)
この論文に出現する遺伝子・タンパク質のUniprot ID
RNAポリメラーゼII, RNAポリメラーゼIII, 5'エキソヌクレアーゼ, RNase P(P28005), Trz1p(P36159), 3'エキソヌクレアーゼ, CCA付加酵素, Exportin-T(P33418), Los1p(P33418), RanGTPase(P32835), Gsp1p(P32835), Pop4p(P38336), Ceg1p, Tgs1p(Q12052), Swm2p(P40342), Cet1p(O13297), Abd1p(P32783), MET22(P32179), Rat1p(Q02792), Xrn1p(P22147), 5'エキソヌクレアーゼXrn1p(P22147), Ceg1, Cet1(O13297), Abd1(P32783), Tgs1(Q12052), Swm2(P40342), Rat1(Q02792), Xrn1(P22147)
要 約
遺伝暗号の解読をつかさどるtRNAは転写ののち多様な修飾をうけることにより成熟し本来の機能を発揮する.しかしながら,tRNA前駆体がプロセシングされる過程においてどのタイミングで修飾されるのか,また,その順序については不明な点が多く残されている.このような背景から,出芽酵母からさまざまなtRNA前駆体を単離しRNA高感度質量分析法を駆使してtRNAの修飾について詳細に解析したところ,5’リーダー配列をもつtRNA前駆体に5’キャップ構造が形成されていることが見い出された.一般に,5’キャップ構造はRNAポリメラーゼIIの転写と共役して形成されることが知られているが,tRNAはRNAポリメラーゼIIIの転写産物であり,この発見はこれまでの常識をくつがえす知見となった.遺伝学的な解析により,この5’キャップ構造はtRNA前駆体を5’エキソヌクレアーゼによる分解から保護する役割のあることが明らかにされた.
はじめに
tRNAはタンパク質の合成の過程においてmRNAのもつ遺伝情報をアミノ酸の配列へと変換するアダプター分子である.活発に増殖する細胞においてはタンパク質の高い合成能を維持するため大量のtRNAを合成する必要がある.真核生物における細胞質でのtRNAの合成に関する研究は出芽酵母を用いて詳細に行われている1).出芽酵母は43種類のtRNAをもち,これらは274個のtRNA遺伝子にコードされている.このうち,59の遺伝子がイントロンを含む2).tRNA遺伝子は核においてRNAポリメラーゼIIIにより転写され1次転写産物が生じる.そののち,RNase Pにより5’リーダー配列が切除され,Trz1pあるいは3’エキソヌクレアーゼにより3’トレーラー配列が取り除かれる.つづいて,CCA付加酵素により3’末端にCCA配列が付加される.5’末端および3’末端がプロセシングされたtRNA前駆体はExportin-T(Los1p)およびRanGTPase(Gsp1p)の作用により細胞質へと輸送される.tRNA前駆体がイントロンをもつ場合には,細胞質へと輸送されたのちミトコンドリア外膜に局在するtRNAスプライシング装置によりイントロンが除去される3).このようなtRNAの成熟化の経路を古典的な(classical)経路とよぶ.ところが,先行研究において,5’末端および3’末端のプロセシングよりさきにイントロンが取り除かれたtRNA前駆体の存在が知られていた4)(図1).tRNAスプライシング装置は細胞質にあることから,プロセシングされるまえのtRNA前駆体が細胞質へと輸送されイントロンが除去されることにより,この特徴的なtRNA前駆体が生じる.このtRNA前駆体は細胞質から核へと逆行的に輸送されることが示されていることから5),このtRNA前駆体は核へと移行したのち,核において5’末端および3’末端がプロセシングされ,ふたたび細胞質へと輸送されるという,もうひとつのtRNAの成熟化の経路であるalternative経路の存在が示唆される.
出芽酵母のtRNAには25種類の修飾が含まれており,tRNAの全残基のおよそ15%が修飾されている.tRNAの修飾はtRNAの安定性やコドンの認識などtRNAの構造や機能において重要な役割をはたしている.tRNAの修飾はtRNA前駆体がプロセシングされる過程において,核や細胞質に局在するtRNA修飾酵素により形成される.以前に,筆者らは,フェニルアラニンtRNAの37位にあるワイブトシン修飾が形成される過程において,tRNA前駆体は細胞質においてスプライシングされたのち核へと逆行的に輸送され,その第1段階の修飾である1-メチルグアノシン修飾をうけたのち,ふたたび細胞質へと輸送されることが必要であることを報告した6).このように,tRNAの成熟の過程において,tRNA前駆体が細胞内をダイナミックに移動しながらプロセシングや修飾をうけていることがしだいに明らかにされつつある.筆者らは,このような背景から,tRNAの成熟の過程をより詳細に理解することを目的として,出芽酵母からさまざまなtRNA前駆体を単離し修飾の状態や末端の構造について解析してきた.
1.5’キャップ構造をもつtRNA前駆体の発見
tRNA前駆体の修飾の状態や末端の構造を調べるため,5’リーダー配列を切除するRNase Pの発現を制御できる出芽酵母の株を用いた.このtetO7-POP4株はドキシサイクリンの添加によりRNase Pの構成成分であるPop4pの発現を転写の段階において抑制することができる.この株においてRNase Pを抑制して5’リーダー配列をもつtRNA前駆体を蓄積させ,筆者らの研究室において開発された往復循環クロマトグラフィー7) およびゲルの切り出しにより単離し,RNA高感度質量分析法8) によりその構造を解析した.その結果,5’リーダー配列の5’末端にm7Gキャップ構造,ジメチルGキャップ構造,トリメチルGキャップ構造というメチルグアノシンキャップ構造をもつtRNA前駆体が見い出された.この結果を検証するため,tetO7-POP4株においてRNase Pを抑制し,抗キャップ抗体を用いて全RNA画分から5’キャップ構造をもつRNAを免疫沈降により濃縮したのち,数種類のtRNAをノーザンブロット法により解析した.その結果,さまざまなtRNA前駆体が免疫沈降され,5’リーダー配列をもつtRNA前駆体は5’キャップ構造をもつことが明らかにされ,この現象を“tRNA前駆体のキャップ化”(pre-tRNA capping)と命名した.
出芽酵母の野生株からtRNA前駆体を単離したところ,やはり5’キャップ構造が検出された.野生株において5’キャップ構造をもつ率は5~25%であり,RNase Pを抑制したtetO7-POP4株における率と比較すると低かったが,この解析は活発に増殖する細胞におけるtRNA前駆体のスナップショットをみており,実際には,さらに多くのtRNA前駆体が5’キャップ構造をもつ可能性があると考えられた.
さまざまなtRNA前駆体の末端の構造の解析により,5’キャップ構造をもつ率はtRNA前駆体の種ごとに大きく異なることがわかった.5’リーダー配列が3’トレーラー配列と対合しやすいと5’キャップ構造をもつ率は低くなり,逆に,5’末端が1本鎖構造をとりやすいtRNA前駆体ほど5’キャップ構造をもつ率が高くなることが示唆された.5’キャップ構造の形成はtRNA前駆体の末端の構造に大きく影響されることが判明した.
ヒトのHeLa細胞を用いて同様に解析したところ,5’キャップ構造をもつtRNA前駆体はヒトの細胞においても観察された.この結果から,tRNA前駆体における5’キャップ構造の形成は真核生物の種をこえて広く保存されている可能性が示唆された.
2.5’キャップ構造をもつtRNA前駆体はRNAポリメラーゼIIIの転写産物である
通常,5’キャップ構造はmRNAやsnRNAなどRNAポリメラーゼIIの転写産物において形成されることが知られている.それは,転写の開始初期の段階においてRNAポリメラーゼIIにキャップ形成酵素が結合することにより,新生したRNA鎖の5’末端に5’キャップ構造が形成されるからである.tRNAは通常はRNAポリメラーゼIIIにより転写されるが,一部がRNAポリメラーゼIIにより転写されることによりtRNA前駆体において5’キャップ構造が形成される可能性が考えられた.そこで,RNAポリメラーゼIIIの温度感受性株を非許容温度において培養してRNAポリメラーゼIIIによる転写を阻害した際に,5’キャップ構造をもつtRNA前駆体の量に変化がみられるかどうかを調べた.その結果,抗キャップ抗体により免疫沈降させた画分においてtRNA前駆体のノーザンブロッティング法におけるシグナルが顕著に減少したことから,5’キャップ構造をもつtRNA前駆体はRNAポリメラーゼIIIの転写産物であることが判明した.
3.tRNA前駆体において5’キャップ構造は既知のキャップ形成酵素により形成される
tRNA前駆体における5’キャップ構造の形成に必要な酵素を同定した.出芽酵母における5’キャップ構造の付加酵素であるCeg1pは必須遺伝子にコードされているため,tetO7-POP4株を背景としてCeg1pの温度感受性変異株および過剰発現株を構築した.それぞれの株を非許容温度にて培養することによりRNase Pを抑制して5’リーダー配列をもつtRNA前駆体を蓄積させたのち,全RNAを抽出しそこからtRNAIleUAUを単離した.RNA高感度質量分析法により解析したところ,Ceg1pの温度感受性変異株では5’キャップ構造の形成が阻害され,Ceg1pの過剰発現株では5’キャップ構造をもつ率が上昇した.したがって,Ceg1pがtRNA前駆体における5’キャップ構造の形成に必須であることが判明した(図1).さらに,5’キャップ構造のメチル化酵素であるTgs1pがジメチルGキャップ構造およびトリメチルGキャップ構造の形成にかかわること,Tgs1pのパートナータンパク質であるSwm2pがTgs1pのはたらきをサポートすることも示された(図1).5’末端に二リン酸を形成する脱リン酸化酵素Cet1p,および,m7Gキャップ構造の形成に必要なメチル化酵素Abd1pの関与については未解析であるが,これらもtRNA前駆体における5’キャップ構造の形成に必要であると推察される.
4.5’キャップ構造はtRNA前駆体を5’エキソヌクレアーゼによる分解から保護する
一般に,5’キャップ構造にはRNAを5’エキソヌクレアーゼから保護する役割が知られている.そこで,tRNA前駆体も同様に5’キャップ構造により分解から保護されているかどうかを検証するため,tetO7-POP4株を背景としたCeg1pの温度感受性変異株および過剰発現株を用い,これらの株を非許容温度にて培養することによりRNase Pを抑制し,蓄積するtRNA前駆体の量をノーザンブロット法により調べた.その結果,Ceg1pの過剰発現株においては蓄積するtRNA前駆体の量が増加したのに対し,Ceg1pの温度感受性変異株においては顕著に減少した.そこで,tRNA前駆体が5’エキソヌクレアーゼにより分解されているかどうかを調べるため,tetO7-POP4株を背景としてMET22遺伝子の欠損株を構築した.MET22遺伝子を欠損させると5’エキソヌクレアーゼ(Rat1pおよびXrn1p)の活性を強力に阻害する5’,3’-ビスホスホアデノシンが細胞に蓄積する.この株においてRNase Pを抑制したところ,一部の5’リーダー配列をもつtRNA前駆体が顕著に蓄積した.また,細胞質に局在する5’エキソヌクレアーゼXrn1pを欠損させたところtRNA前駆体が蓄積した.これらの結果から,5’リーダー配列をもつtRNA前駆体は通常は5’エキソヌクレアーゼの標的になっており,tRNA前駆体における5’キャップ構造の形成は5’エキソヌクレアーゼによる分解からtRNA前駆体を保護する役割のあることが明らかにされた(図1).
おわりに
この研究により,tRNA前駆体が5’キャップ構造をもつことがはじめて示された.また,5’キャップ構造はtRNA前駆体を5’エキソヌクレアーゼによる分解から保護する役割のあることが明らかにされた.tRNAの成熟の過程においてtRNA前駆体は細胞内をダイナミックに移動しながらプロセシングされることから,5’キャップ構造によるtRNA前駆体の保護はtRNAの成熟において重要な役割を担うことが推察された.とくに,alternative経路においては5’リーダー配列を保持したtRNA前駆体が細胞内を長距離かつ長時間にわたり移動するため5’キャップ構造が必要であると考えられた(図1).
tRNA前駆体において5’キャップ構造の役割はほかにもあるだろう.5’キャップ構造はtRNA前駆体とプロセシング酵素や輸送タンパク質との相互作用に影響をおよぼす可能性がある.また,5’キャップ構造がキャップ結合タンパク質との相互作用を介してtRNAの成熟を制御する可能性についても,今後,検証する予定である.また,5’キャップ構造をもつtRNA前駆体がキャップ結合タンパク質と相互作用すれば,5’キャップ構造をもつmRNAやほかのRNAポリメラーゼIIの転写産物と競合することになり,遺伝子発現になんらかの影響をおよぼす可能性が考えられる.今後のさらなる解析により,tRNA前駆体における5’キャップ構造の形成が関与する新しい遺伝子発現の制御機構を見い出したいと考えている.
文 献
- Hopper, A. K.: Transfer RNA post-transcriptional processing, turnover, and subcellular dynamics in the yeast Saccharomyces cerevisiae. Genetics, 194, 43-67 (2013)[PubMed]
- Chan, P. P. & Lowe, T. M.: GtRNAdb: a database of transfer RNA genes detected in genomic sequence. Nucleic Acids Res., 37, D93-D97 (2009)[PubMed]
- Yoshihisa, T., Yunoki-Esaki, K., Ohshima, C. et al.: Possibility of cytoplasmic pre-tRNA splicing: the yeast tRNA splicing endonuclease mainly localizes on the mitochondria. Mol. Biol. Cell, 14, 3266-3279 (2003)[PubMed]
- O’Connor, J. P. & Peebles, C. L.: In vivo pre-tRNA processing in Saccharomyces cerevisiae. Mol. Cell. Biol., 11, 425-439 (1991)[PubMed]
- Kramer, E. B. & Hopper, A. K.: Retrograde transfer RNA nuclear import provides a new level of tRNA quality control in Saccharomyces cerevisiae. Proc. Natl. Acad. Sci. USA, 110, 21042-21047 (2013)[PubMed]
- Ohira, T. & Suzuki, T.: Retrograde nuclear import of tRNA precursors is required for modified base biogenesis in yeast. Proc. Natl. Acad. Sci. USA, 108, 10502-10507 (2011)[PubMed]
- Miyauchi, K., Ohara, T. & Suzuki, T.: Automated parallel isolation of multiple species of non-coding RNAs by the reciprocal circulating chromatography method. Nucleic Acids Res., 35, e24 (2007)[PubMed]
- Suzuki, T., Ikeuchi, Y., Noma, A. et al.: Mass spectrometric identification and characterization of RNA-modifying enzymes. Methods Enzymol., 425, 211-229 (2007)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2010年 東京大学大学院工学系研究科博士課程 修了,同年 同 学術支援専門職員,2013年 同 特任研究員を経て,同年より同 助教.
研究テーマ:tRNAの成熟化の機構.
抱負:急行に善歩なし.
鈴木 勉(Tsutomu Suzuki)
東京大学大学院工学系研究科 教授.
研究室URL:http://rna.chem.t.u-tokyo.ac.jp/
© 2016 大平高之・鈴木 勉 Licensed under CC 表示 2.1 日本