生細胞におけるリアルタイムでの1分子のmRNAからの翻訳のダイナミクスの可視化および定量
森崎達也・Timothy J. Stasevich
(米国Colorado州立大学Department of Biochemistry and Molecular Biology)
email:森崎達也
DOI: 10.7875/first.author.2016.049
Real-time quantification of single RNA translation dynamics in living cells.
Tatsuya Morisaki, Kenneth Lyon, Keith F. DeLuca, Jennifer G. DeLuca, Brian P. English, Zhengjian Zhang, Luke D. Lavis, Jonathan B. Grimm, Sarada Viswanathan, Loren L. Looger, Timothee Lionnet, Timothy J. Stasevich
Science, 352,1425-1429 (2016)
RNAからタンパク質への翻訳は遺伝子の発現においてもっとも重要な過程のひとつであるが,翻訳の過程を生細胞において1分子レベルの精度で可視化することに成功した例はない.筆者らは,複数のエピトープタグおよび抗体をもとにした蛍光プローブを用いることにより,生細胞において1分子のmRNAからの翻訳を可視化しそのダイナミクスを定量する技術を開発し,NCT法と命名した.このNCT法を用いることにより,翻訳は毎秒約10アミノ酸残基を伸長する速度で進むこと,また,30秒ごとに確率的に開始することが明らかにされた.また,mRNAは200~900塩基ごとにひとつのリボソームを含むポリソームを形成することがわかった.さらに,多色の蛍光プローブを開発し2つの異なるタンパク質を標識することにより,ほとんどのポリソームは独立して機能するが,一部のポリソームは2つの異なるmRNAを同時に翻訳する高次複合体を形成しうることが見い出された.NCT法はすぐれた感度と多様性をもち,今後,生細胞における1分子のmRNAからの翻訳のダイナミクスを定量する強力なツールとして期待される.
遺伝子の発現はDNAからRNAへの転写,および,RNAからタンパク質への翻訳という2つの過程からなり,この2つの過程が遺伝子発現制御の根幹をなす.現在,転写の過程は,生細胞においてリアルタイムでの1遺伝子の精度の可視化および定量が実現されているが1),翻訳の過程に関してはいまだ実例がない.原理的には,GFPに代表される蛍光タンパク質を用いることにより翻訳の可視化は可能であるが,現実的には,蛍光タンパク質は蛍光を発するまでにフォールディングおよび蛍光団の形成に時間を必要とすることから,蛍光タンパク質を用いて1分子のmRNAからの翻訳をリアルタイムで可視化することはきわめて困難である2).近年,翻訳反応をつかさどるリボソームがmRNAを通過することでmRNAから解離するRNAバイオセンサーを用いることにより生細胞における翻訳の過程を可視化した研究が報告されたが,この系では1回の翻訳の過程でシグナルが失われるため,継続した翻訳の過程のダイナミクスを可視化し定量することは不可能である3).筆者らは,複数のエピトープタグおよび抗体をもとにした蛍光プローブ,および,きわめて明るく退色に強い蛍光色素を組み合わせることにより,生細胞における1分子のmRNAからの翻訳ダイナミクスの可視化および定量に取り組んだ.
1分子のmRNAからの翻訳をリアルタイムで可視化するのに抗原抗体反応を利用した.N末端に複数のエピトープタグを融合させた対象となるタンパク質の遺伝子をコードするDNA,および,蛍光色素により標識したそのエピトープタグを認識する抗体のFab領域を細胞に導入することにより,対象となるタンパク質がリボソームにおいて翻訳されるやいなや,そのフォールディングを待つことなく蛍光標識されたFab領域がそのエピトープタグを認識し集積するため,翻訳されてすぐの新生ペプチド鎖を可視化することができるのではないかと考えたのである.
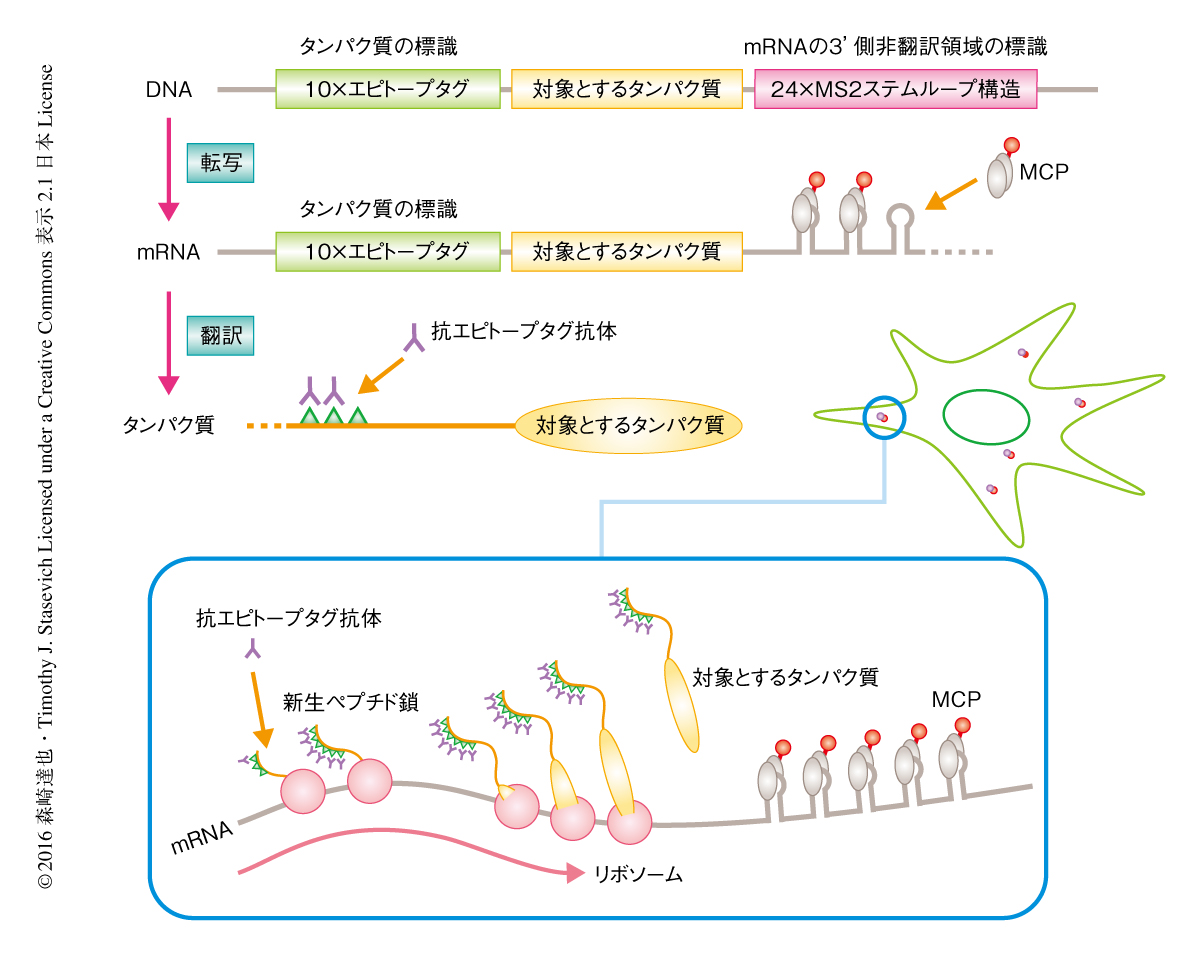
このアイデアの実現可能性を確認するため,エピトープタグとして10×FLAGタグ 4) を,対象となるタンパク質として1544アミノ酸残基と比較的大きい核タンパク質KDM5Bを選択し,これに,mRNAを可視化するために3’側非翻訳領域に24×MS2ステムループ構造5) を組み込んだDNAを作製し,ヒト骨肉腫細胞であるU2OS細胞に導入した(図1).つづいて,蛍光色素Cy3により標識した抗FLAGタグ抗体のFab領域,および,蛍光色素JF646 6) により標識したMS2ステムループ構造に結合するタンパク質MCPを細胞に導入した(図1).24時間後,MCPは細胞質に存在するmRNAを,Fab領域は核に存在するKDM5Bを標識し,複数のエピトープタグおよびFab領域の存在はKDM5Bの翻訳および局在に干渉しないことが確認された.Fab領域がどれだけ早くタンパク質を標識できるか確認するため,DNAの導入から6時間後に観察したところ,まだ多くのKDM5Bが翻訳されていないため,Fab領域は核をうっすらと標識するのみで,多くのFab領域はMCPにより標識された細胞質のmRNAと共局在し,mRNAとともに細胞質において移動していた.
このmRNAとFab領域の共局在したスポットが,翻訳中のmRNAとその翻訳産物のスポットであるかどうかを2種類の翻訳阻害剤を用いて確かめた.翻訳中のタンパク質をリボソームから放出することの知られる翻訳阻害剤ピューロマイシンを添加したところ,mRNAに共局在していたFab領域のシグナルが失われた.また,翻訳伸長速度を低下させmRNAあたりのリボソームの数を増加させることの知られる翻訳阻害剤シクロヘキシミドを添加したところ,共局在したスポットにおけるFab領域のシグナルが増加した.これらの結果より,このスポットはまさに翻訳中のmRNAとその翻訳産物であると考えられた.
より小さなタンパク質の翻訳の観察が可能かどうか調べるため,KDM5Bをβアクチン(374アミノ酸残基),あるいは,ヒストンH2B(125アミノ酸残基)に置き換えて同様に実験した.その結果,24時間後にβアクチンは細胞質にフィラメント様の局在,ヒストンH2Bは核に局在を示していたが,6時間後の時点においてはβアクチンおよびヒストンH2BともKDM5Bと同様に,細胞質においてmRNAとその翻訳産物が共局在したスポットを示した.以上より,この系により,さまざまな大きさのタンパク質の翻訳が1分子レベルで可視化できることが示唆された(図1).
タイムラプスイメージングにより翻訳中の1分子のmRNAとその翻訳産物の共局在したスポットを追跡する,新生ペプチド鎖追跡(nascent chain tracking:NCT)法に取り組んだ.翻訳中のタンパク質のイメージングに特化するため,翻訳済みのタンパク質を可視化せず,mRNAと共局在するタンパク質のみを可視化できるようレーザーの強度を調整した.この条件では,ポリソーム(1分子のmRNAとそれを翻訳する複数のリボソーム,さらに,これらのリボソームにより翻訳され新規に合成中のタンパク質)のみが可視化される.このNCT法を用いることにより,KDM5B,βアクチン,ヒストンH2BをコードするmRNAの翻訳の頻度およびその程度,ポリソームの移動性,ポリソームの大きさの測定を試みた.
細胞におけるすべてのmRNAおよびポリソームの数を定量し,その比率を計算することにより翻訳の頻度を調べた.約90%のKDM5BのmRNAが翻訳中であったのに対し,βアクチンは約20%,ヒストンH2Bは約5%のmRNAが翻訳中であった(ひとつのリボソームにより翻訳されるmRNAはこの割合には含まれない).さらに,ポリソームにおけるFab領域のシグナルは,KDM5Bはβアクチンに比べ約1.5倍強く,βアクチンはヒストンH2Bに比べ約1.5倍強かった.Fab領域のシグナルの蛍光強度を較正することにより,KDM5Bのポリソームは約5個,βアクチンのポリソームは約3個,ヒストンH2Bのポリソームは約2個のリボソームをもつことが見い出された.それぞれのmRNAの長さをリボソームの数で割ることにより,mRNAは200~900塩基ごとにひとつのリボソームを含むポリソームを形成することがわかった.
追跡したポリソームの移動性を平均2乗変異(mean-square displacement:MSD)法により定量した.核タンパク質であるKDM5BおよびヒストンH2Bのポリソームはその平均2乗変異が時間に対し線形に増加したことから,細胞質において通常拡散をしていることがわかった.また,KDM5Bのポリソームに比べて小さいヒストンH2Bのポリソームの多くは,KDM5Bより顕著に速く拡散した.βアクチンのポリソームは非線形拡散をしていたが,βアクチンはKDM5BやヒストンH2Bと異なり細胞質において機能するため,翻訳されたそばからほかのタンパク質と相互作用する可能性があり,その結果,非線形拡散を示すことが考えられた.これらの傾向とは別に,個々のポリソームの移動性はきわめて多様であり,非常に速く拡散するKDM5Bのポリソームや,ほぼ動かないヒストンH2Bのポリソームもみられた.これまで,翻訳中のmRNAの移動性は低下すると考えられていたが,mRNAの移動性のみからその翻訳の状態を知ることはきわめて困難であることがわかった.
これまで,電子顕微鏡により,ヘアピン構造や球状構造をとるポリソームが観察されている7).生細胞におけるポリソームの大きさおよび形に関する情報を得るため,KDM5B,βアクチン,ヒストンH2Bのポリソームにおいて,翻訳中のタンパク質の平均位置(Fab領域のシグナル)とmRNAの3’側非翻訳領域(MCPのシグナル)との距離を調べた.もし,ポリソームがヘアピン構造をとるなら,mRNAの長さに比例してこの距離は増加しているはずである.しかし,実際はKDM5Bのポリソームにおける距離がもっとも短く,このことより,観察されたポリソームはより球状に近い構造をしていることが示唆された.このことは,最近の原子間力顕微鏡による観察とも一致した8).
ポリソームの基本的な性質に関する情報が得られたところで,ポリソームにおける翻訳のダイナミクスを明らかにすることに取り組んだ.DNAからRNAへの転写伸長速度を定量する非侵襲的な方法として,蛍光相関分光法(fluorescence correlation spectroscopy:FCS)を用いてシグナルのゆらぎの相関から算出する方法が報告されている9).翻訳伸長速度を定量するため,ポリソームにおけるFab領域のシグナルのゆらぎの相関を蛍光相関分光法により調べた.これにさきだち,抗FLAGタグ抗体のFab領域はFLAGタグと数秒で結合すること,また,結合すると5分以上も解離しないことを確認した.以上の結果より,翻訳伸長反応が数秒から5分以内であれば,抗FLAGタグ抗体のFab領域のFLAGタグへの結合のダイナミクスは翻訳のダイナミクスの定量に影響をあたえないと考えられた.
KDM5BのポリソームにおけるFabシグナルの蛍光相関より,KDM5Bの翻訳伸長反応には約150秒を要することが見い出された.翻訳阻害剤であるシクロヘキシミドを添加したところこの蛍光相関は失われた.光退色後蛍光回復(fluorescence recovery after photobleaching:FRAP)法を用いてKDM5Bのポリソームにおける翻訳伸長速度を定量することを試みた.レーザーの強度を調整することにより,MCPのシグナルを維持しFab領域のシグナルのみを光退色させたのち,mRNAを追跡しFab領域のシグナルの蛍光強度が回復するまでの時間を測定したところ,蛍光強度の回復には125秒~180秒を要し,蛍光相関分光法による結果とほぼ同じタイムスケールであることが確認された.さらに,蛍光相関分光法により,βアクチンおよびヒストンH2Bは翻訳伸長反応にそれぞれ約32秒および約16秒を要することが明らかにされた.それぞれのタンパク質のアミノ酸残基数を翻訳伸長反応に要した時間で割ることにより翻訳伸長速度を算出したところ,3つのタンパク質の翻訳伸長速度に顕著な差はなく,1秒あたり約10アミノ酸残基を伸長することがわかった.全ゲノムを対象としたリボソームプロファイリングにおいて翻訳伸長速度は1秒あたり5.6アミノ酸残基と算出されており10),算出された翻訳伸長速度とは大きな差はなく,このことからも蛍光相関分光法を用いた翻訳のダイナミクスの定量の有用性が確認された.さらに,蛍光相関分光法により,翻訳の過程において翻訳のバーストおよび記憶は観察されず,翻訳の開始は確率的に起こると考えられ,また,毎秒10アミノ酸残基の翻訳伸長速度でKDM5BのmRNAに約5個のリボソームが含まれたことから,翻訳は30秒ごとに確率的に開始すると算出された.
翻訳の可視化に抗体を用いる利点のひとつとして,異なるエピトープタグを高い親和性で認識するさまざまな抗体が存在するため,多色のイメージングに応用できることがあげられる.この応用例として,KDM5Bのタグを10×FLAGタグから10×HAタグ4) に変更し,蛍光色素Cy3により標識した抗FLAGタグ抗体のFab領域および蛍光色素Alexa488により標識した抗HAタグ抗体のFab領域により,2つの異なるmRNAの翻訳を単一の生細胞において同時に観察した.ほとんどすべてのポリソームはFLAGタグを付加したKDM5BあるいはHAタグを付加したKDM5Bのどちらかを翻訳したが,約5%のポリソームは両方を翻訳する高次複合体を形成しうることが見い出された.この高次複合体の機能は謎であるが,2つの異なる遺伝子の発現を翻訳の段階において同時に制御したり,翻訳中のタンパク質がその場で多量体化するのを促進したりといった,より一般的な遺伝子発現制御の機能をもつのかもしれない.
複数のエピトープタグおよび抗体をもとにした蛍光プローブを用いたNCT法は,生細胞においてリアルタイムで1分子のmRNAからの翻訳を可視化し定量することを可能にした.Science誌にこの論文と同時に掲載されたSunタグ 11) を使った技術によっても,筆者らの得た翻訳伸長速度および翻訳開始速度ときわめて近い値が算出されており12),異なる研究室のあいだでも再現性の得られることが示された.Sunタグを使った技術とNCT法は類似しているが,抗体を用いたNCT法はSunタグを使った技術では不可能な多色のイメージングへの応用が即座に可能であり,また,エピトープタグが1/3ほどの短さであるため時空間分解能が約3倍であること,翻訳への影響が小さいこと,などの利点があげられる.今後,NCT法は翻訳の過程における遺伝子発現ネットワークの制御の解明,および,翻訳の過程の制御のミスに起因する疾病の解明に貢献すると考えられる.
略歴:2010年 京都大学大学院薬学研究科博士課程 修了,同年 米国NIH National Cancer Instituteポスドクを経て,2014年より米国Colorado州立大学Research Scientist.
研究テーマ:遺伝子発現のダイナミクス.
抱負:新規のイメージング技術の開発をつうじて遺伝子発現制御の理解を深め,関連した疾病の治療の基礎開発に貢献したい.
Timothy J. Stasevich
米国Colorado州立大学Assistant Professor.
研究室URL:http://sites.bmb.colostate.edu/stasevichlab/
© 2016 森崎達也・Timothy J. Stasevich Licensed under CC 表示 2.1 日本
(米国Colorado州立大学Department of Biochemistry and Molecular Biology)
email:森崎達也
DOI: 10.7875/first.author.2016.049
Real-time quantification of single RNA translation dynamics in living cells.
Tatsuya Morisaki, Kenneth Lyon, Keith F. DeLuca, Jennifer G. DeLuca, Brian P. English, Zhengjian Zhang, Luke D. Lavis, Jonathan B. Grimm, Sarada Viswanathan, Loren L. Looger, Timothee Lionnet, Timothy J. Stasevich
Science, 352,1425-1429 (2016)
この論文に出現する遺伝子・タンパク質のUniprot ID
要 約
RNAからタンパク質への翻訳は遺伝子の発現においてもっとも重要な過程のひとつであるが,翻訳の過程を生細胞において1分子レベルの精度で可視化することに成功した例はない.筆者らは,複数のエピトープタグおよび抗体をもとにした蛍光プローブを用いることにより,生細胞において1分子のmRNAからの翻訳を可視化しそのダイナミクスを定量する技術を開発し,NCT法と命名した.このNCT法を用いることにより,翻訳は毎秒約10アミノ酸残基を伸長する速度で進むこと,また,30秒ごとに確率的に開始することが明らかにされた.また,mRNAは200~900塩基ごとにひとつのリボソームを含むポリソームを形成することがわかった.さらに,多色の蛍光プローブを開発し2つの異なるタンパク質を標識することにより,ほとんどのポリソームは独立して機能するが,一部のポリソームは2つの異なるmRNAを同時に翻訳する高次複合体を形成しうることが見い出された.NCT法はすぐれた感度と多様性をもち,今後,生細胞における1分子のmRNAからの翻訳のダイナミクスを定量する強力なツールとして期待される.
はじめに
遺伝子の発現はDNAからRNAへの転写,および,RNAからタンパク質への翻訳という2つの過程からなり,この2つの過程が遺伝子発現制御の根幹をなす.現在,転写の過程は,生細胞においてリアルタイムでの1遺伝子の精度の可視化および定量が実現されているが1),翻訳の過程に関してはいまだ実例がない.原理的には,GFPに代表される蛍光タンパク質を用いることにより翻訳の可視化は可能であるが,現実的には,蛍光タンパク質は蛍光を発するまでにフォールディングおよび蛍光団の形成に時間を必要とすることから,蛍光タンパク質を用いて1分子のmRNAからの翻訳をリアルタイムで可視化することはきわめて困難である2).近年,翻訳反応をつかさどるリボソームがmRNAを通過することでmRNAから解離するRNAバイオセンサーを用いることにより生細胞における翻訳の過程を可視化した研究が報告されたが,この系では1回の翻訳の過程でシグナルが失われるため,継続した翻訳の過程のダイナミクスを可視化し定量することは不可能である3).筆者らは,複数のエピトープタグおよび抗体をもとにした蛍光プローブ,および,きわめて明るく退色に強い蛍光色素を組み合わせることにより,生細胞における1分子のmRNAからの翻訳ダイナミクスの可視化および定量に取り組んだ.
1.生細胞において1分子のmRNAからの翻訳を可視化する手法の開発
1分子のmRNAからの翻訳をリアルタイムで可視化するのに抗原抗体反応を利用した.N末端に複数のエピトープタグを融合させた対象となるタンパク質の遺伝子をコードするDNA,および,蛍光色素により標識したそのエピトープタグを認識する抗体のFab領域を細胞に導入することにより,対象となるタンパク質がリボソームにおいて翻訳されるやいなや,そのフォールディングを待つことなく蛍光標識されたFab領域がそのエピトープタグを認識し集積するため,翻訳されてすぐの新生ペプチド鎖を可視化することができるのではないかと考えたのである.
このアイデアの実現可能性を確認するため,エピトープタグとして10×FLAGタグ 4) を,対象となるタンパク質として1544アミノ酸残基と比較的大きい核タンパク質KDM5Bを選択し,これに,mRNAを可視化するために3’側非翻訳領域に24×MS2ステムループ構造5) を組み込んだDNAを作製し,ヒト骨肉腫細胞であるU2OS細胞に導入した(図1).つづいて,蛍光色素Cy3により標識した抗FLAGタグ抗体のFab領域,および,蛍光色素JF646 6) により標識したMS2ステムループ構造に結合するタンパク質MCPを細胞に導入した(図1).24時間後,MCPは細胞質に存在するmRNAを,Fab領域は核に存在するKDM5Bを標識し,複数のエピトープタグおよびFab領域の存在はKDM5Bの翻訳および局在に干渉しないことが確認された.Fab領域がどれだけ早くタンパク質を標識できるか確認するため,DNAの導入から6時間後に観察したところ,まだ多くのKDM5Bが翻訳されていないため,Fab領域は核をうっすらと標識するのみで,多くのFab領域はMCPにより標識された細胞質のmRNAと共局在し,mRNAとともに細胞質において移動していた.
このmRNAとFab領域の共局在したスポットが,翻訳中のmRNAとその翻訳産物のスポットであるかどうかを2種類の翻訳阻害剤を用いて確かめた.翻訳中のタンパク質をリボソームから放出することの知られる翻訳阻害剤ピューロマイシンを添加したところ,mRNAに共局在していたFab領域のシグナルが失われた.また,翻訳伸長速度を低下させmRNAあたりのリボソームの数を増加させることの知られる翻訳阻害剤シクロヘキシミドを添加したところ,共局在したスポットにおけるFab領域のシグナルが増加した.これらの結果より,このスポットはまさに翻訳中のmRNAとその翻訳産物であると考えられた.
より小さなタンパク質の翻訳の観察が可能かどうか調べるため,KDM5Bをβアクチン(374アミノ酸残基),あるいは,ヒストンH2B(125アミノ酸残基)に置き換えて同様に実験した.その結果,24時間後にβアクチンは細胞質にフィラメント様の局在,ヒストンH2Bは核に局在を示していたが,6時間後の時点においてはβアクチンおよびヒストンH2BともKDM5Bと同様に,細胞質においてmRNAとその翻訳産物が共局在したスポットを示した.以上より,この系により,さまざまな大きさのタンパク質の翻訳が1分子レベルで可視化できることが示唆された(図1).
2.NCT法によるポリソームの移動性および大きさの定量
タイムラプスイメージングにより翻訳中の1分子のmRNAとその翻訳産物の共局在したスポットを追跡する,新生ペプチド鎖追跡(nascent chain tracking:NCT)法に取り組んだ.翻訳中のタンパク質のイメージングに特化するため,翻訳済みのタンパク質を可視化せず,mRNAと共局在するタンパク質のみを可視化できるようレーザーの強度を調整した.この条件では,ポリソーム(1分子のmRNAとそれを翻訳する複数のリボソーム,さらに,これらのリボソームにより翻訳され新規に合成中のタンパク質)のみが可視化される.このNCT法を用いることにより,KDM5B,βアクチン,ヒストンH2BをコードするmRNAの翻訳の頻度およびその程度,ポリソームの移動性,ポリソームの大きさの測定を試みた.
細胞におけるすべてのmRNAおよびポリソームの数を定量し,その比率を計算することにより翻訳の頻度を調べた.約90%のKDM5BのmRNAが翻訳中であったのに対し,βアクチンは約20%,ヒストンH2Bは約5%のmRNAが翻訳中であった(ひとつのリボソームにより翻訳されるmRNAはこの割合には含まれない).さらに,ポリソームにおけるFab領域のシグナルは,KDM5Bはβアクチンに比べ約1.5倍強く,βアクチンはヒストンH2Bに比べ約1.5倍強かった.Fab領域のシグナルの蛍光強度を較正することにより,KDM5Bのポリソームは約5個,βアクチンのポリソームは約3個,ヒストンH2Bのポリソームは約2個のリボソームをもつことが見い出された.それぞれのmRNAの長さをリボソームの数で割ることにより,mRNAは200~900塩基ごとにひとつのリボソームを含むポリソームを形成することがわかった.
追跡したポリソームの移動性を平均2乗変異(mean-square displacement:MSD)法により定量した.核タンパク質であるKDM5BおよびヒストンH2Bのポリソームはその平均2乗変異が時間に対し線形に増加したことから,細胞質において通常拡散をしていることがわかった.また,KDM5Bのポリソームに比べて小さいヒストンH2Bのポリソームの多くは,KDM5Bより顕著に速く拡散した.βアクチンのポリソームは非線形拡散をしていたが,βアクチンはKDM5BやヒストンH2Bと異なり細胞質において機能するため,翻訳されたそばからほかのタンパク質と相互作用する可能性があり,その結果,非線形拡散を示すことが考えられた.これらの傾向とは別に,個々のポリソームの移動性はきわめて多様であり,非常に速く拡散するKDM5Bのポリソームや,ほぼ動かないヒストンH2Bのポリソームもみられた.これまで,翻訳中のmRNAの移動性は低下すると考えられていたが,mRNAの移動性のみからその翻訳の状態を知ることはきわめて困難であることがわかった.
これまで,電子顕微鏡により,ヘアピン構造や球状構造をとるポリソームが観察されている7).生細胞におけるポリソームの大きさおよび形に関する情報を得るため,KDM5B,βアクチン,ヒストンH2Bのポリソームにおいて,翻訳中のタンパク質の平均位置(Fab領域のシグナル)とmRNAの3’側非翻訳領域(MCPのシグナル)との距離を調べた.もし,ポリソームがヘアピン構造をとるなら,mRNAの長さに比例してこの距離は増加しているはずである.しかし,実際はKDM5Bのポリソームにおける距離がもっとも短く,このことより,観察されたポリソームはより球状に近い構造をしていることが示唆された.このことは,最近の原子間力顕微鏡による観察とも一致した8).
3.蛍光相関分光法および光退色後蛍光回復法による翻訳のダイナミクスの定量
ポリソームの基本的な性質に関する情報が得られたところで,ポリソームにおける翻訳のダイナミクスを明らかにすることに取り組んだ.DNAからRNAへの転写伸長速度を定量する非侵襲的な方法として,蛍光相関分光法(fluorescence correlation spectroscopy:FCS)を用いてシグナルのゆらぎの相関から算出する方法が報告されている9).翻訳伸長速度を定量するため,ポリソームにおけるFab領域のシグナルのゆらぎの相関を蛍光相関分光法により調べた.これにさきだち,抗FLAGタグ抗体のFab領域はFLAGタグと数秒で結合すること,また,結合すると5分以上も解離しないことを確認した.以上の結果より,翻訳伸長反応が数秒から5分以内であれば,抗FLAGタグ抗体のFab領域のFLAGタグへの結合のダイナミクスは翻訳のダイナミクスの定量に影響をあたえないと考えられた.
KDM5BのポリソームにおけるFabシグナルの蛍光相関より,KDM5Bの翻訳伸長反応には約150秒を要することが見い出された.翻訳阻害剤であるシクロヘキシミドを添加したところこの蛍光相関は失われた.光退色後蛍光回復(fluorescence recovery after photobleaching:FRAP)法を用いてKDM5Bのポリソームにおける翻訳伸長速度を定量することを試みた.レーザーの強度を調整することにより,MCPのシグナルを維持しFab領域のシグナルのみを光退色させたのち,mRNAを追跡しFab領域のシグナルの蛍光強度が回復するまでの時間を測定したところ,蛍光強度の回復には125秒~180秒を要し,蛍光相関分光法による結果とほぼ同じタイムスケールであることが確認された.さらに,蛍光相関分光法により,βアクチンおよびヒストンH2Bは翻訳伸長反応にそれぞれ約32秒および約16秒を要することが明らかにされた.それぞれのタンパク質のアミノ酸残基数を翻訳伸長反応に要した時間で割ることにより翻訳伸長速度を算出したところ,3つのタンパク質の翻訳伸長速度に顕著な差はなく,1秒あたり約10アミノ酸残基を伸長することがわかった.全ゲノムを対象としたリボソームプロファイリングにおいて翻訳伸長速度は1秒あたり5.6アミノ酸残基と算出されており10),算出された翻訳伸長速度とは大きな差はなく,このことからも蛍光相関分光法を用いた翻訳のダイナミクスの定量の有用性が確認された.さらに,蛍光相関分光法により,翻訳の過程において翻訳のバーストおよび記憶は観察されず,翻訳の開始は確率的に起こると考えられ,また,毎秒10アミノ酸残基の翻訳伸長速度でKDM5BのmRNAに約5個のリボソームが含まれたことから,翻訳は30秒ごとに確率的に開始すると算出された.
4.多色のイメージングによる2つの異なるmRNAの翻訳の同時観察
翻訳の可視化に抗体を用いる利点のひとつとして,異なるエピトープタグを高い親和性で認識するさまざまな抗体が存在するため,多色のイメージングに応用できることがあげられる.この応用例として,KDM5Bのタグを10×FLAGタグから10×HAタグ4) に変更し,蛍光色素Cy3により標識した抗FLAGタグ抗体のFab領域および蛍光色素Alexa488により標識した抗HAタグ抗体のFab領域により,2つの異なるmRNAの翻訳を単一の生細胞において同時に観察した.ほとんどすべてのポリソームはFLAGタグを付加したKDM5BあるいはHAタグを付加したKDM5Bのどちらかを翻訳したが,約5%のポリソームは両方を翻訳する高次複合体を形成しうることが見い出された.この高次複合体の機能は謎であるが,2つの異なる遺伝子の発現を翻訳の段階において同時に制御したり,翻訳中のタンパク質がその場で多量体化するのを促進したりといった,より一般的な遺伝子発現制御の機能をもつのかもしれない.
おわりに
複数のエピトープタグおよび抗体をもとにした蛍光プローブを用いたNCT法は,生細胞においてリアルタイムで1分子のmRNAからの翻訳を可視化し定量することを可能にした.Science誌にこの論文と同時に掲載されたSunタグ 11) を使った技術によっても,筆者らの得た翻訳伸長速度および翻訳開始速度ときわめて近い値が算出されており12),異なる研究室のあいだでも再現性の得られることが示された.Sunタグを使った技術とNCT法は類似しているが,抗体を用いたNCT法はSunタグを使った技術では不可能な多色のイメージングへの応用が即座に可能であり,また,エピトープタグが1/3ほどの短さであるため時空間分解能が約3倍であること,翻訳への影響が小さいこと,などの利点があげられる.今後,NCT法は翻訳の過程における遺伝子発現ネットワークの制御の解明,および,翻訳の過程の制御のミスに起因する疾病の解明に貢献すると考えられる.
文 献
- Hager, G. L., McNally, J. G. & Misteli, T.: Transcription dynamics. Mol. Cell, 35, 741-753 (2009)[PubMed]
- Chao, J. A., Yoon, Y. J. & Singer, R. H.: Imaging translation in single cells using fluorescent microscopy. Cold Spring Harb. Perspect. Biol., 4, a012310 (2012)[PubMed]
- Halstead, J. M., Lionnet, T., Wilbertz, J. H. et al.: An RNA biosensor for imaging the first round of translation from single cells to living animals. Science, 347, 1367-1671 (2015)[PubMed]
- Viswanathan, S., Williams, M. E., Bloss, E. B. et al.: High-performance probes for light and electron microscopy. Nat. Methods, 12, 568-576 (2015)[PubMed]
- Bertrand, E., Chartrand, P., Schaefer, M. et al.: Localization of ASH1 mRNA particles in living yeast. Mol. Cell, 2, 437-445 (1998)[PubMed]
- Grimm, J. B., English, B. P., Chen, J. et al.: A general method to improve fluorophores for live-cell and single-molecule microscopy. Nat. Methods, 12, 244-250 (2015)[PubMed]
- Christensen, A. K. & Bourne, C. M.: Shape of large bound polysomes in cultured fibroblasts and thyroid epithelial cells. Anat. Rec., 255, 116-129 (1999)[PubMed]
- Viero, G., Lunelli, L., Passerini, A. et al.: Three distinct ribosome assemblies modulated by translation are the building blocks of polysomes. J. Cell Biol., 208, 581-596 (2015)[PubMed]
- Larson, D. R., Zenklusen, D., Wu, B. et al.: Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science, 332, 475-478 (2011)[PubMed]
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. et al.: Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science, 324, 218-223 (2009)[PubMed]
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S. et al.: A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 159, 635-646 (2014)[PubMed]
- Wu, B., Eliscovich, C., Yoon, Y. J. et al.: Translation dynamics of single mRNAs in live cells and neurons. Science, 352, 1430-1435 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2010年 京都大学大学院薬学研究科博士課程 修了,同年 米国NIH National Cancer Instituteポスドクを経て,2014年より米国Colorado州立大学Research Scientist.
研究テーマ:遺伝子発現のダイナミクス.
抱負:新規のイメージング技術の開発をつうじて遺伝子発現制御の理解を深め,関連した疾病の治療の基礎開発に貢献したい.
Timothy J. Stasevich
米国Colorado州立大学Assistant Professor.
研究室URL:http://sites.bmb.colostate.edu/stasevichlab/
© 2016 森崎達也・Timothy J. Stasevich Licensed under CC 表示 2.1 日本